昨天我们分享了:《 细胞亚群细分的时候仍然是要选择harmony等算法去除样品差异 》,有粉丝留言提到这个使用harmony等算法去除样品差异,不应该是最开始就弄吗。为什么要到细分亚群的时候才做呢?
我们仍然是以 这个单细胞转录组文献,《Single-cell transcriptomics reveals regulators underlying immune cell diversity and immune subtypes associated with prognosis in nasopharyngeal carcinoma》为例子,15个鼻咽癌样品,加上1个正常人样品。全部的样品的单细胞转录组数据整合后,如果不使用harmony等算法去除样品差异,默认的降维聚类分群,如下所示:
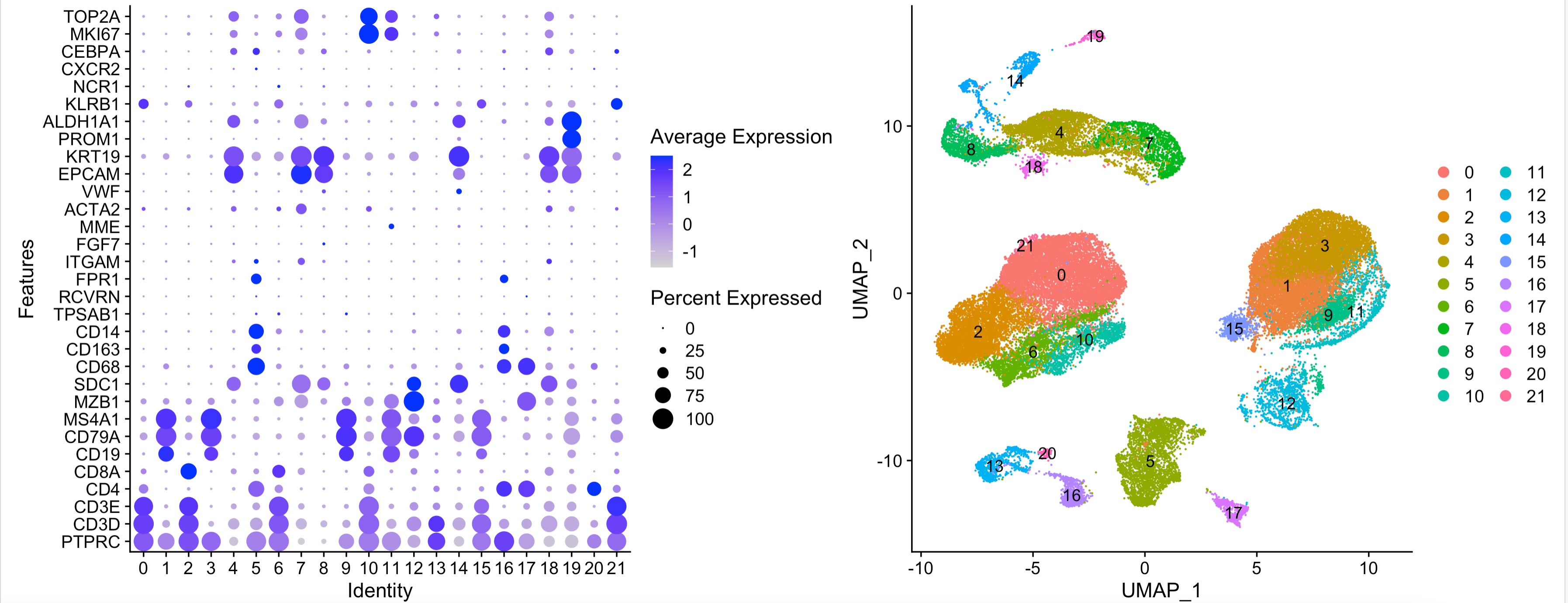
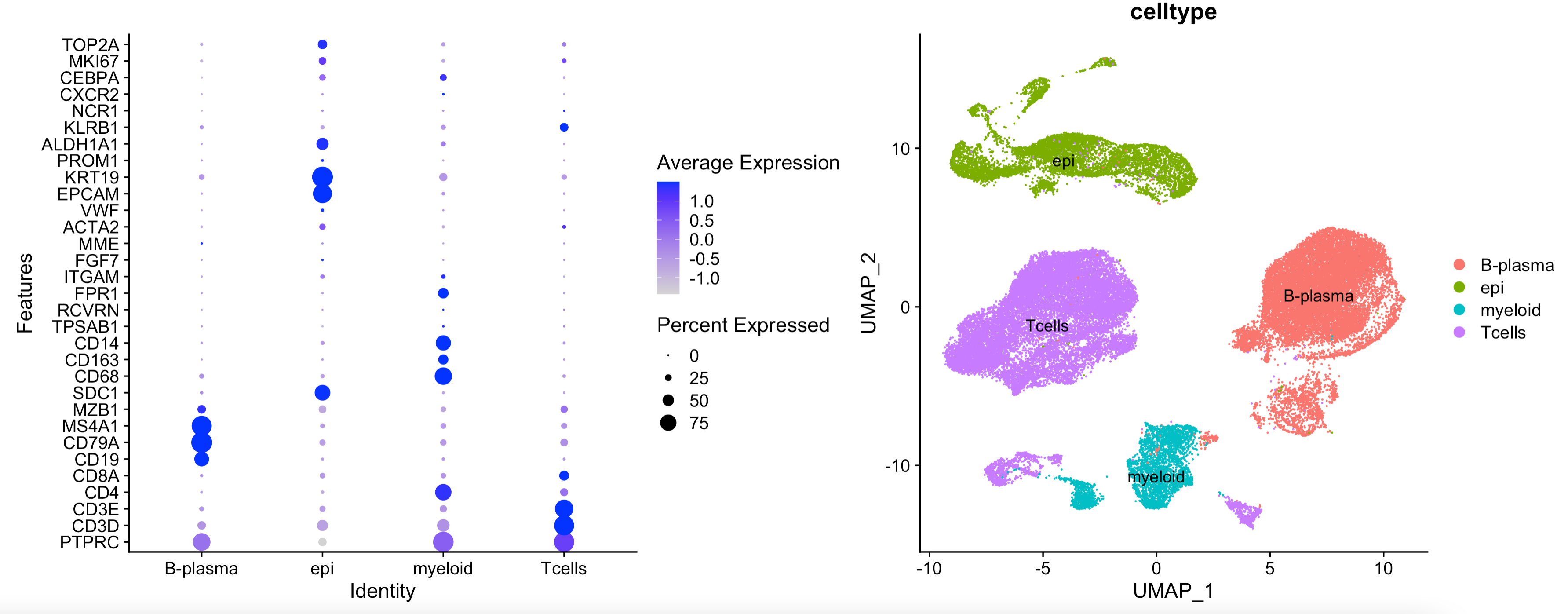
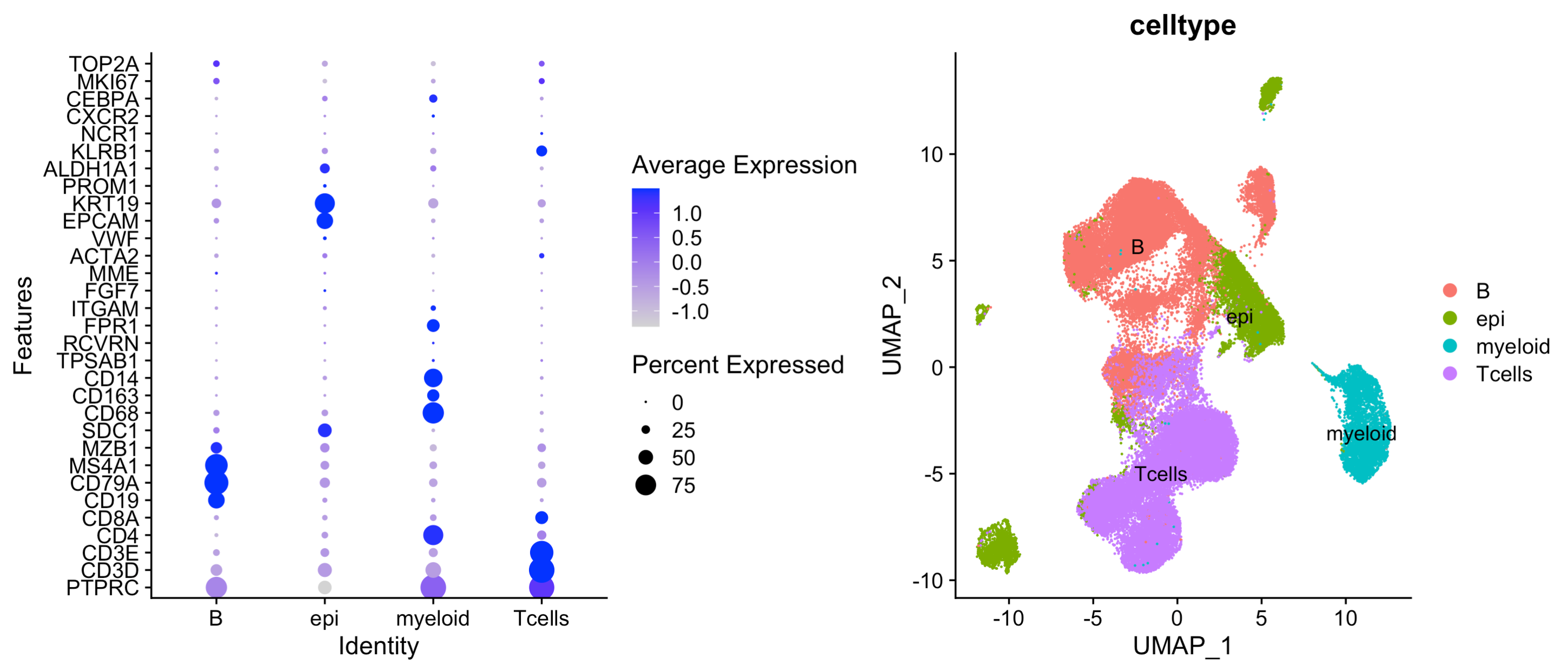
我们根据左边的标记基因以及生物学背景知识,可以进行如下所示的命名:
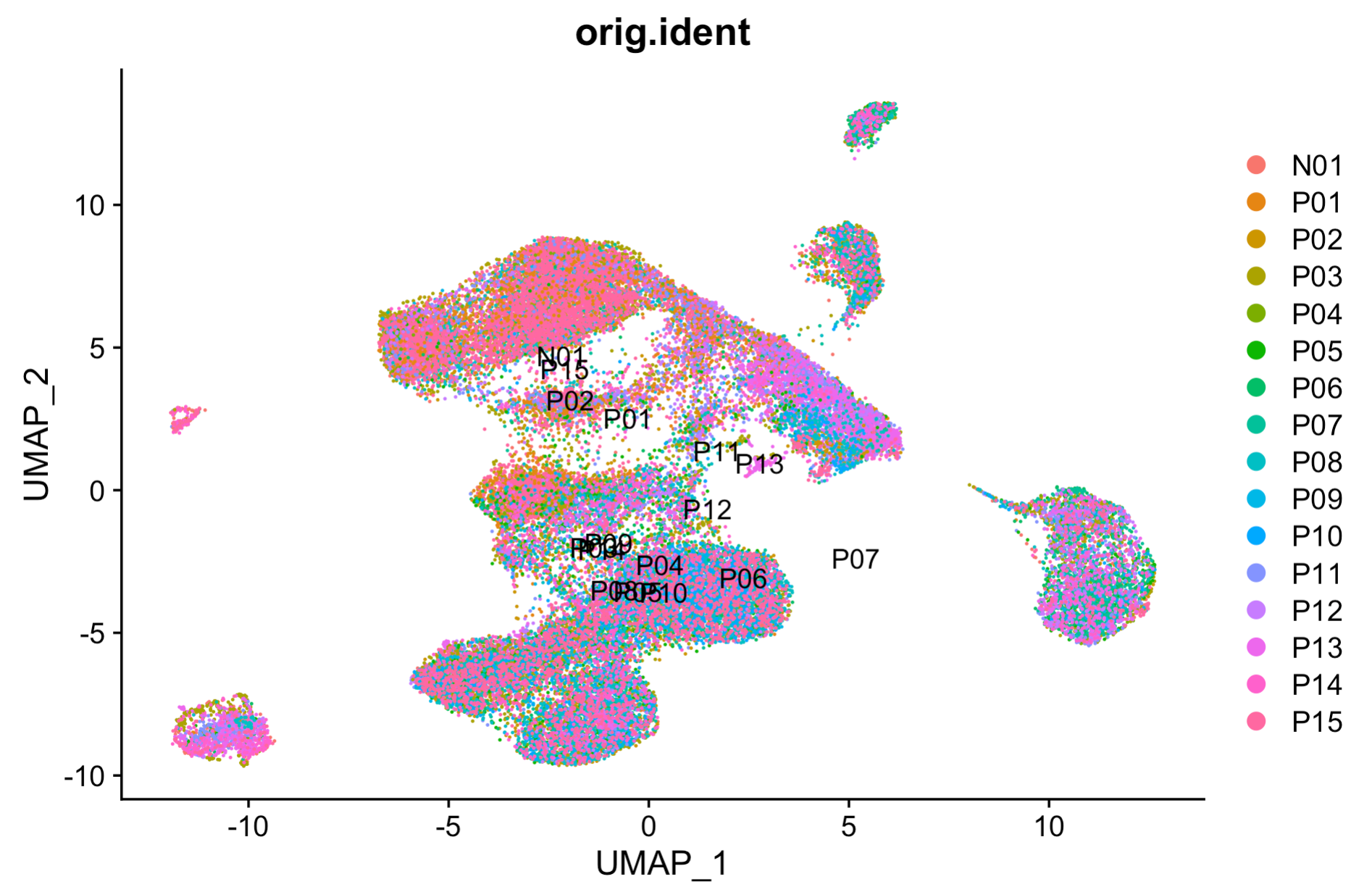
可以看到,效果还不错,很有意思, 给大家的感觉是 harmony等算法去除样品差异并不是必须的。但是如果我们具体到每个样品,有如下所示的现象:
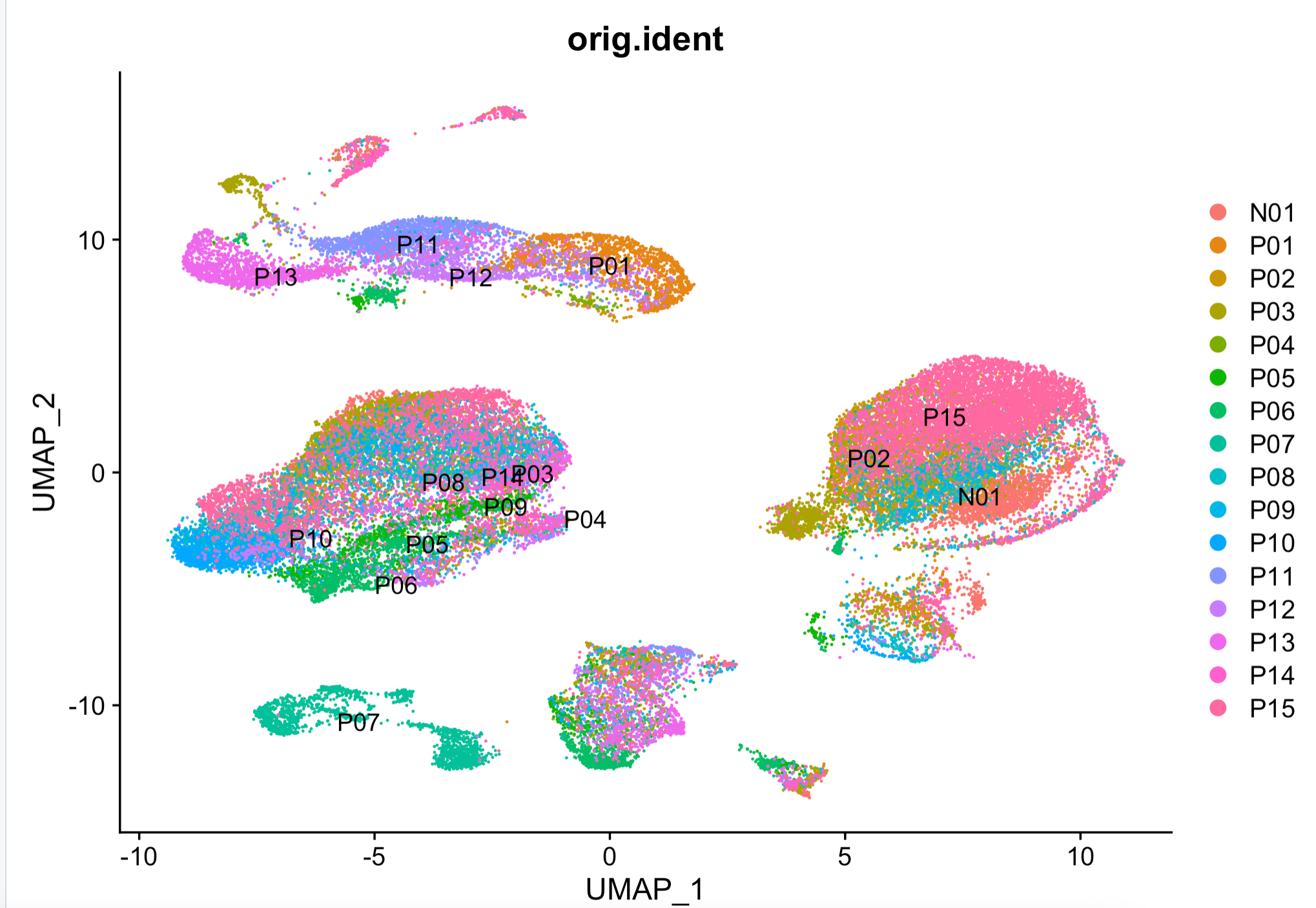
可以看到,首先上皮细胞大的亚群里面,每个病人独立成为小亚群,泾渭分明,这个符合预期,因为每个肿瘤病人都有自己的特异性。但是免疫细胞各个亚群里面,病人之间的界限就模糊很多。值得注意的是P07这个病人的样品,它主要是T细胞和髓系细胞,而且是独立成为一个亚群了,这就是单细胞转录组的样品差异,理论上是需要去除的!
有意思的事情就来了
如果我们在样品层面就开始使用harmony等算法去除样品差异,又会导致另外一个可怕的事情发生,如下所示:
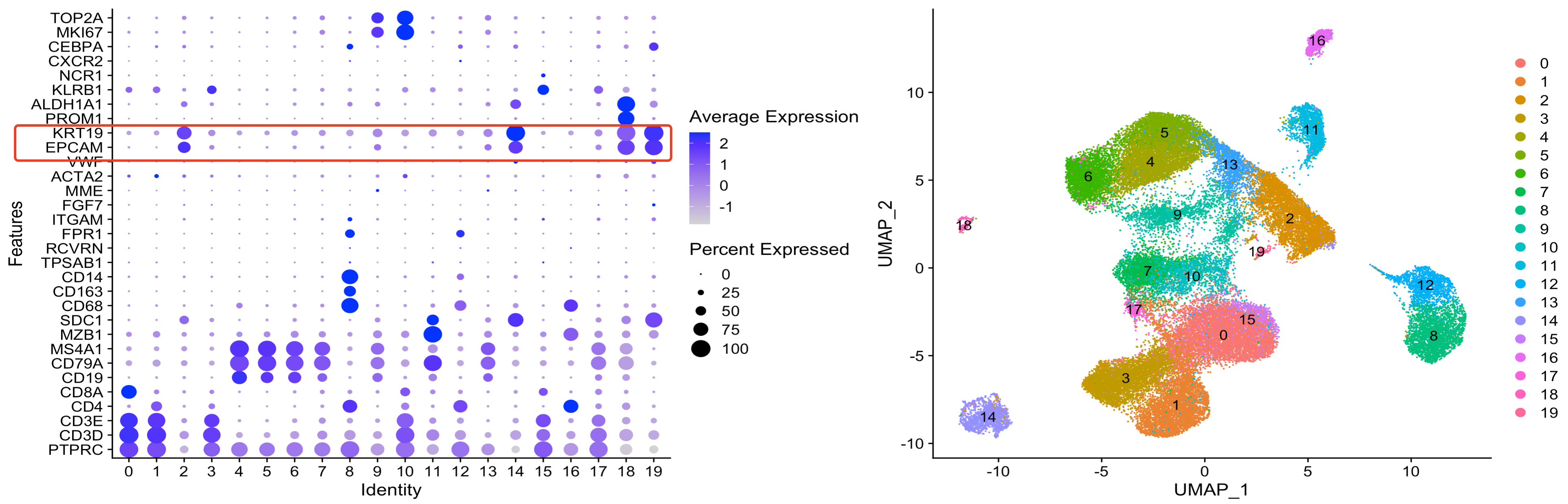
就是本来是应该是具备病人特异性的上皮细胞,这个时候被抹除了样品差异。
好好的上皮细胞,被拆分的七零八落,如下所示:
我们也可以以病人样品视角来看:
这个算法真的是太可怕了,样品差异被抹除的干干净净了!这不是最可怕的,真正的问题是,这个上皮细胞被打散到了其它免疫细胞里面,因为这个harmony算法!我们可以对上皮细胞的最重要的marker基因EPCAM进行如下所示可视化,并且使用harmony等算法去除样品差异前后可以对比看看。
如果你对单细胞数据分析还没有基础认知,可以看基础10讲: