绝大部分还没有接触单细胞的小伙伴很容易被无良自媒体带节奏,误以为单细胞就等价于CNS级别文章,或者再差也是子刊级别。实际上新技术的红利窗口期非常短暂,从样品量数据量的要求一直在水涨船高!比如我们看看某公司2020第四季度单细胞文章合辑,如下所示:
| 文章标题: | 影响因子: | 发表期刊: | 发表时间: | |
|---|---|---|---|---|
| Immunosuppressive effects of mesenchymal stem cells on lung B cell gene expression in LPS-induced acute lung injury | 5.116 | Stem Cell Research & Therapy | 2020.09.25 | |
| Single-cell transcriptome atlas of lung adenocarcinoma featured with ground glass nodules | 6.255 | Cell Discovery | 2020.10.06 | |
| Mesenchymal stem cells alleviate LPS-induced acute lung injury by inhibiting the proinflammatory function of Ly6C+ CD8+ T cells | 6.304 | Cell Death & Disease | 2020.10.06 | |
| Single-cell RNA sequencing highlights the role of inflammatory cancer-associated fibroblasts in bladder urothelial carcinoma | 12.121 | Nature Communications | 2020.10.08 | |
| Single-cell analysis of developing and azoospermia human testicles reveals central role of Sertoli cells | 12.121 | Nature Communications | 2020.10.10 | |
| Single-Cell RNA Sequencing Efficiently Predicts Transcription Factor Targets in Plants | 4.402 | Frontiers in Plant Science | 2020.12.08 | |
| Combinatorial Photothermal 3D‐Printing Scaffold and Checkpoint Blockade Inhibits Growth/Metastasis of Breast Cancer to Bone and Accelerates Osteogenesis | 16.836 | Advanced Functional Materials | 2020.12.13 | |
| The T Cell Receptor Immune Repertoire Protects the Liver From Reconsitution | 5.085 | Frontiers in Immunology | 2020.12.18 |
能发 Nature Communications 这样的子刊已经是佼佼者啦!而且这还是一年前,现在,单细胞基本上已经是“烂大街”了,去外面开会交流不说自己有单细胞项目都不好意思!
而且确实目前做一个单细胞项目从经费上来说已经不再是下血本了,比如某公司推出来的2个分组的单细胞项目标准分析,一般来说每个分组是3个单细胞10x样品,也就是说6个单细胞样品从送样,建库,测序(100G)数据量,平均每个10x的单细胞转录组样品出8000个左右的细胞。再加上降维聚类分群,生物学命名,看2个分组的不同细胞亚群比例差异,看同一个细胞亚群在2个分组是否有表达量上下调以及通路变化。这一套,就10万块钱!
我相信你肯定是有自己想探索的生命科学领域问题,如果你不知道这样的2个分组的单细胞项目标准分析可以出哪些图表,得到哪些生物学问题,这里可以跟我们一起读一下2021年5月发表的文章,标题是:《Single-cell RNA-seq reveals the transcriptional landscape in ischemic stroke》,它比较好的展现了单细胞这个计算可以做什么。
标准的2个分组的单细胞项目
实验设计如下所示:
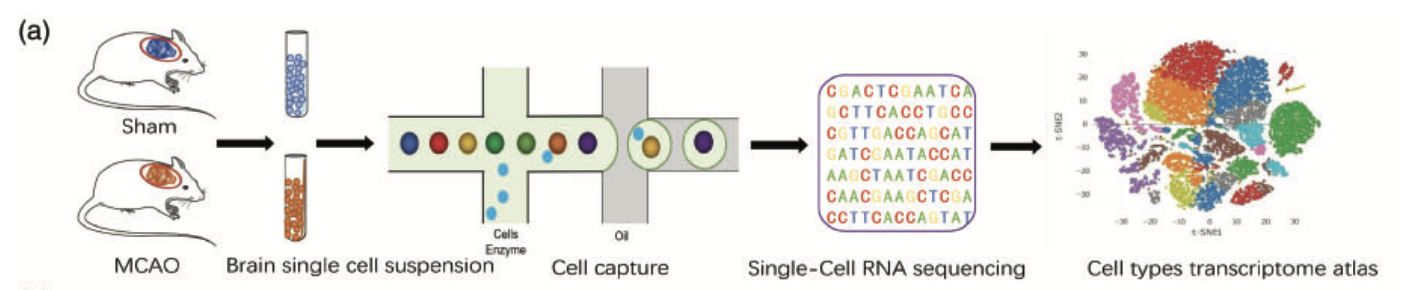
目前都是10x方法的单细胞转录组,全部自动化出数据,就可以进行标准分析啦!6个单细胞样品从送样,建库,测序(100G)数据量,平均每个10x的单细胞转录组样品出8000个左右的细胞。再加上降维聚类分群,生物学命名,看2个分组的不同细胞亚群比例差异,看同一个细胞亚群在2个分组是否有表达量上下调以及通路变化。这一套,就10万块钱!
降维聚类分群
初始降维聚类分群得到的是各个亚群编号,然后需要根据背景知识看各个亚群各自高表达量基因去给它生物学名字!
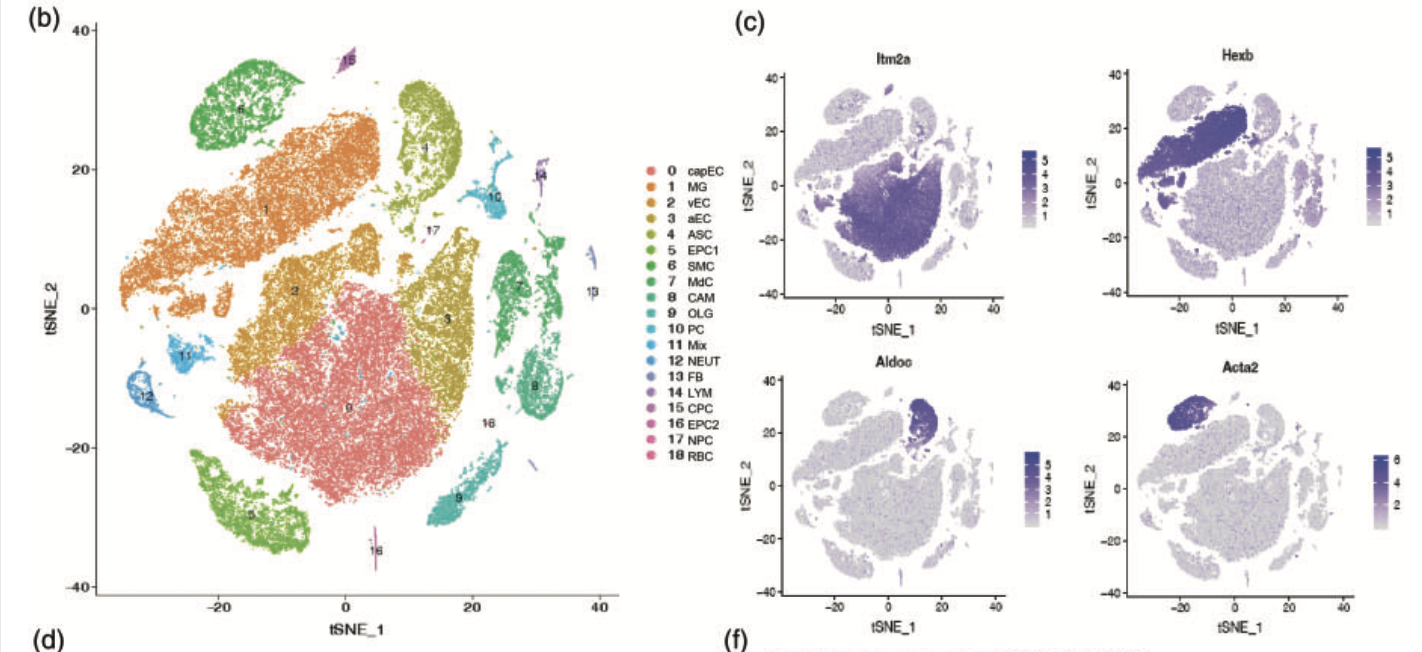
比如这个文章,就是得到如下所示的单细胞亚群 :
- vascular smooth muscle cells (SMC)
- perivascular fibroblast-like cells (FB)
- central nervous system (CNS)-associated macrophages (CAM)
- monocyte-derived cells (MdC)
- venous endothelial cells (vEC)
- capillary endothelial cells (capEC)
- arterial endothelial cells (aEC)
- pericytes (PC)
- choroid plexus capillary endothelial cells (CPC)
- ependymocytes (EPC)
- microglia (MG)
- neutrophils (NEUT)
- astro- cytes (ASC)
- oligodendrocytes (OLG)
- neural progeni- tor cells (NPC);lymphocytes(LYM)
- red blood cell (RBC).
单细胞转录组数据分析的标准降维聚类分群,并且进行生物学注释后的结果。可以参考前面的例子:人人都能学会的单细胞聚类分群注释 ,我们演示了第一层次的分群。
如果你对单细胞数据分析还没有基础认知,可以看基础10讲:
细胞亚群比例差异
细胞亚群可以按照之前的编号去看细胞比例,也可以根据生物学名字看差异,各个细胞亚群也有各自的top基因列表和生物学功能富集列表。
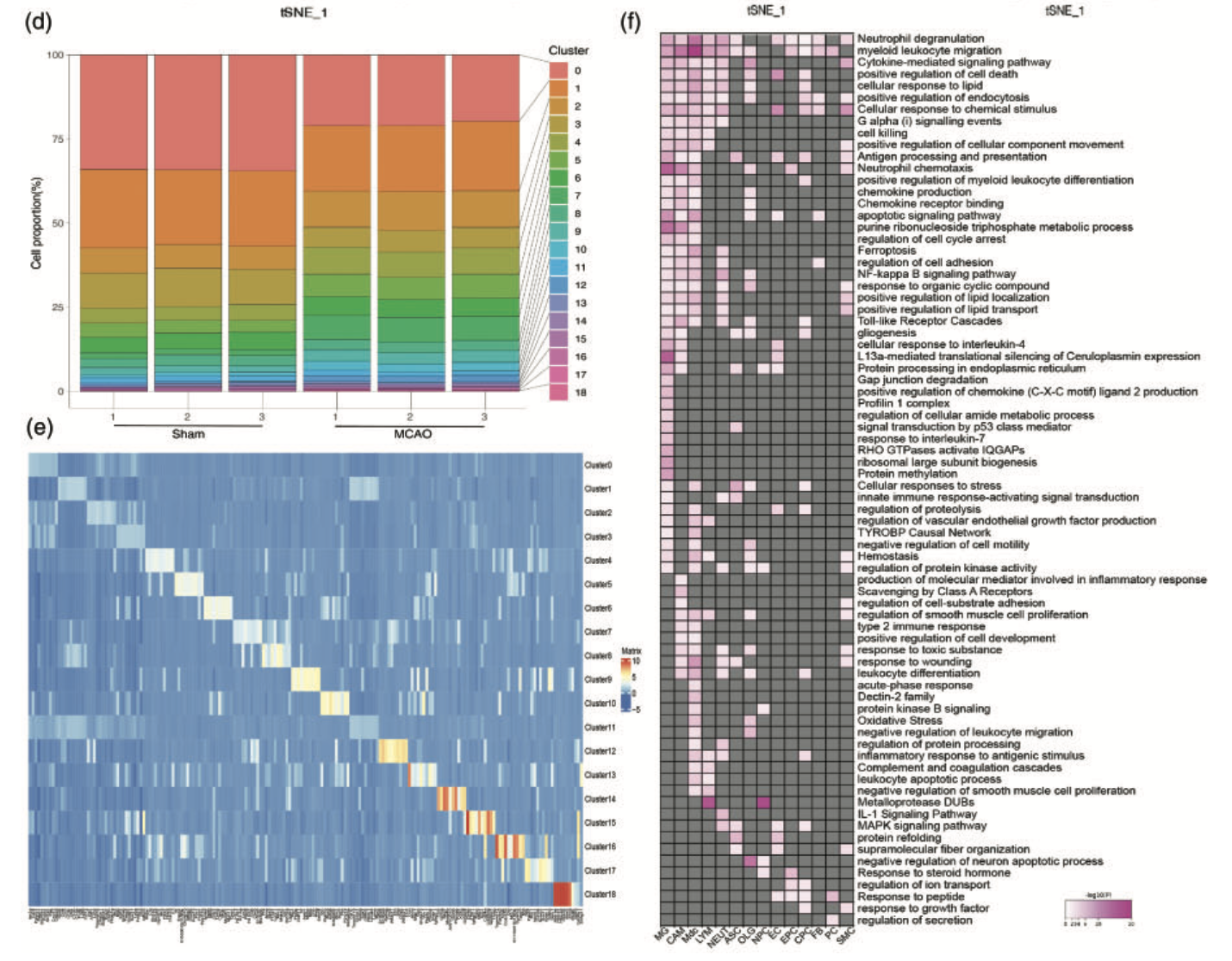
这个文章的图表非常的粗糙,如果我们来展现各个单细胞亚群的细胞比例变化,会比它好看百倍!这里作者使用的gsva和gsea这样的策略,GSEA分析,我也多次讲解:
但实际上,绝大部分读者并没有去细看这个统计学原理,也不需要知道gsea分析的nes值如何计算,需要知道的是nes值的生物学意义。
而单细胞转录组数据的批量GSVA代码我在:借鉴escape包的一些可视化GSVA或者ssGSEA结果矩阵的方法 剧透过,其实跟 对单细胞表达矩阵做gsea分析的代码实现环节大同小异。无论是gsva还是gsea,本质上都是看某个生物学基因集的得分。
各自细胞亚群在2分组的表达量差异分析
前面的数据分成了十多个细胞亚群,就可以在2分组做十多次差异分析,每次分析都有上下调基因列表。多个列表就可以取交集:
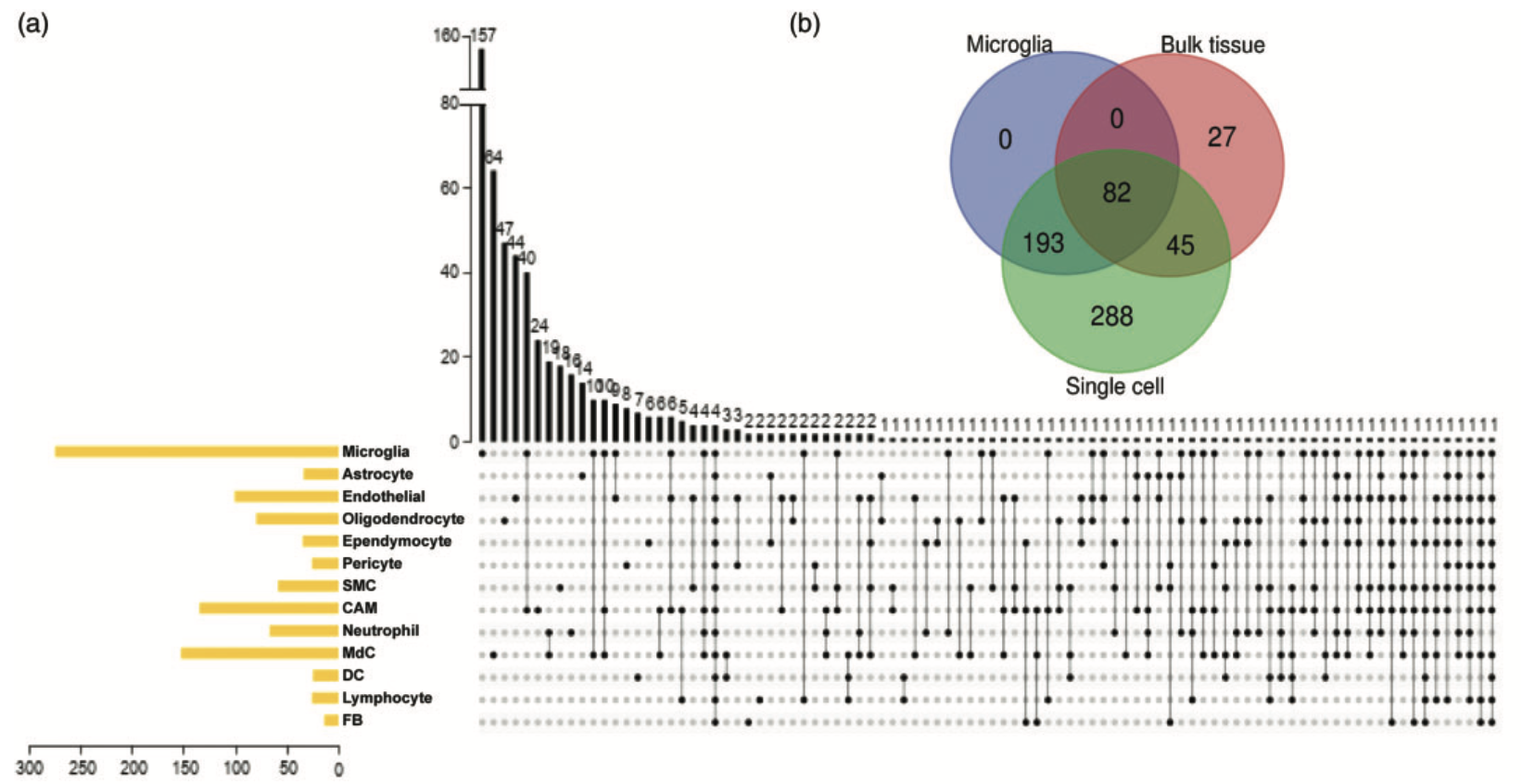
这个图看起来平平无奇,其实背后的数据分析工作量还是有的!至少需要把每个单细胞亚群独立提取,然后进行重新鸡尾酒疗法并且根据生物学2分组进行差异分析,并且对多个差异分析的上下调基因进行交集展现。
细分亚群继续降维聚类分群和差异
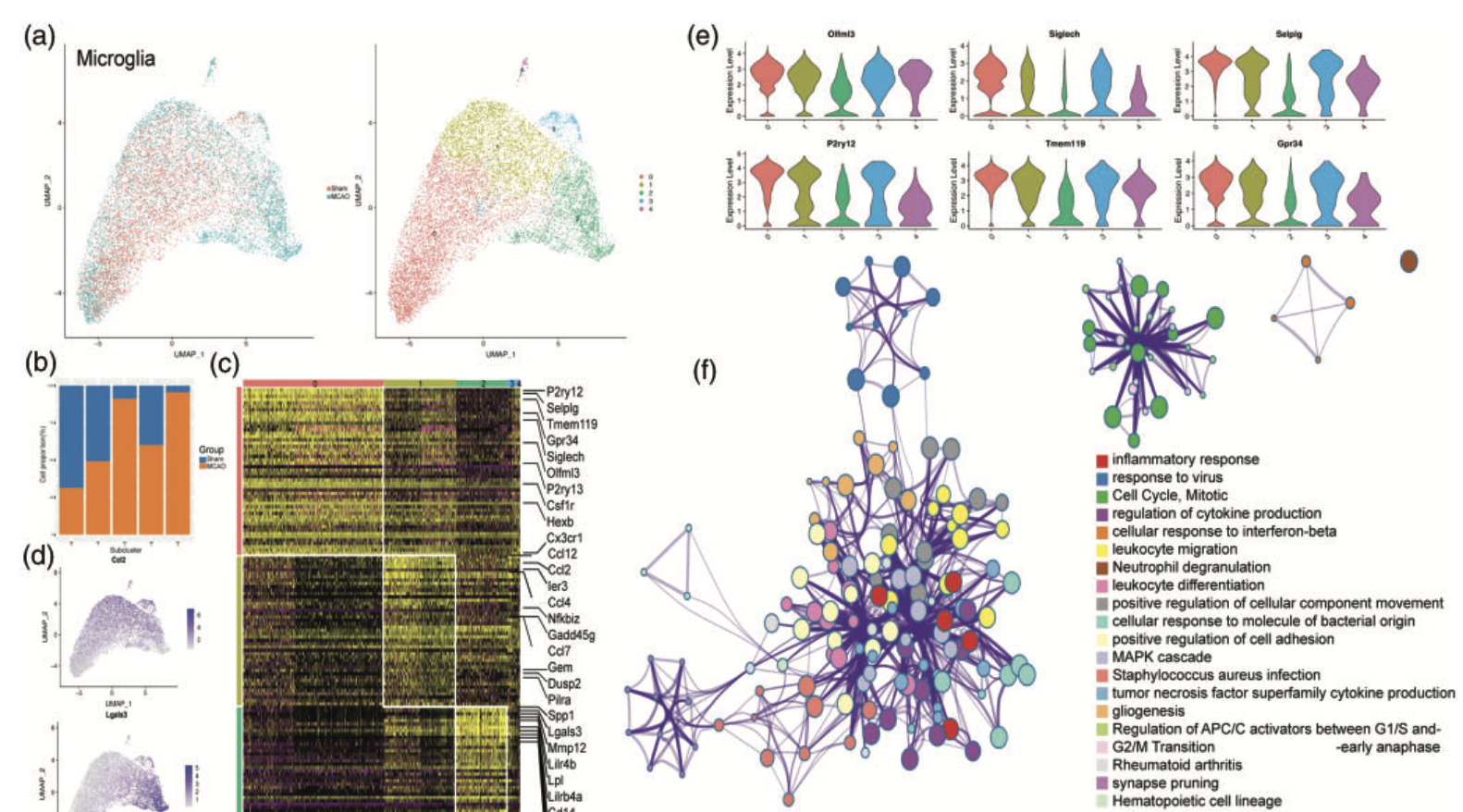
比如本文就是进行了研究者自己感兴趣的4个单细胞亚群的细分研究 :
- Figure 4. Single-cell transcriptome analysis of the microglia.
- Figure 5. Single-cell trajectory analysis of monocyte/macrophage clusters.
- Figure 6. Differential gene expression patterns in neutrophils clusters.
3大主流高级分析有一个即可
这个文章选择的是细胞通讯:Figure 7. Comparative cell-cell interactions in sham and MCAO mice.
其它高级分析,比如转录因子分析 ,主要是对计算机资源消耗会比较大,我也多次分享过细节教程: