ngs技术多如牛毛,在它之前每个技术都有一个对应的芯片版本。
比如RNA-seq的技术基本上取代了affymetrix的表达量芯片,但是甲基化测序技术,无论是WGBS还是RRBS都无法取代illumina公司的甲基化芯片,反而是其自己从27K进化到了450K,以及目前的850K甲基化芯片。每个技术在火爆之前都是一个红利期,那个时候只需要一个简单的实验设计,使用了最新技术,进行了基础分析就可以发表在很好的杂志。
比如发表于《HEPATOLOGY》杂志的2012的文章:《 Genome-wide DNA Methylation Profiles in Hepatocellular Carcinoma 》,就是使用了 illumina公司的 27K芯片,针对:
- 62 HCC Patients With Tissue DNA Methylation Array Data
- 8 Additional Cases With Only Plasma DNA Data
其实他并不算是第一个吃illumina公司的甲基化芯片红利的团队,文章自己也提到了已经发表了的类似的4个研究: - Two studies used Illumina 1,500 Golden Gate arrays on five paired samples from Korea and 30 from France
- the third used Illumina Human Methylation27K arrays on 12 samples from Germany
- methylated CpG-island amplification microarrays to study 6,458 CpG islands in 10 paired samples from Japan
涵盖了科研重地,比如韩国,法国,德国,日本。所以作者的普通实验设计想吃红利,就还有个创新点,人种。哈哈哈哈,The current study is the largest to date and is com- prised of Taiwanese cases who are predominantly HBV positive.
算了,不嘲笑人家了,毕竟他还是发表在了《HEPATOLOGY》杂志,更多的无名英雄其实勇于尝试了新技术,可惜啊,不懂得珍惜或者寻找新技术数据分析人才,所以把数据束之高阁,暴殄天物啊,折戟沉沙也是意料之中。
言归正传,让我们看看研究者们对这样的一个简单的二分组实验设计做了哪些分析吧!
当然少不了简单的差异分析啦:
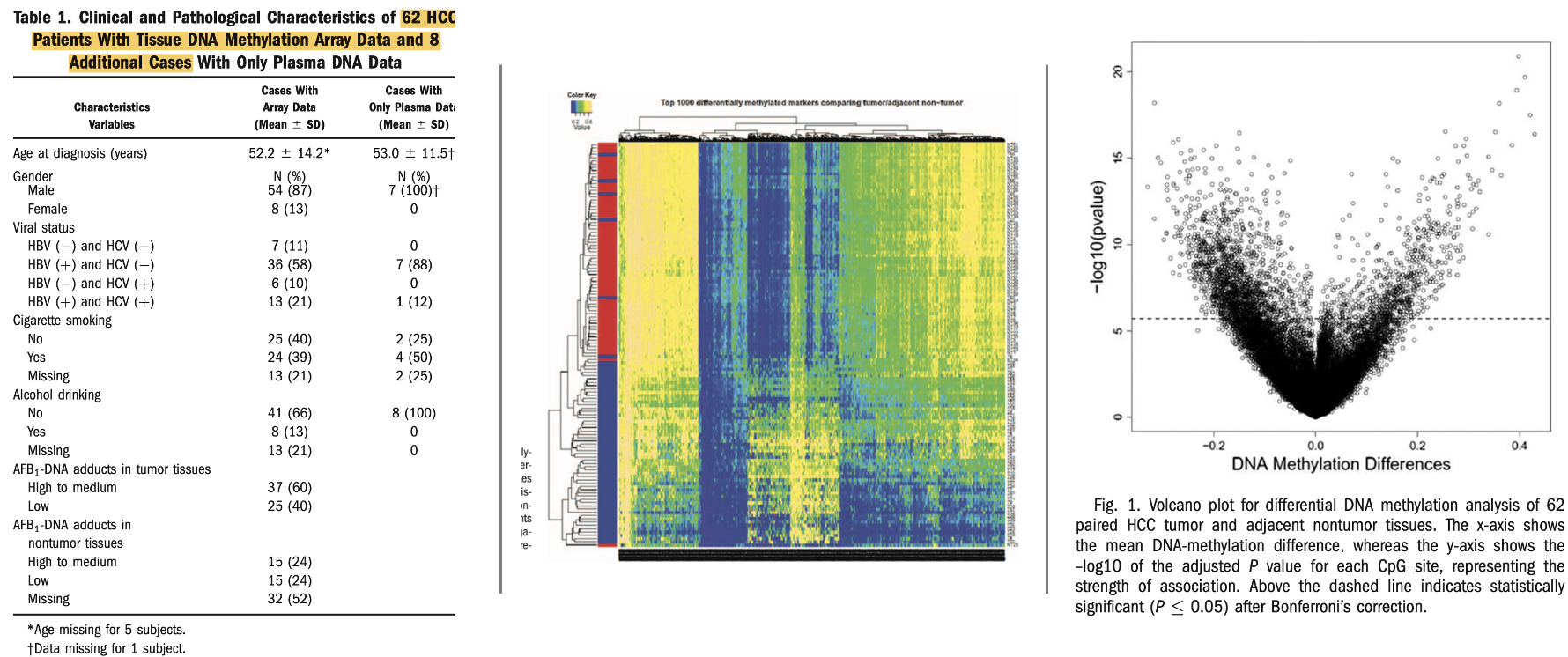
可以看到,对26,486 autosomal CpG sites, tumor, compared to nontumor, tissues. 得到了: - 684 were significantly hyperme- thylated (covering 548 genes)
- 1,640 were signifi- cantly hypomethylated (covering 1,290 genes)
有了差异甲基化位点及其对应的基因,可以进行功能注释,也做了列表展现出来 。
然后就没有了。学徒作业
去TCGA数据库看这个 HCC 癌症的甲基化芯片数据,从里面挑选全部的正常样品的甲基化数据集和对应数量的HCC癌症的甲基化芯片数据,走前面介绍的文献里面的流程,拿到同样的图表!
甲基化背景知识
甲基化测序的 WGBS和RRBS,还有 芯片是最高频的甲基化技术,其中甲基化芯片数据处理我是有视频课程的,首先需要阅读我在生信技能树的甲基化系列教程,目录如下:
- 01-甲基化的一些基础知识.pdf
- 02-甲基化芯片的一般分析流程.pdf
- 03-甲基化芯片数据下载的多种技巧.pdf
- 04-甲基化芯片数据下载如何读入到R里面.pdf
- 05-甲基化芯片数据的一些质控指标.pdf
- 06-甲基化信号值矩阵差异分析哪家强.pdf
- 07-甲基化芯片信号值矩阵差异分析的标准代码.pdf
- 08-TCGA数据库的各个癌症甲基化芯片数据重新分析.pdf
- 09-TCGA数据库的癌症甲基化芯片数据重分析.pdf
- 10-TCGA数据辅助甲基化区域的功能研究.pdf
- 11-按基因在染色体上的顺序画差异甲基化热图.pdf
- 850K甲基化芯片数据的分析.pdf
- 使用DSS包多种方式检验差异甲基化信号区域.pdf
然后就可以看我在B站免费分享的视频课程《甲基化芯片(450K或者850K)数据处理 》 - 教学视频免费在:https://www.bilibili.com/video/BV177411U7oj
- 课程配套思维导图:https://mubu.com/doc/1cwlFgcXMg