看到了一个 J Cancer . 2020 Jan的文献蛮有意思的,文章标题是:《A Stemness and EMT Based Gene Expression Signature Identifies Phenotypic Plasticity and is A Predictive but Not Prognostic Biomarker for Breast Cancer》,也就是说,标题就指出来了他们的研究对象 癌细胞干性指数以及EMT打分,这两个指标都跟癌症预后没有关系!
是不是很惊喜!
标题其实很有意思,研究者得到的基因集是 Predictive but Not Prognostic Biomarker!
其中 癌细胞干性指数以及EMT打分的背景知识大家需要自行去搜索学习一下:
- EMT (epithelial to mesenchymal transition) state of tumors
- CSC(cancer stem cell)-like and non-CSC-like
作者的研究重点是a novel robust gene signature ,简称和缩略词是:CSC/non-CSC gene list (CNCL) ,可以把乳腺癌细胞系区分成为:
- cancer-stem-cell-like and mesenchymal (CS/M)
- non-cancer-stem-cell-like and epithelial (NS/E)
这样的研究基于一个假说,就是non-CSC cells would be generated from CSC cells. 而且CD44+/CD24- cells that possess the capacity of self-renewal,所以简化成为:
- CD44+/CD24- stem cells
- CD44-/CD24+ non stem cells
不过并不是所有人都认可癌症干细胞这个理论哦,但是因为癌症干细胞跟癌症发生发展,转移,甚至治疗耐药有密切 关系,所以这方面的研究也是多如牛毛。
在两个细胞系药物数据库找共有差异基因
提到细胞系药物作用数据库,最出名的是 Cancer Cell Line Encyclopedia (CCLE) 和 Cancer Genome Project (CGP) / Genomics of Drug Sensitivity in Cancer (GDSC) 这两个:
- CCLE (www.broadinstitute.org/ccle/)
- GDSC/CGP (www.cancerrxgene.org/)
其实还有broad的 CTRP和Roche的geneTech公司的数据库也类似两个细胞系药物数据库,这里先略过。
很简单,把两个数据库里面的乳腺癌细胞系区分 成为干性与否,进行差异分析即可:
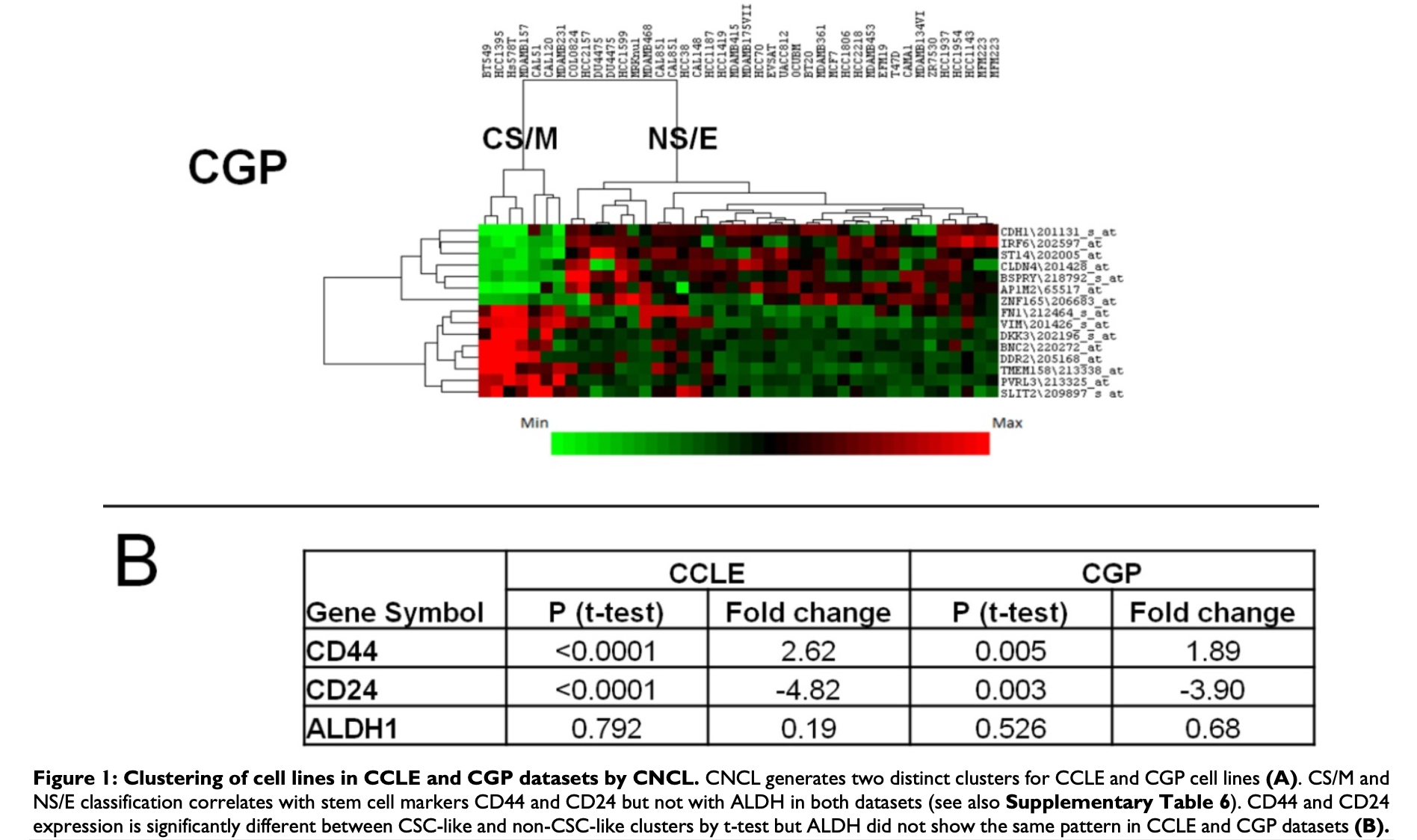
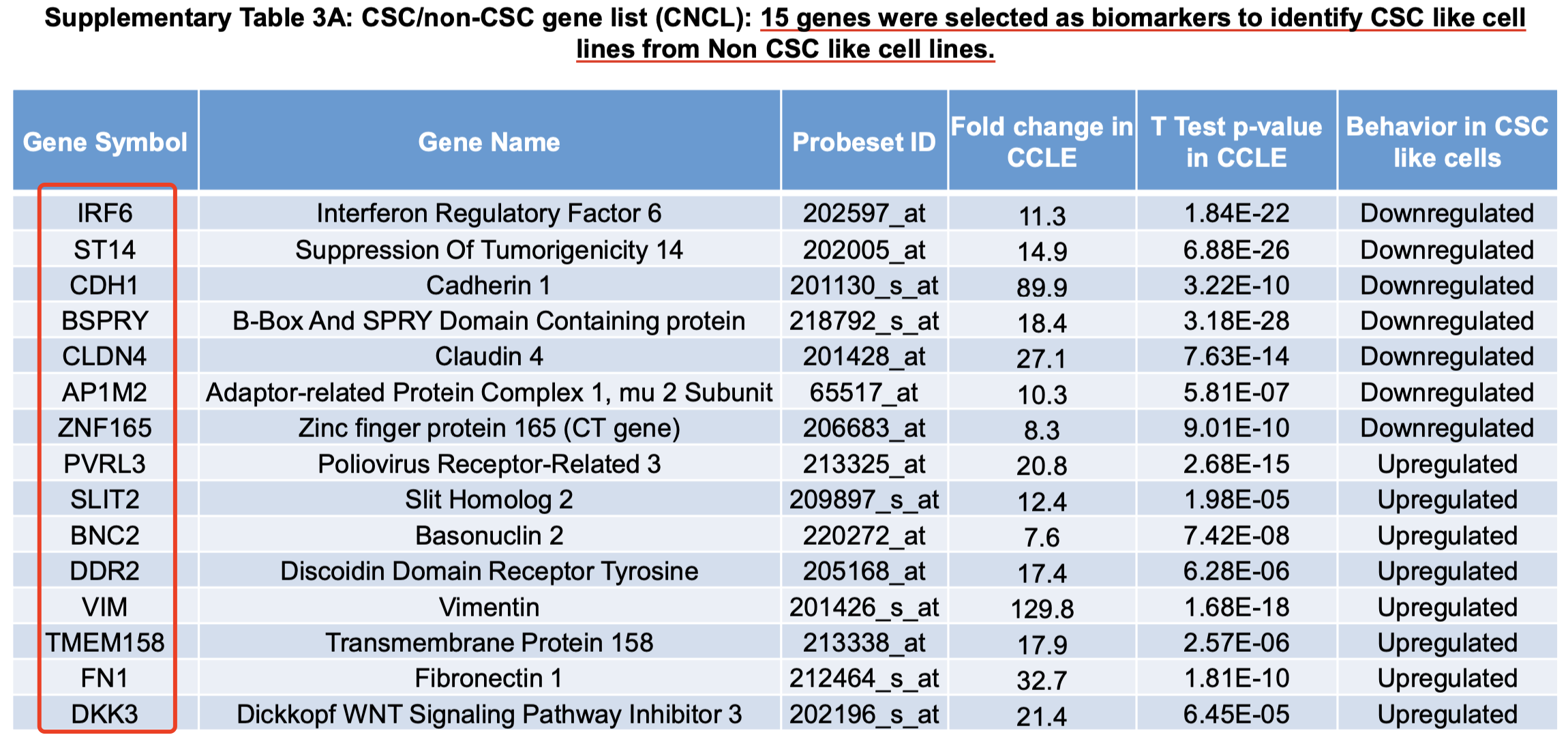
大家可以自己去下 GSE36139 (CCLE).和E-MTAB-783 (CGP). 的表达量矩阵,试试看这个差异分析。两次差异分析各自选取top200探针,然后有129个交集,对应97个基因,最后提高阈值,后 得到 15 genes/probesets (8 up and 7 down-regulated in CSC-like cell lines), 作为最后的CSC/non-CSC gene list (CNCL).基因集。全部的基因如下所示:
而且这15个基因集比较好的在其它数据集里面得到了验证,包括:
- GSE15192
- GSE36643
- GSE52327
多个gse数据集验证基因集的临床预后能力
这些数据集涉及到了多种临床结局:
- DSS: disease specific survival
- OS: Overall survival
- RFS: Relapse free survival
- BR: Bone relapse
- MFS: metastasis free survival
- LNMFS: Lymph node metastasis free survival
- BMFS: Bone metastasis free survival
- DMFS: distant metastasis free survival
- DRFS: Distant relapse free survival
- DFS: disease free survival
- TDM: time to distant metastasis
- ND: not disclosed
详细信息如下所示:
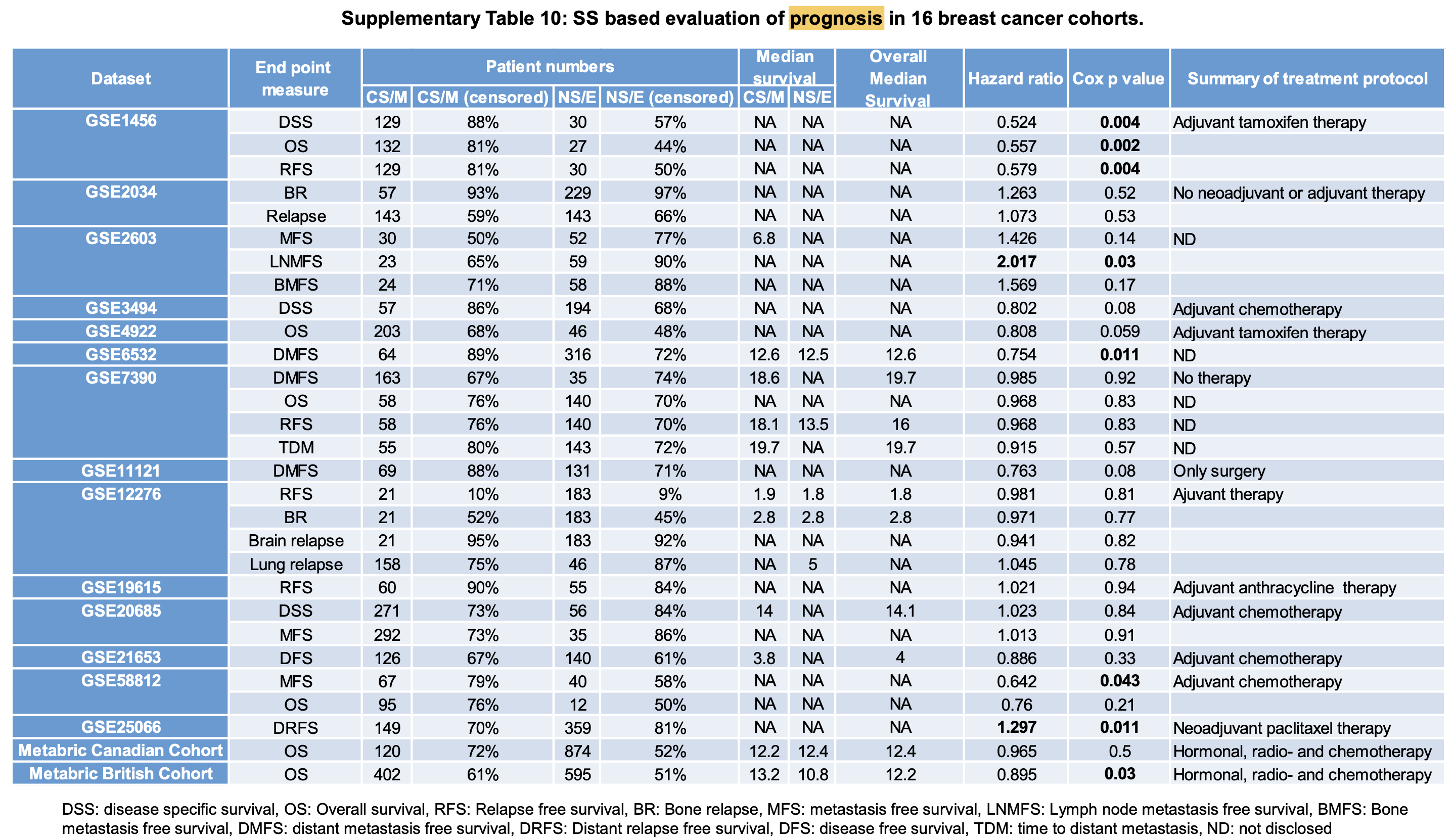
数据集虽然只有15个,但是有几个数据集有多种结局事件信息,所以总体上是29次生存分析检验。
但是有统计学显著的就7次,其中GSE25066 和 GSE2603数据集里面,该基因集是风险因子(CS/M tumors 组 死得快 ),但是在其它数据集是保护因子。
而GSE25066数据集是一个Taxane (incl. Paclitaxel) 的新辅助化疗队列,NS/E tumors 的distant relapse free survival 时间要长一点,也就是说 该基因集是风险因子(CS/M tumors 组 死得快 )。
也就是说,这个基因组在不同数据集的预后能力不一致,但是在Taxene的新辅助化疗队列里面还是有一定作用。
生存分析是目前肿瘤等疾病研究领域的点睛之笔
我在生信技能树多次分享过生存分析的细节;
- 人人都可以学会生存分析(学徒数据挖掘)
- 学徒数据挖掘之谁说生存分析一定要按照表达量中位值或者平均值分组呢?
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值