目前Seurat都已经更新到了V5,假如你不喜欢最新版的Seurat包的单细胞理念,我也是在几百个交流群里面被成百上千人问到了它V5版本的数据结构问题,烦不胜烦啊。我很清晰的摆明了自己的态度, 就是不喜欢它这个“重大”升级,而且我也号召了自己的粉丝们半年内不要升级它:它想强迫我升级一系列seurat相关的单细胞R包
主要是因为我们依赖于这个V4的版本的Seurat流程做出来了大量的公共数据集的单细胞转录组降维聚类分群流程,100多个公共单细胞数据集全部的处理,链接:https://pan.baidu.com/s/1MzfqW07P9ZqEA_URQ6rLbA?pwd=3heo,而且也有海量的配套视频教程在b站,视频号等渠道,基本上大家能看到的中文笔记都是我们分享的。。。。
但是,我表明态度的多个推文里面都被“匿名者”阴阳怪气的怼了一下,说我这样的写教程(英文教程搬运工)的渣渣没有资格骂开发者。。。
单细胞不等于单细胞转录组
单细胞研究可以涉及多个层面,包括但不限于:
- 细胞形态学特征: 单细胞研究可以包括对细胞形态学特征的观察,例如细胞大小、形状等。
- 基因组: 单细胞基因组学研究可以揭示细胞的遗传信息,例如突变、拷贝数变异等。
- 表观基因组: 研究细胞的表观基因组学变化,如DNA甲基化状态。
- 蛋白质组: 单细胞蛋白质组学研究关注细胞内蛋白质的表达水平和变化。
- 转录组: 揭示单个细胞的基因表达谱,即单细胞转录组。
但是”单细胞转录组”则是单细胞层面上的基因表达谱。它通过测量单个细胞中基因的表达水平来揭示细胞在基因表达层面的异质性。单细胞转录组学是近年来发展迅速的技术,它使用高通量测序技术,例如单细胞RNA测序(scRNA-seq),以获取每个细胞中所有基因的表达水平。这样的整理汇总单细胞层面的多种组学的综述文献有很多,比如:《Single-cell sequencing technology applied to epigenetics for the study of tumor heterogeneity》
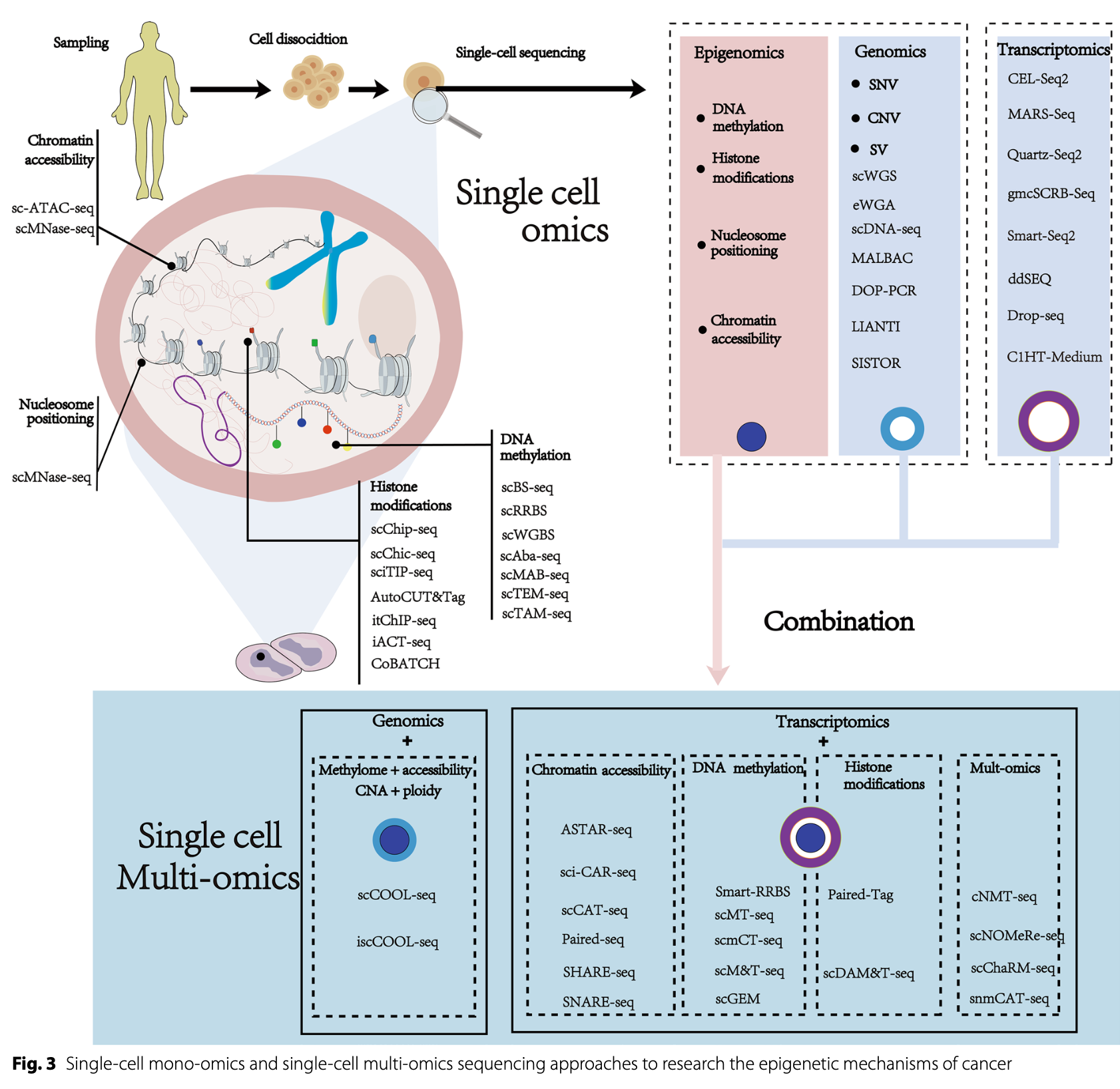
单细胞转录组不等于10x
虽然说单细胞不等于单细胞转录组,但是绝大部分人接触到的就是单细胞转录组而已。而且Smart-seq2和10x这两个单细胞转录组技术是现在初学者进入单细胞领域最需要掌握的,它们代表着单细胞转录组的两个全然不同的发展策略。绝大部分的技术原理介绍会从 单细胞悬浮液制备到测序细节面面俱到,其实并不那么的初学者友好,最近有粉丝在公众号后台留言说他们的博士课程有一个思考题是:简要概述smart-seq2和10x技术的单细胞差异。然后他给大家推荐了一个高度精炼的综述,这个综述于2020年9月发表在 《Experimental & Molecular Medicine》杂志,标题是:《Single-cell sequencing techniques from individual to multiomics analyses》,链接是:https://www.nature.com/articles/s12276-020-00499-2
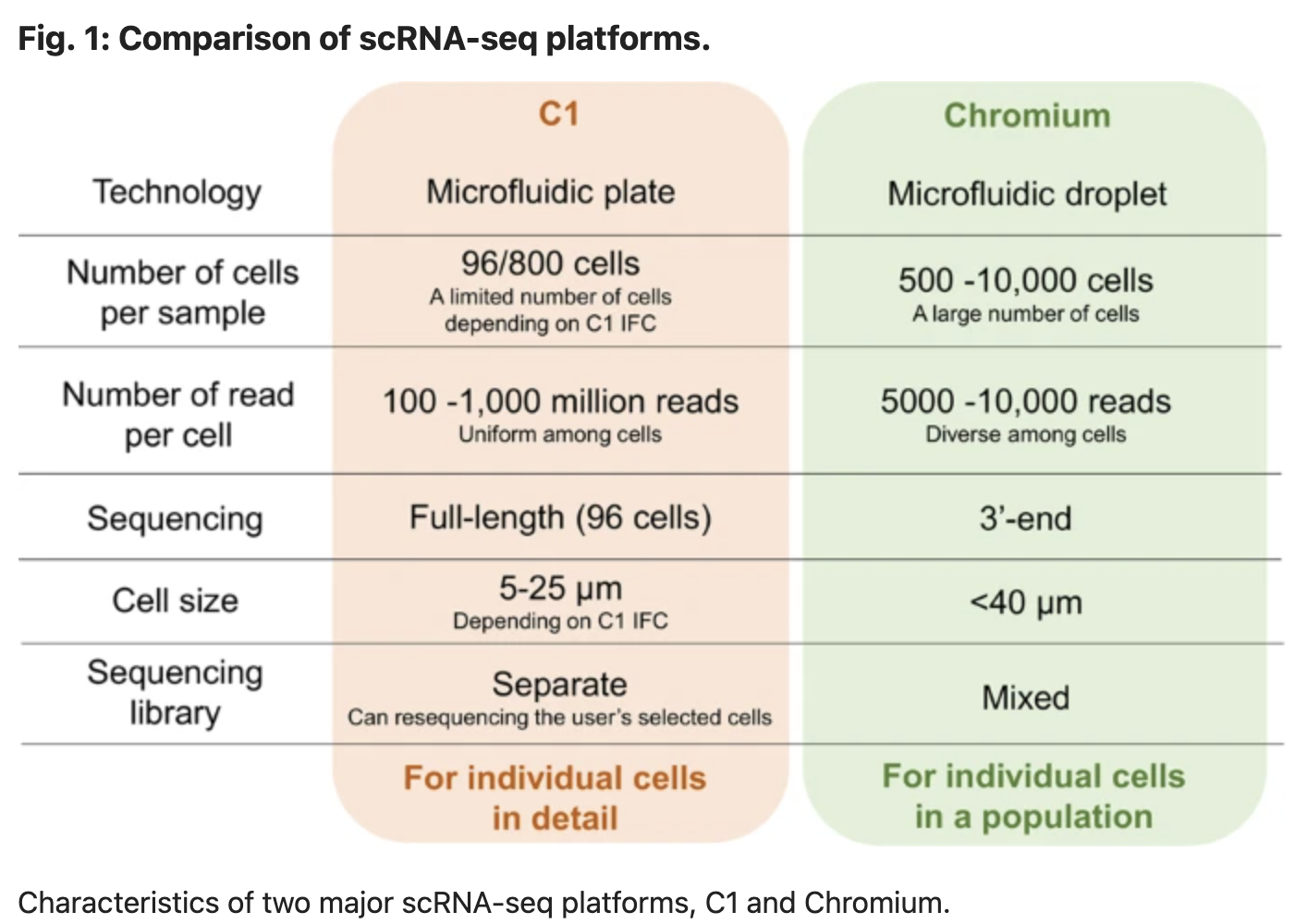
可以看到: - smart-seq2技术依赖于C1这个仪器,每次都是96个细胞一起测序,每个细胞的测序量这个综述可能是写错了,应该是1M-10M为佳,不太可能是100-1000个M,最重要的是它是整个RNA分子的全长测序,每个细胞都是独立的测序。
- 但是10X呢,每次可以测好几千的细胞,每个细胞只需要5-10K的reads,而且仅仅是测RNA分子的一段即可,全部的细胞都混合在一起,虽然说有barcode可以区分。
单细胞转录组数据分析不等于Seurat流程
前面提到的Smart-seq2和10x当然并不是仅有的单细胞转录组技术了,同样的大家可以任意搜索最新综述都可以看到汇总好的技术大全的列表,但是还是那句话,对初学者来说单细胞转录组其实就等于10x技术,其它都可以在入门学习过程中忽略掉。同样的道理,单细胞转录组数据分析也是不等于Seurat流程,但是因为有我们生物信息学自媒体的推广,最基础的往往是降维聚类分群,参考前面的例子:人人都能学会的单细胞聚类分群注释 ,详细的拆分成为基础10讲:
- 01. 上游分析流程
- 02.课题多少个样品,测序数据量如何
- 03. 过滤不合格细胞和基因(数据质控很重要)
- 04. 过滤线粒体核糖体基因
- 05. 去除细胞效应和基因效应
- 06.单细胞转录组数据的降维聚类分群
- 07.单细胞转录组数据处理之细胞亚群注释
- 08.把拿到的亚群进行更细致的分群
- 09.单细胞转录组数据处理之细胞亚群比例比较
虽然说,单细胞转录组数据分析也是不等于Seurat流程,但是最后的结果仍然是Seurat一家独大。最后为什么是Seurat一家独大呢
我大胆的推测,就是因为我们生物信息学自媒体的推广,我们大力宣传生物信息学入门编程语言是R语言,虽然说基于R语言的单细胞转录组数据分析也有大量的其它类似于Seurat的流程。比如
scater,这个也是用于单细胞RNA测序数据的包,主要聚焦于质控、可视化和探索性数据分析。它提供了一套工具,帮助研究人员理解和处理单细胞数据。 - 主要特点:
- 质控和过滤: 提供一系列函数进行细胞和基因的质控和过滤。
- 可视化: 提供丰富的绘图工具,包括散点图、箱线图、热图等,方便用户进行数据可视化。
- 探索性分析: 具备一些探索性数据分析的功能,例如主成分分析(PCA)和聚类分析。
- 集成其他包: 能够方便地与其他单细胞分析包(如
SingleCellExperiment)进行集成。
- GitHub 地址: davismcc/scater