最近授课转录组数据分析实战的时候, 到了下游分析环节,主要是表达量矩阵质量控制,差异分析,以及后续的生物学功能数据库注释。
学员提出来了一个很有意思的问题, 就是表达量矩阵质量控制环节里面的PCA分析,可以看出来不同样品在二维图里面的距离,其中PC1是可以区分两个分组,如下所示:
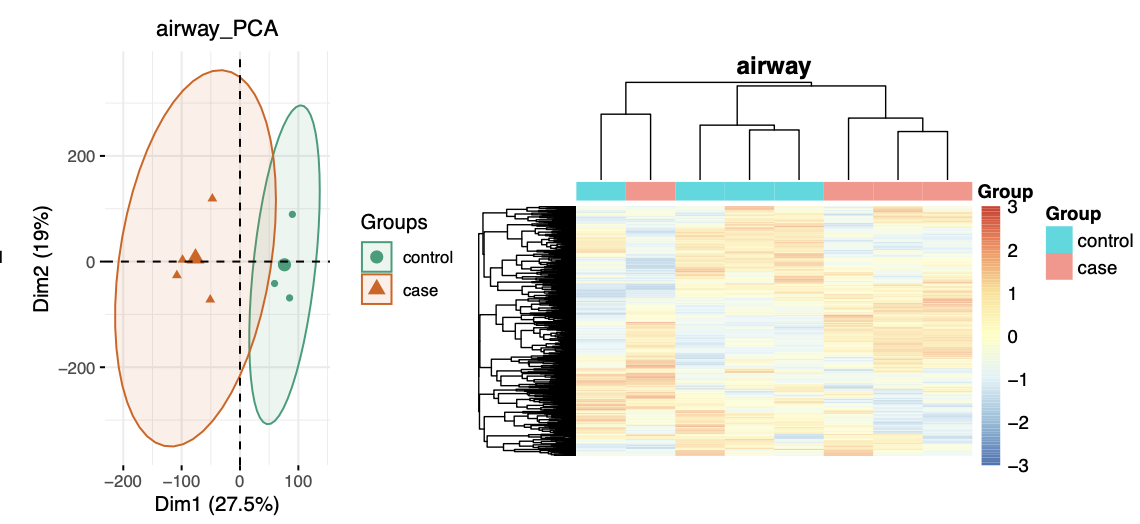
而我们做这两个分组的完差异分析后,我们的基因也是根据它在两个分组的差异程度排序的,如下所示:
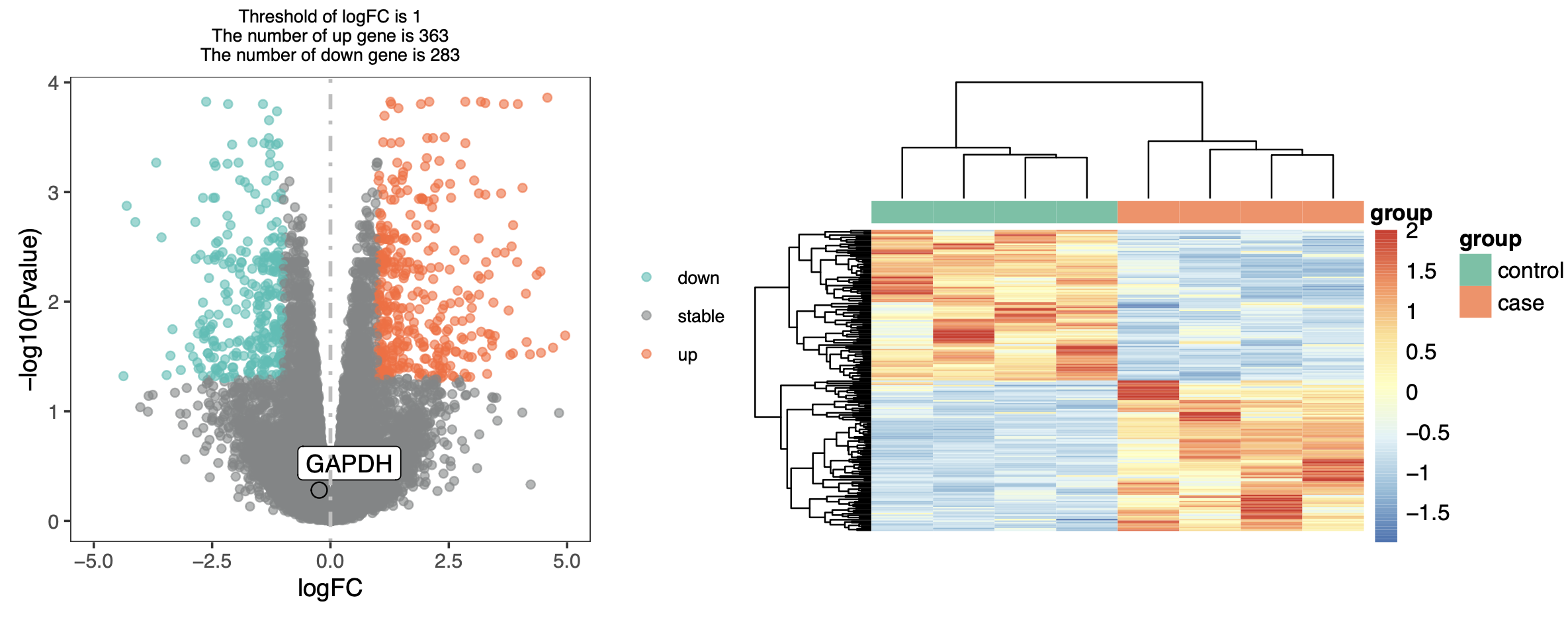
这两个分析看起来是“风马牛不相及”,但是学员还是感兴趣两分组差异分析的上下调基因跟PCA分析的主成分基因的交集如何呢?
我们还是以代码来说明这个问题:
首先是表达量矩阵的PCA分析
如下所示的代码:
rm(list = ls())
options(stringsAsFactors = F)
library(ggplot2)
library(ggstatsplot)
library(ggsci)
library(RColorBrewer)
library(patchwork)
library(ggplotify)
library("FactoMineR")#画主成分分析图需要加载这两个包
library("factoextra")
library(airway,quietly = T)
data(airway)
mat <- assay(airway)
mat[1:4,1:4]
keep_feature <- rowSums (mat > 1) > 1
mat <- mat[keep_feature, ]
dat = log2(edgeR::cpm(mat)+1)
dat[1:4,1:4]
dat.pca <- PCA(as.data.frame(t(dat)) , graph = FALSE)
dim(dat.pca$var$coord)
head(dat.pca$var$coord)
然后是默认的差异分析代码:
这里使用DESeq2包即可:
library(DESeq2)
exprSet = mat
group_list = airway@colData$dex
(colData <- data.frame(row.names=colnames(exprSet),
group_list=group_list) )
dds <- DESeqDataSetFromMatrix(countData = exprSet,
colData = colData,
design = ~ group_list)
dds <- DESeq(dds)
res <- results(dds )
resOrdered <- res[order(res$padj),]
DEG_deseq2 = na.omit(as.data.frame(resOrdered))
最后是比较两分组差异分析的上下调基因跟PCA分析的主成分基因,已经可视化
代码如下所示:
tmp = dat.pca$var$coord
colnames(tmp)
ids=intersect(rownames(DEG_deseq2),
rownames(tmp))
df = data.frame(logFC = DEG_deseq2[ids,'log2FoldChange'],
pca = tmp [ids, "Dim.1"])
ggplot(data = df,
aes(x = logFC,
y = pca)) +
geom_point(alpha=0.6, size=1.5 ) +
geom_point(size = 3, shape = 1 ) +
geom_vline(xintercept= 0 ,lty=4,col="grey",lwd=0.8) +
#geom_vline(xintercept=c(-logFC_t,logFC_t),lty=4,col="black",lwd=0.8) +
geom_hline(yintercept = 0 ,lty=4,col="black",lwd=0.8) +
#coord_flip()+
theme_bw() +
ggpubr::stat_cor(method = "pearson")+
theme( text = element_text(size=18))
可以看到两分组差异分析的上下调基因跟PCA分析的主成分基因相关性还挺好的 :
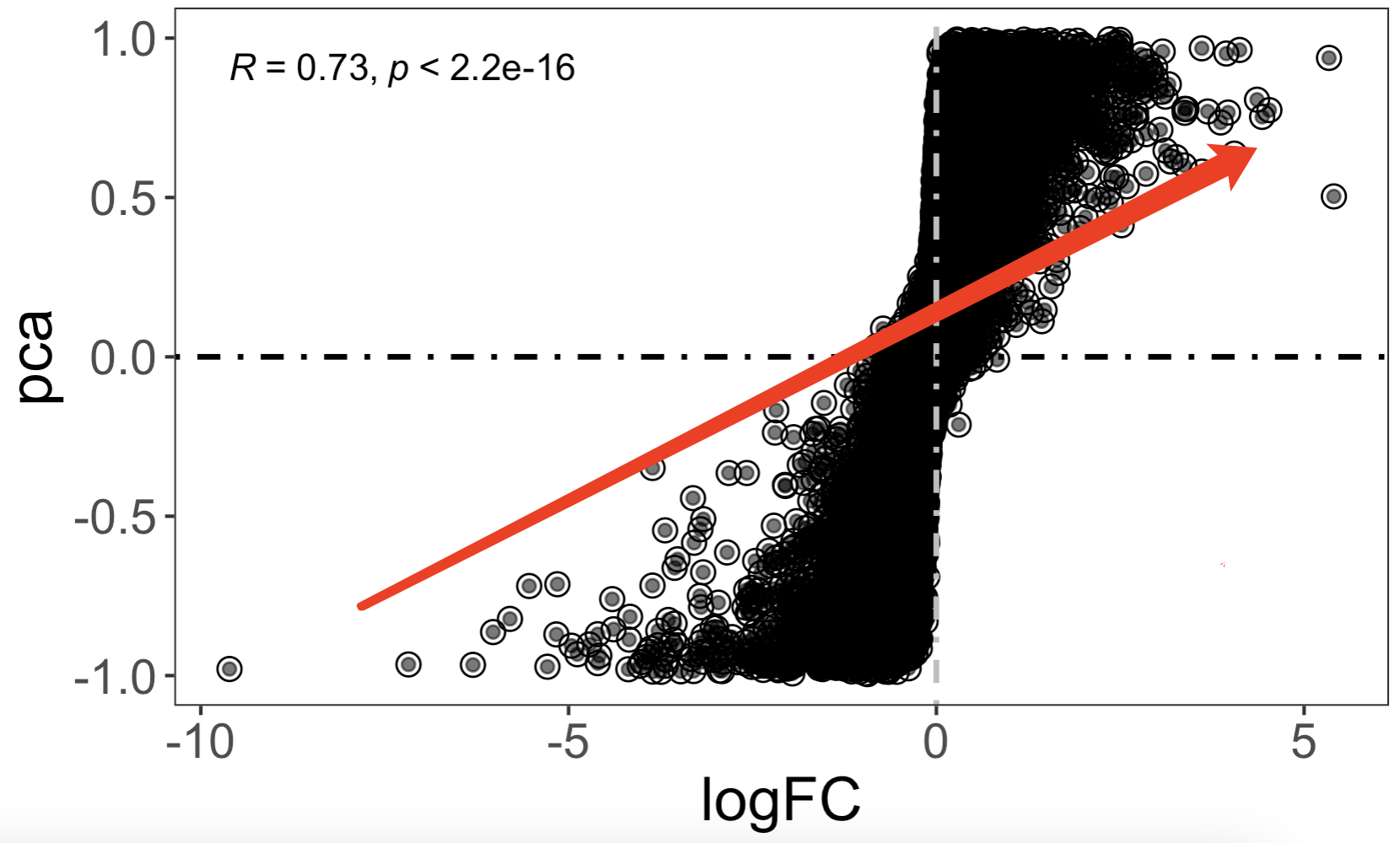
这背后的 统计学原理?
看到了一本有意思的书籍:《现代生物学所需要的现代统计学》,名字是我自己翻译的。
主要是因为太多小伙伴在咱们《生信技能树》后台咨询过想不错生物学知识和统计学知识,恰好这个《Modern Statistics for Modern Biology》把二者涵盖了,在线阅读链接:https://www.huber.embl.de/msmb/index.html
全书还配套代码哦:
source("https://www.huber.embl.de/msmb/install_packages.R")
Data
Code