今年的暑期实习生开启了他的单细胞学习笔记系列,就是《暑假一起学单细胞》,其中第二讲是 一文搞定基本cellranger定量,就有小伙伴留言说:用最新版本的cellranger出的单细胞数量比老版本的多很多,该怎么办?
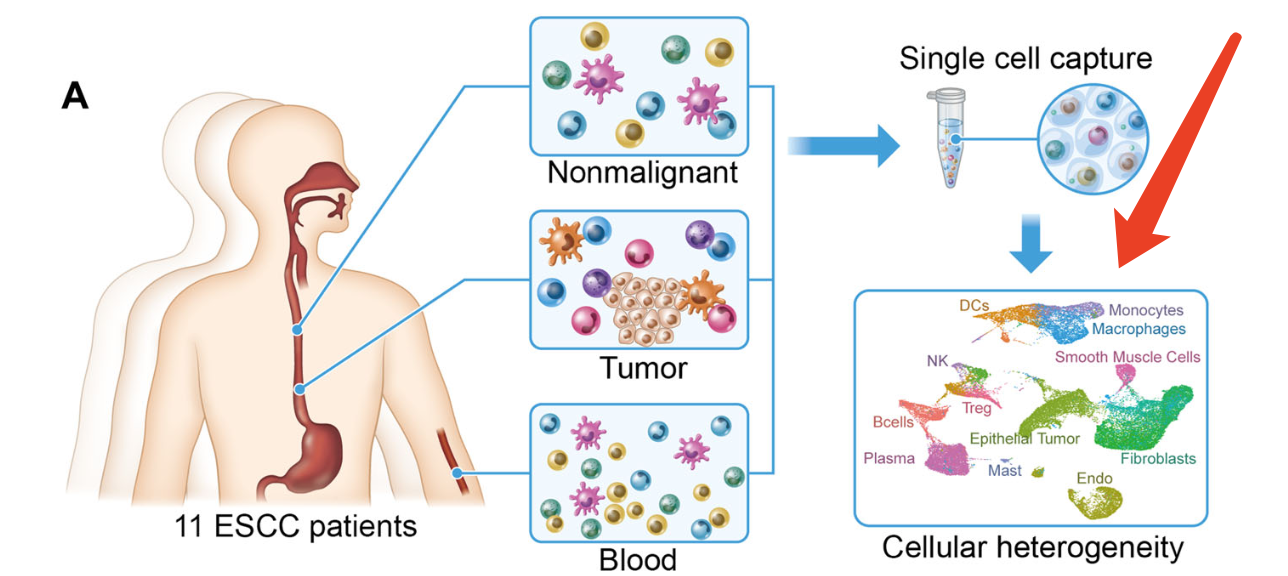
这个问题,在我们的各种微信交流群确实是看到过,恰好有一个小伙伴也在数据集(PRJNA777911)进行了测试,这个是汕头大学那边课题组的2021的食管癌单细胞成果,是发表在NC的文章:《Integrated single-cell transcriptome analysis reveals heterogeneity of esophageal squamous cell carcinoma microenvironment》
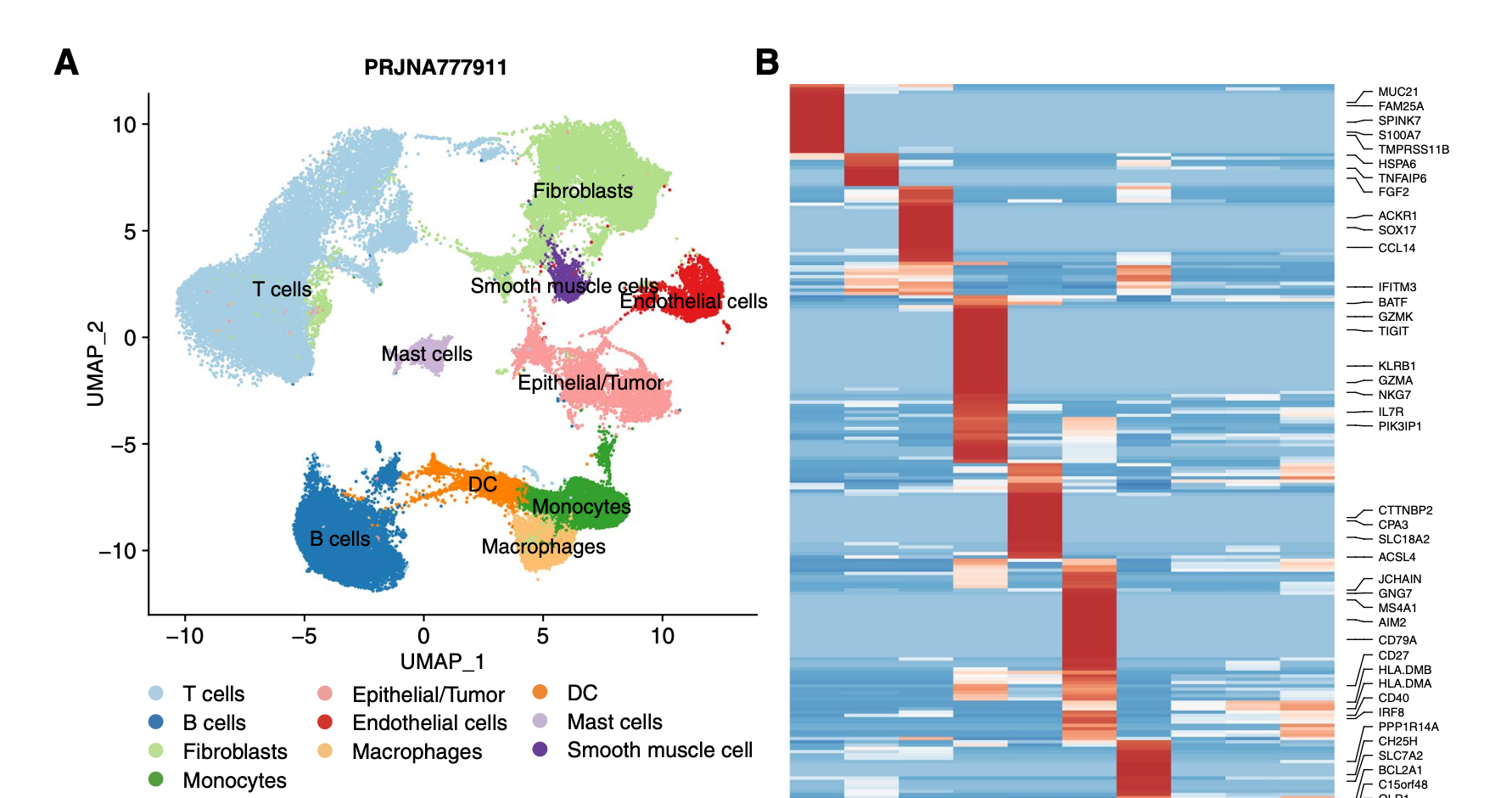
当时看过了之后并没有留下很深刻的印象,因为它数据集是(PRJNA777911)也就是说并没有给出来表达量矩阵,需要自己去下载fq文件走cellranger软件对10x技术单细胞定量流程,很耗费时间,当时仅仅是觉得 它的降维聚类分群似乎是不理想,如下所示:
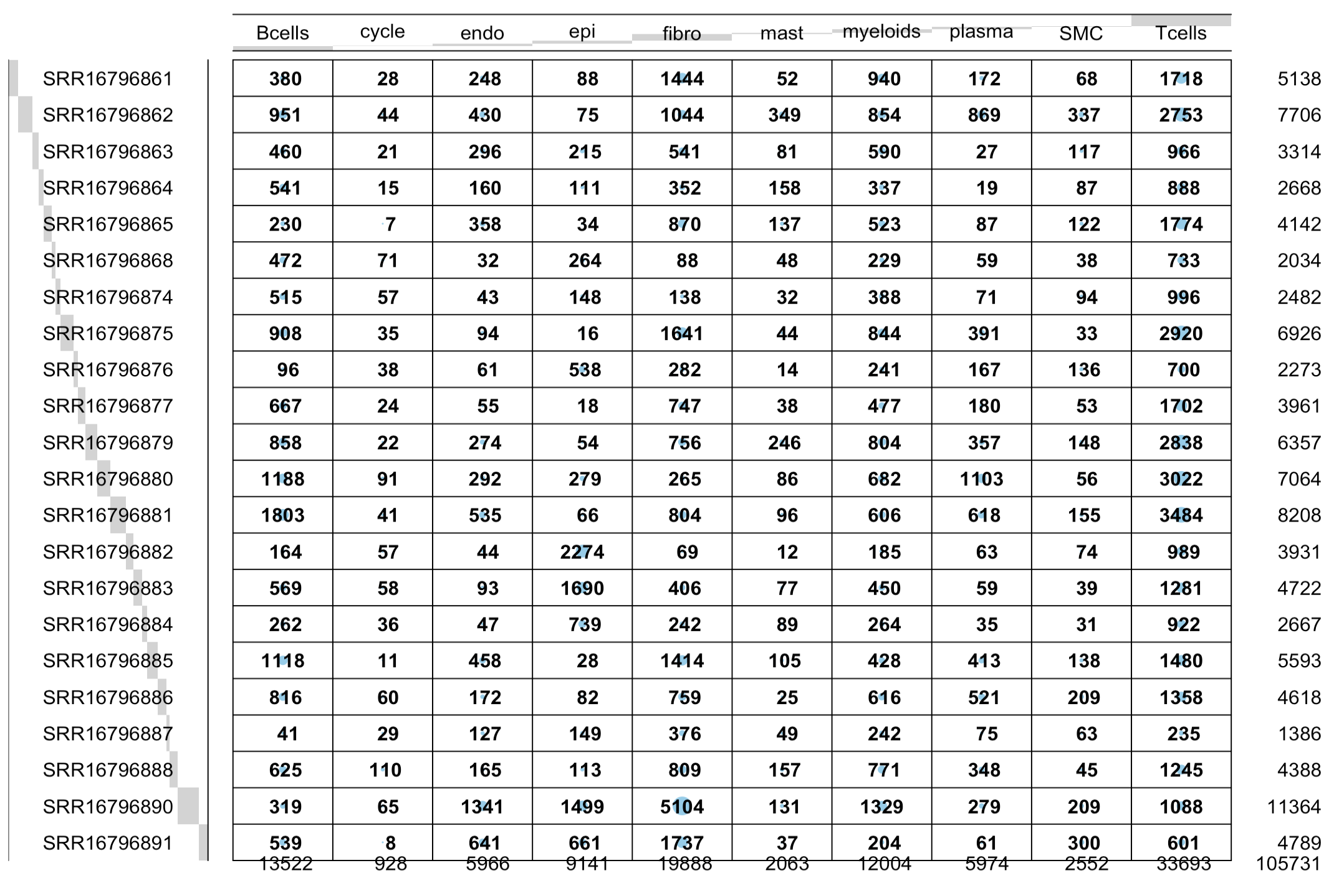
如果仔细的看文章描述: 41,237 cells from all 22 nonmalignant and tumor samples, 就会比较奇怪,因为一般来说,10x技术单细胞定量建议是5~8K的细胞数量,这个22个样品起码得十多万细胞数量了,文章确实有点少。
然后看到了一个基于这个数据集(PRJNA777911)的2023发表的数据挖掘文章:《Integrative analysis of bulk and single-cell gene expression profiles to identify tumor-associated macrophage-derived CCL18 as a therapeutic target of esophageal squamous cell carcinoma》,也是提到了同样的细胞数量:
Single cell RNA-seq data of ESCC with 41,237 cells from PRJNA777911 [29] were selected for the analysis of cell–cell communication and to derive the deconvolution marker gene reference data source. Specifically, 19,882 ESCC primary tumor and 21,355 matched adjacent nonmalignant esophageal cells from 11 treatment naive ESCC patients (10×Genomics Chromium, 3’ assay)
虽然说这次看起来降维聚类分群效果好一点:
并没有什么逻辑层面的硬伤,但是微信交流群的粉丝耗费时间精力去下载了fq文件后走了这个cellranger软件对10x技术单细胞定量,然后发现居然是细胞数量真的是10万以上!!!如下所示:
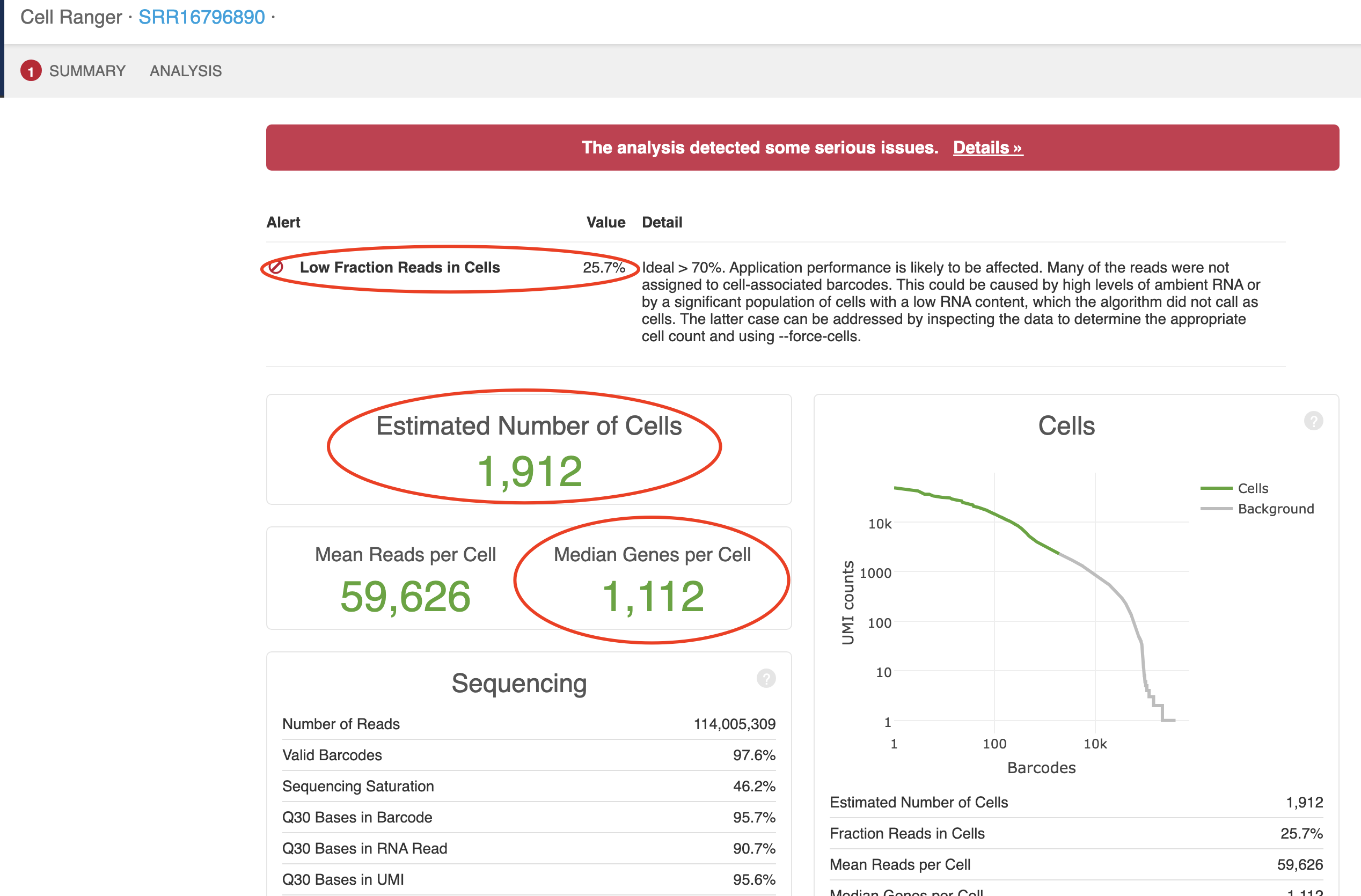
这样就很尴尬,细胞数量如此差异,很让初学者怀疑是不是什么地方有问题,交流了代码就发现确实是不同版本的cellranger软件对10x技术单细胞定量结果可以相差两倍以上,让他重新使用v2的版本定量了看看,这个· SRR16796890 ·样品就是最为特殊的, 居然最后得到的单细胞数量相差五倍以上。。。。
原因就是最新版是V7,其中使用cellranger count命令进行定量,其中的一个参数—include-introns默认是True,这样的话之前的版本cellranger大量的测序的reads本来是落在了基因的内含子区域是不会被计入表达量的,但是最新版改变了策略,落在基因的内含子区域的测序的reads计入基因表达量,所以之前很多不合格的细胞这次合格了。其它cellranger的定量流程详解:
正常走cellranger的定量流程即可,代码我已经是多次分享了。参考: