前面我们提议了 让我们来一起背诵5000个基因吧 ,从大家的留言互动可以看到这个主意确实是“毁誉参半”。虽然说不能完全抹杀掉通过背诵基因来串联起生物学背景知识点的好处,但确实是过于枯燥乏味且效率低下。
比如单细胞亚群每个都有各自的高表达量特异性的基因列表,我们实际上是会打包在代码里面给大家:
last_markers = c('PTPRC', 'CD3D', 'CD3E', 'CD4','CD8A',
'CD19', 'CD79A', 'MS4A1' ,
'IGHG1', 'MZB1', 'SDC1',
'CD68', 'CD163', 'CD14',
'TPSAB1' , 'TPSB2', # mast cells,
'RCVRN','FPR1' , 'ITGAM' ,
'C1QA', 'C1QB', # mac
'S100A9', 'S100A8', 'MMP19',# monocyte
'FCGR3A',
'LAMP3', 'IDO1','IDO2',## DC3
'CD1E','CD1C', # DC2
'KLRB1','NCR1', # NK
'FGF7','MME', 'ACTA2', ## human fibo
'GJB2', 'RGS5',
'DCN', 'LUM', 'GSN' , ## mouse PDAC fibo
'MKI67' , 'TOP2A',
'PECAM1', 'VWF', ## endo
"PLVAP",'PROX1','ACKR1','CA4','HEY1',
'EPCAM' , 'KRT19','KRT7', # epi
'FYXD2', 'TM4SF4', 'ANXA4',# cholangiocytes
'APOC3', 'FABP1', 'APOA1', # hepatocytes
'Serpina1c',
'PROM1', 'ALDH1A1' )
这样的话大家很容易去人工给单细胞亚群命名,只需要把基因对应到各自的亚群即可 :
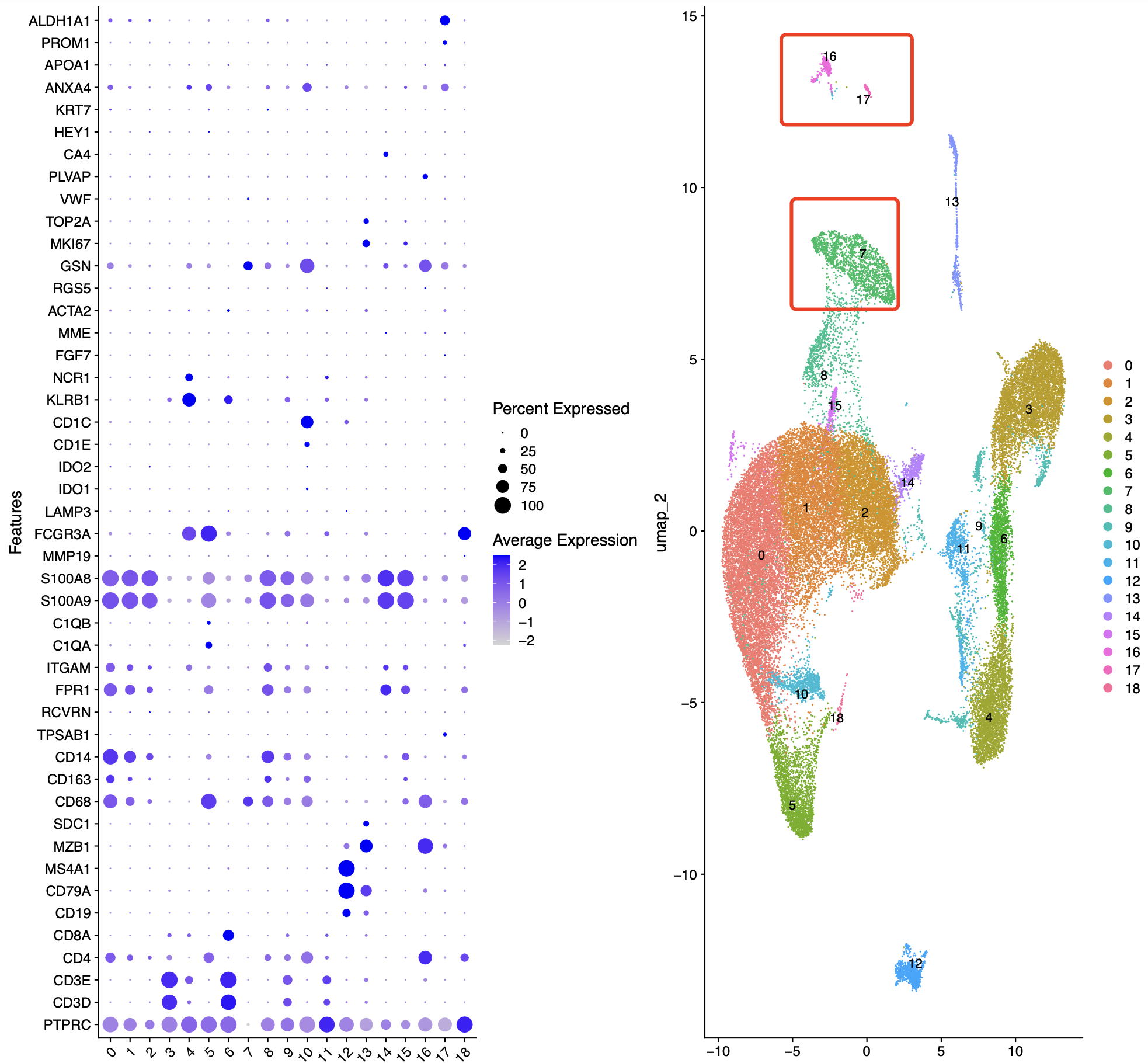
但是大家可以看到,其实是并不是所有的亚群都是可以对应的,所以我们一直在给这个单细胞转录组数据分析标准代码打补丁,其中上皮细胞我们介绍了不同器官:
免疫细胞呢,主要是包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群,但是目前我们才介绍到:
stromal 里面的fibo 和endo,我们就介绍了内皮细胞细分亚群,其实还有周细胞和平滑肌细胞,我们后期再慢慢补充。而且这些是无穷无尽的,比如上面的如果我补充了髓系细胞相关基因就会看到树突相关亚群啦 :
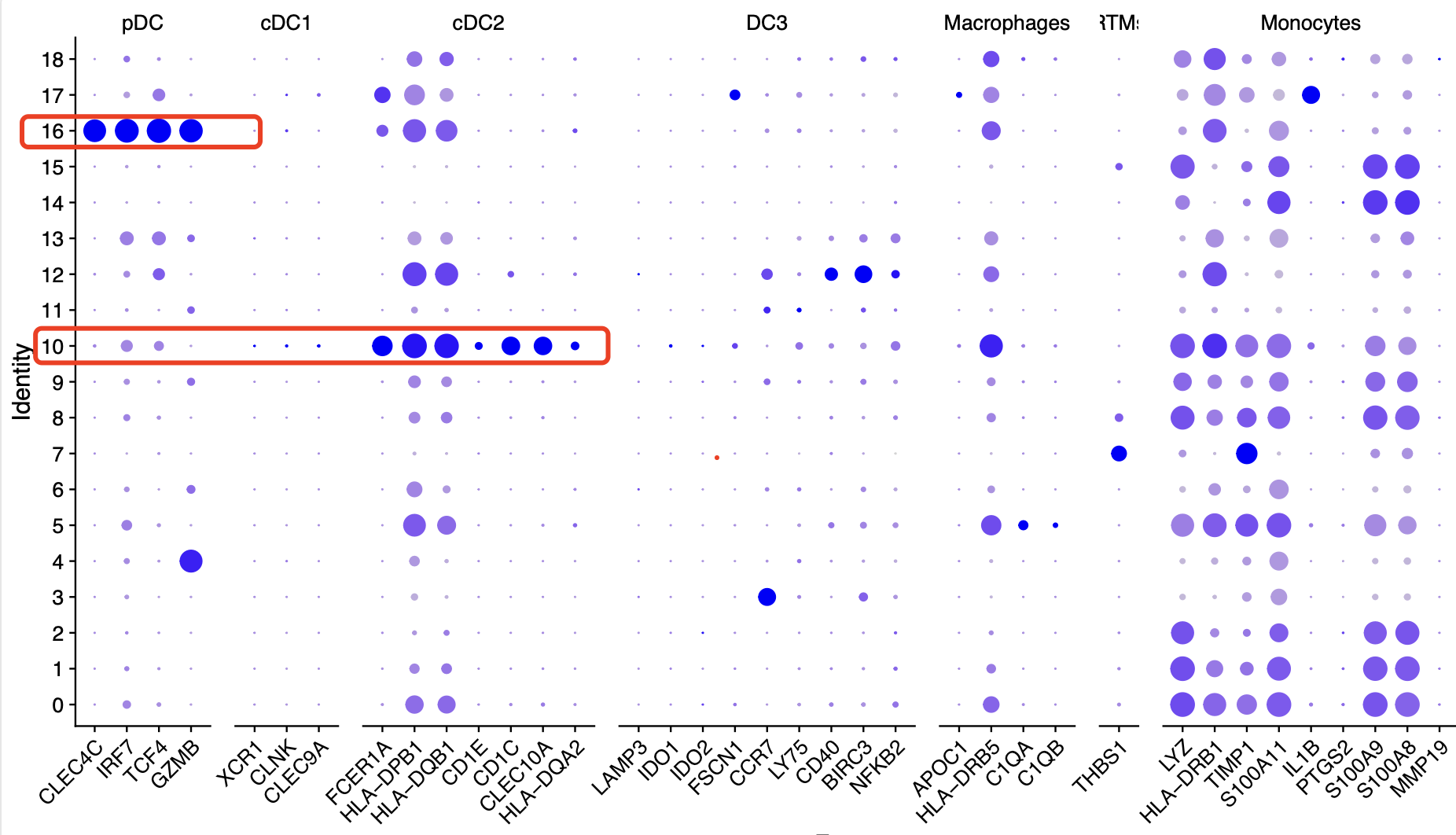
但是上面的7那个亚群仍然是孤零零的,没办法人工注释,起码在我的生物学背景里面是缺失的,我们先看看它的高表达量基因,如下所示:
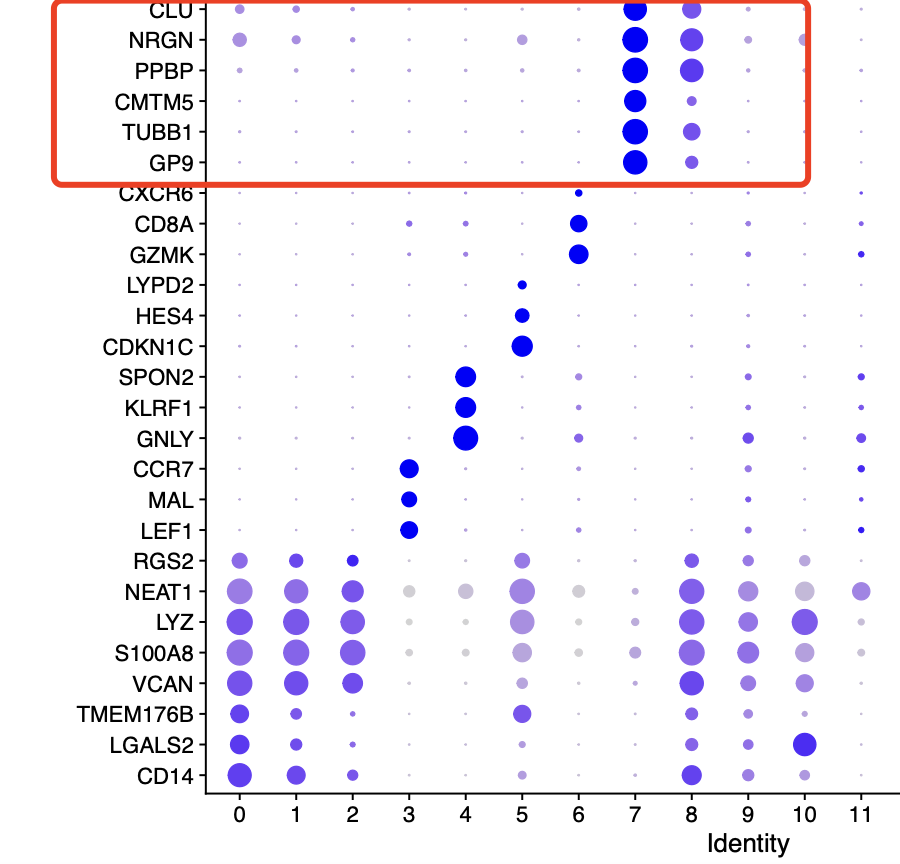
可以看到亚群7旁边的那个8其实是可能的双细胞,因为它兼具了亚群7和单核细胞的两个 特性。而亚群7的那些基因我不认识,第一个办法就是去一个个搜索认识,如果是我们已经是成功的背诵了五千多个基因其实是就可以省略搜索这个步骤啦!另外一个办法是使用哈医大的一个在线工具:
- 链接:https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-023-01249-5
- 文章标题:《》
- 网页工具链接:
很简单的就给出来了如下所示的亚群名字:
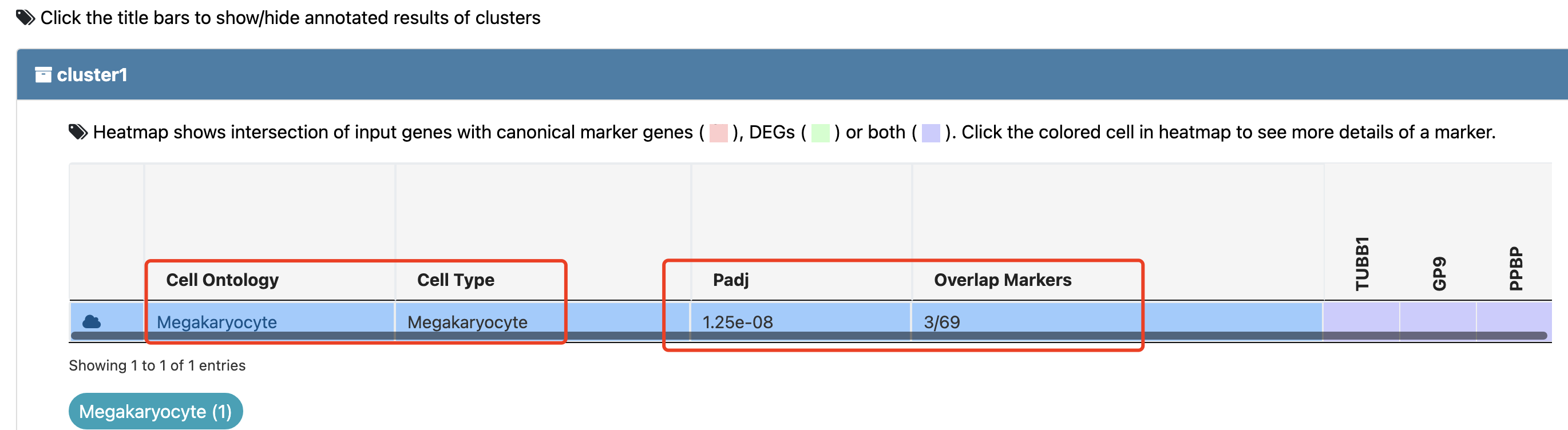
只需要在网页工具的主页输入我们的亚群7 的基因列表而已,按照网页工具要求的格式哦,如下所示:cluster1:TUBB1 ,GP9 ,PPBP
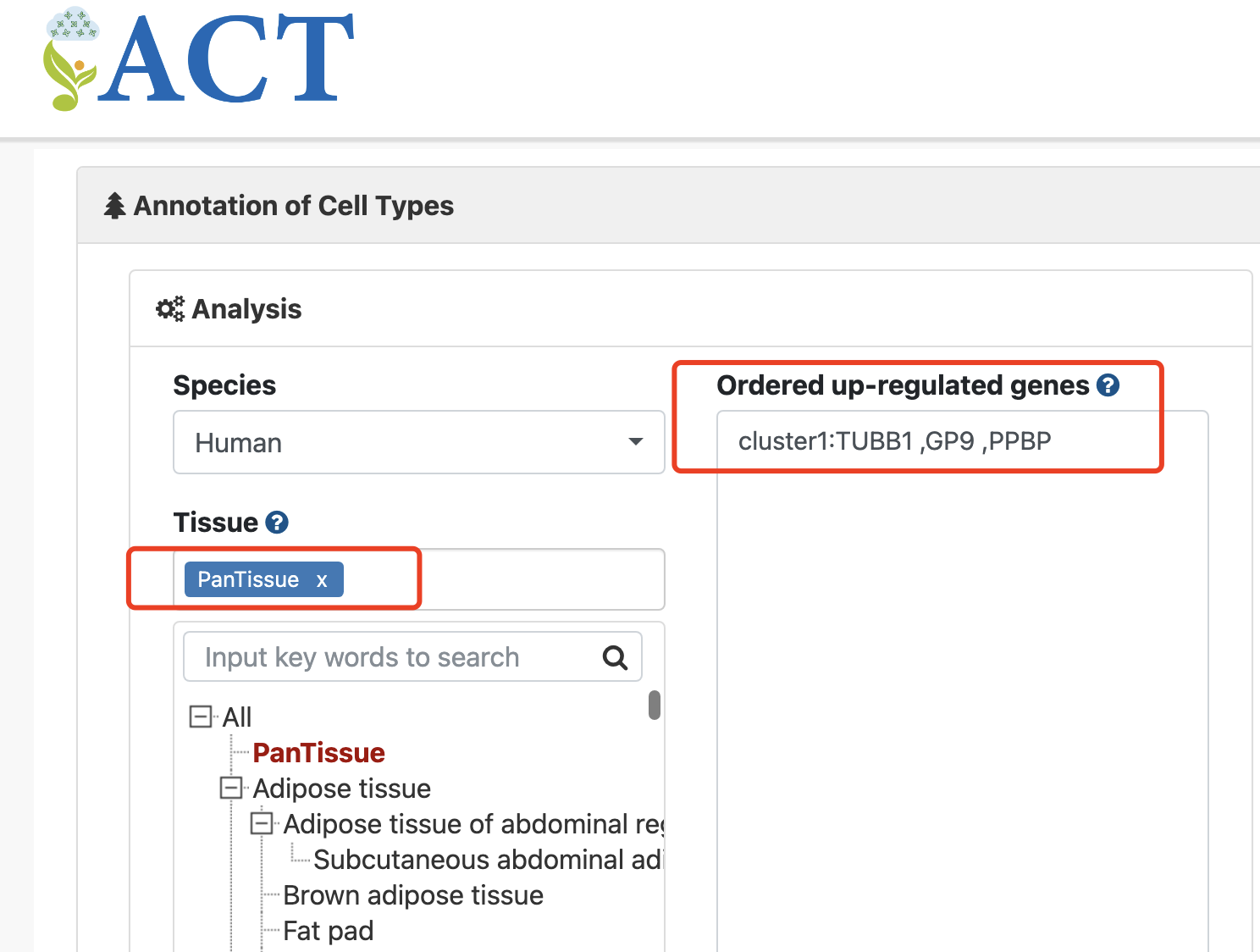
当然了,我们可以很轻松的针对全部的亚群都输出top的高表达量的基因列表,并且符合网页工具的输入格式,即可一次性全部的自动化注释单细胞亚群的生物学名字啦!
load(file = 'check-by-0.5/qc-_marker_cosg.Rdata')
head(marker_cosg)
## Top10 genes
library(dplyr)
cat(paste0('cluster',0:18,':',
unlist(apply(marker_cosg$names,2,function(x){
paste(head(x),collapse=',')
})),'\n'))
得到如下所示的各个亚群以及其配套的top6的基因列表:
cluster0:CD14,LGALS2,TMEM176B,CPVL,BLVRB,TMEM176A
cluster1:VCAN,S100A8,LYZ,MNDA,S100A12,S100A9
cluster2:S100A8,NEAT1,RGS2,CSF3R,VCAN,HBB
cluster3:LEF1,MAL,CCR7,TCF7,IL7R,LTB
cluster4:GNLY,KLRF1,SPON2,FGFBP2,KLRD1,HOPX
cluster5:CDKN1C,HES4,LYPD2,C1QA,CKB,TCF7L2
cluster6:GZMK,CD8A,CXCR6,CD8B,LAG3,KLRG1
cluster7:GP9,TUBB1,CMTM5,C6orf25,SDPR,GNG11
cluster8:PPBP,NRGN,CLU,PF4,PRKAR2B,F13A1
cluster9:IFITM1,IL32,NKG7,PTPRCAP,CALM1,GIMAP7
cluster10:FCER1A,ENHO,CD1C,CLEC10A,PKIB,CLIC2
cluster11:ANK3,PARP15,SYNE2,ITK,RORA,SLFN12L
cluster12:MS4A1,LINC00926,CD79A,VPREB3,FCER2,RP11-693J15.5
cluster13:TNFRSF17,TXNDC5,IGLL5,IGF1,POU2AF1,IGJ
cluster14:MMP8,MMP9,LTF,LCN2,PGLYRP1,ANXA3
cluster15:RETN,SLC39A8,CENPF,MKI67,IL1R2,C19orf59
cluster16:LILRA4,CLEC4C,LRRC26,SERPINF1,SCT,PTPRS
cluster17:CD34,C19orf77,EMID1,AP001171.1,PRSS57,NPR3
cluster18:LINC00278,KCNQ1OT1,RP11-362F19.1,CCRN4L,HCAR3,SPRED1
这个时候输入这么多信息到网页工具,就会很慢哦!成功了之后会有如下所示的单细胞亚群注释信息:
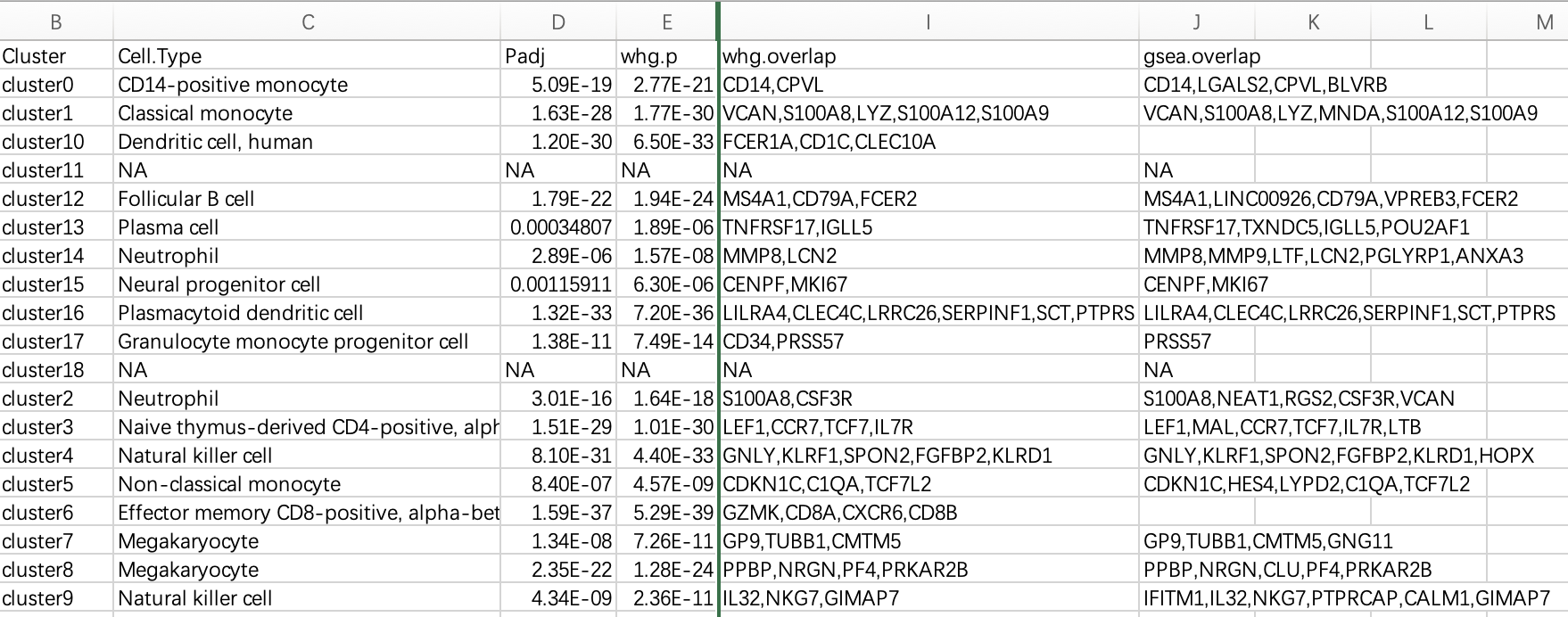
基本上跟我们自己的手动的亚群命名大差不差啦:
celltype[celltype$ClusterID %in% c( 0,1,2,14,15 ),2]='cd14-mono'
celltype[celltype$ClusterID %in% c( 5,18),2]='cd16-mono'
celltype[celltype$ClusterID %in% c( 10 ),2]='cDC'
celltype[celltype$ClusterID %in% c( 16 ),2]='pDC'
celltype[celltype$ClusterID %in% c( 3 ),2]='CD4'
celltype[celltype$ClusterID %in% c( 4 ),2]='NK'
celltype[celltype$ClusterID %in% c( 6,9,11 ),2]='CD8'
celltype[celltype$ClusterID %in% c( 12 ),2]='Bcells'
celltype[celltype$ClusterID %in% c( 13 ),2]='plasma'
celltype[celltype$ClusterID %in% c( 7,8 ),2]='megakaryocyte'