看到了朋友圈好友转发了一个2024新鲜出炉的基于蛋白质组数据对阿兹海默症进行早期诊断的文章:《Alzheimer’s disease early diagnostic and staging biomarkers revealed by large-scale cerebrospinal fluid and serum proteomic profiling》,文章的摘要写的非常棒,很清晰,如下所示:
- 深入分析配对脑嵴液和血清蛋白质组的蛋白质组学工作流程。
- 独立的多中心集合,结合多种验证方法。
- 开发19个脑脊液和8个血清蛋白质组用于早期阿兹海默病(AD)诊断。
- 21个脑脊液和18个血清蛋白在不同的 AD 阶段失调。‧
- 为临床筛查和分期进行 AD 血液测试奠定基础。
让我们认真读一下这个文章吧,因为是《The Innovation》期刊,目前看起来是开源的,pdf很容易下载。总体上来说实验设计还算是比较容易理解的:To explore early diagnostic and staging biomarkers of AD, we performed tandem mass tag (TMT)–based proteomic analysis of paired CSF and serum samples
也就是说,队列的样品是: 成对的脑脊液和血清样本,包括 AD 患者、 AD 引起的 MCI 患者、 HD 患者、 ALS 患者和 CN组 。 - cognitive normal (CN) group
- mild cognitive impairment (MCI) due to AD group
- amyotrophic lateral sclerosis (ALS)/Huntington disease (HD)
- Alzheimer disease (AD)
首先是多分组的蛋白质组差异分析
重点是在脑脊液数据集里面, (MCI) due to AD group与 CN组 相比,185种 CSF 蛋白表达水平上调(p < 0.05,log2倍数变化[ FC ] > 0.25) ,5种 CSF 蛋白表达水平下调(p < 0.05,log2 FC < -0.25)
以及在血清蛋白质组数据集里面, (MCI) due to AD group与 CN组 相比,鉴定了49个失调蛋白(p < 0.05,| log2 FC | > 0.25),不知道为什么这个时候不区分上下调啦。
因为还涉及到HD,ALS等疾病,其它差异分析,总体上来说可以看这个韦恩图啦:
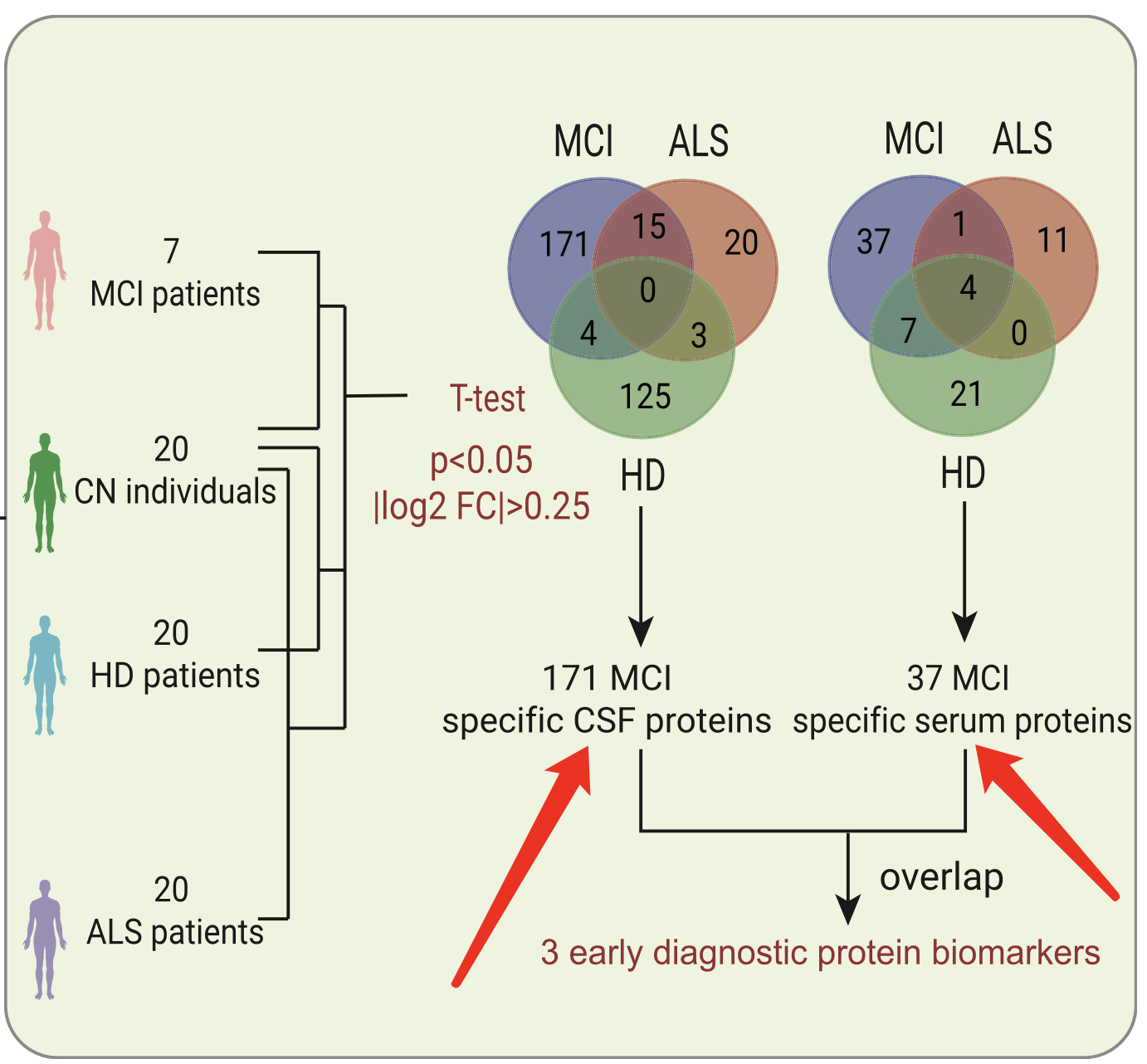
其实到这里,仅仅是一个简单的多分组的差异分析而已,无论是表达量芯片还是转录组测序,还是蛋白质组代谢组,亦或是单细胞,都是没什么科研含量的,很常规啦。
然后是关上面的韦恩图该如何聚焦后续的分析,这个研究者的操作是:在排除与 HD 组和 ALS 组重叠的失调血清蛋白后,选择了37个由 AD 引起的 MCI 的潜在特异性血清生物标志物进行进一步验证。扩大队列验证差异分析结果
有意思的地方来了,基于上面的多分组差异分析结果,这个时候其实有两个诊断模型需要构建,首先是可以区分由 AD 引起的 MCI 患者和其他神经退行性疾病患者,其次是准确区分 (MCI) due to AD group与 CN组 。
而且前面已经是有差异分析结果,所以可以使用聚焦后的蛋白质组,这样会省钱,而且可以扩大队列:

然后我们来看看研究者的实验设计吧: - 在一个独立的多中心队列中进行了基于并行反应监测(PRM)的靶向蛋白质组学实验。验证队列包括221名脑脊液样本参与者和288血清样本参与者
- 总共有57个和12个失调的脑脊液和血清蛋白在 MCI 由于 AD 组分别得到验证
也就是说,超过三分之二的差异分析结果因为扩大队列而没办法验证???机器学习模型
前面有了被验证的差异分析结果,接下来就进行如下所示机器学习:
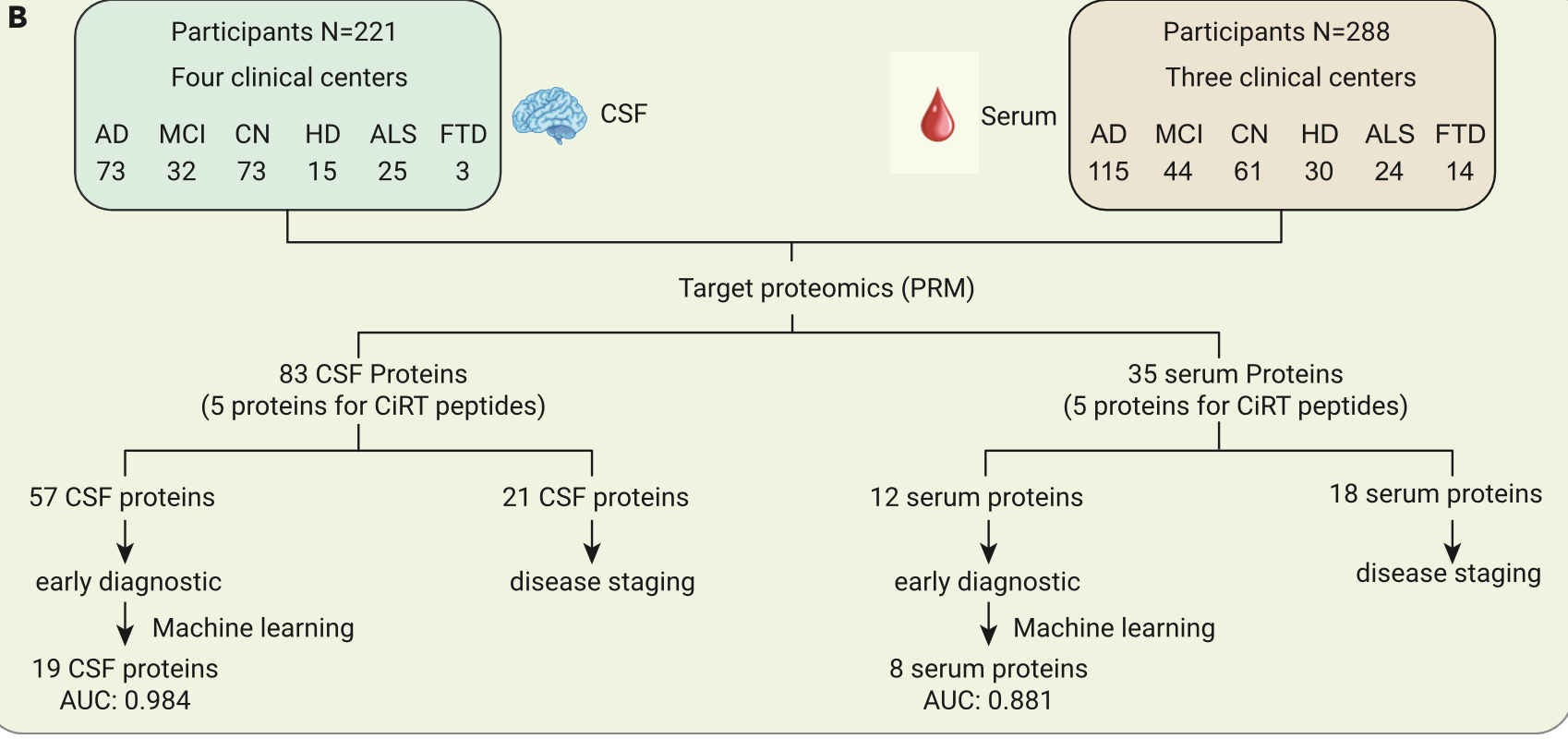
两个随机森林机器学习模型: - 23名 AD 患者和45名 CN 的血清蛋白质组数据
- 8 core proteins was selected to distinguish MCI due to AD from CN
- 0.881的 AUC ( A cohort of 8 MCI due to AD and 21 CN participants )
- 21例 AD 患者和52例 CN 的脑脊液蛋白质组数据
- serum GFAP achieved an AUC of 0.714, whereas NEFL achieved an AUC of 0.511
这些结果都是在附图里面,没有出现在正文,呵呵哈哈哈!!!
后面还有其它分析,因为阿兹海默症患者有轻重缓急,所以实验设计分组是:MCI due to AD mild stage (ADM), AD moderate stage (ADMO), and AD severe stage (ADS). 可以做时间序列分析,找到那些随着疾病进展而起关键作用的分子,如下所示:
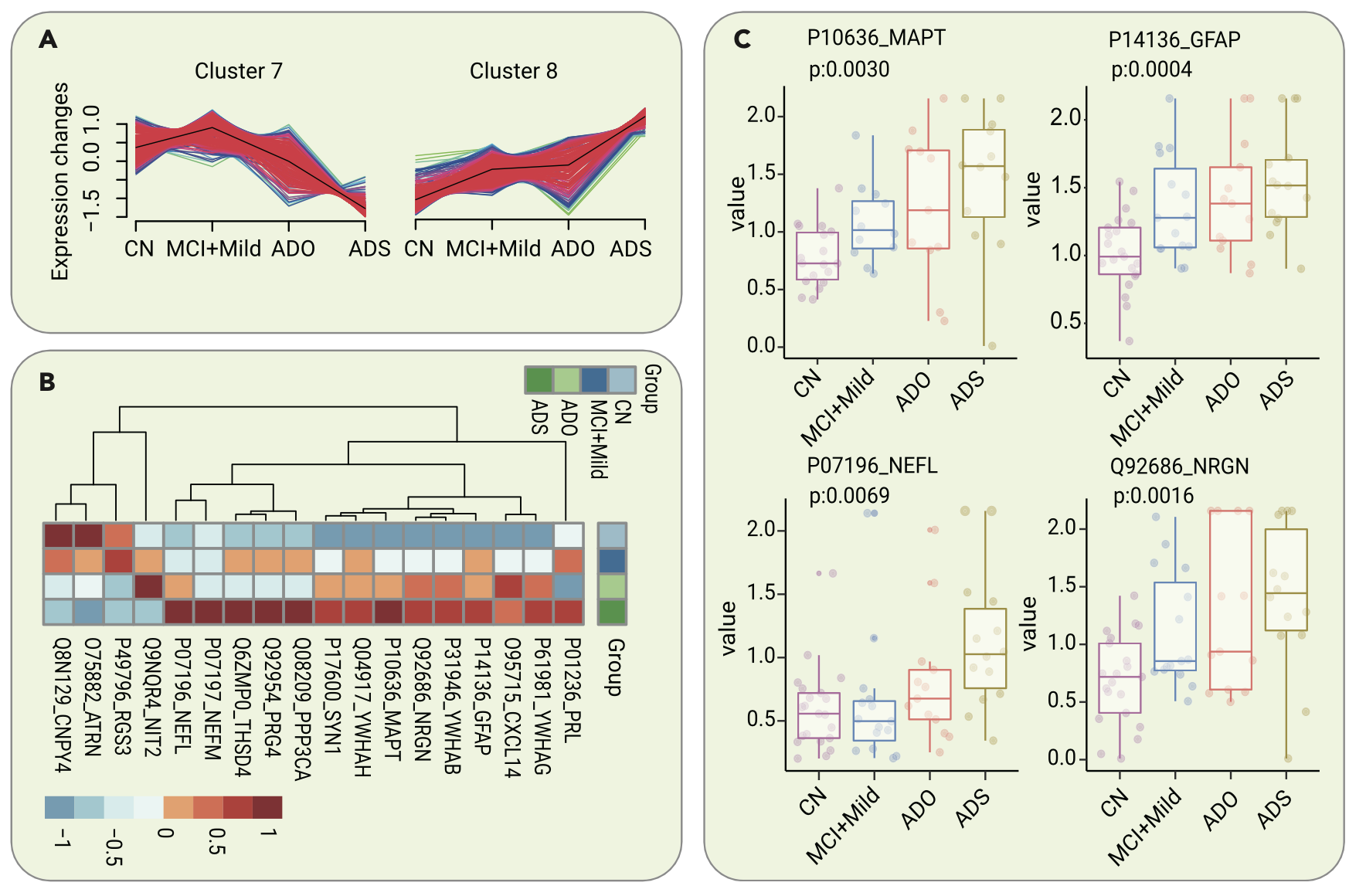
都是2024了,让我们看看这个文章创新之处在哪里吧? - 临床病人资源吗?
- 发现队列一百人以下,验证队列1000人以下,临床队列万人附近?
- 组学吗?
- 表达量芯片过时,转录组太普通,蛋白质组代谢组不稳定,单细胞还是有点贵
- 数据分析吗?
- 简单的差异分析,富集分析,取交集,时间序列趋势分析,wgcna等等
- 机器学习吗?
- 对我们屌爆侠来说,问题不大吧,反正就是某个包的某个函数的使用。
- 临床价值吗?
- 虽然在文章摘要或者总结的时候,我们总会写“为癌症的治疗提供了新的方向”、“提出新的治疗靶点”、“有望成为精准靶向治疗的潜在策略”、“前景远大”、“意义深远”……看上去好像真的做出点有意义的东西一样
我们来做一个模拟科研吧
假设我们想说明一种基于服装判断人群职业的方法:
- 虽然在文章摘要或者总结的时候,我们总会写“为癌症的治疗提供了新的方向”、“提出新的治疗靶点”、“有望成为精准靶向治疗的潜在策略”、“前景远大”、“意义深远”……看上去好像真的做出点有意义的东西一样
- 在中关村的一个红绿灯路口观察到了一个有趣的现象,即朝左走的人群大多是西装革履的人,而朝右走的人群则主要是清洁工大妈和美团外卖员。作者认为他们发现的这个规律可以构建一个高达100%准确性的诊断模型。
- 然而,当作者扩大队列并在其他红绿灯路口进行验证时,发现效果并不如他们最初观察到的那么好,成功率只有60%左右。尽管如此,为了发表文章,他们似乎选择继续推进研究。
- 最后,作者使用了一个”万能算法” (机器学习,炼丹师),在最初发现规律的路口继续寻找更严格的标准来区分人群职业,并发现服装问题仍然是最具区分度的标准。