看到了之前的实习生离开后还在持之以恒学习做生物信息学基础知识整理,非常欣慰。而且是春节期间也在不间断更新,这一点就超过了我, 起码我春节前后两周都是在各种转载“混日子”了 。比如这2024-02-12 更新了:芯片代码实操(1)提前安装所需R包,里面提到的批量管理各种r包的小技巧是我十年前整理的。
当时以为是很厉害了的经验,也大力宣传了,所以绝大部分小伙伴都是直接就使用了我的经验。使用传统的install.packages或者BiocManager::install来安装一系列r包,如下所示:
cran.packages <- c('knitr', 'mixOmics', 'xtable', 'ggplot2', 'vegan', 'cluster',
'gridExtra', 'pheatmap', 'ruv', 'lmerTest', 'bapred')
install.packages(cran.packages)
bioconductor.packages <- c('sva', 'limma', 'AgiMicroRna',
'variancePartition', 'pvca')
if (!requireNamespace('BiocManager', quietly = TRUE))
install.packages('BiocManager')
BiocManager::install(bioconductor.packages, version = '3.8')
亦或者是我们想批量加载一系列包,如下所示:
library(Seurat)
library(ggplot2)
library(clustree)
library(cowplot)
library(dplyr)
library(stringr)
library(ggsci)
library(patchwork)
library(ggsci)
library(ggpubr)
library(RColorBrewer)
library(monocle)
library(dplyr)
library(Seurat)
library(tidyverse)
library(patchwork)
# pak::pkg_install('aertslab/SCENIC')
library(SCENIC)
library(harmony)
上面的代码当然是可以使用的,实际上我们有一个更好的解决方案,俗称造好的轮子。就是pacman这个r包啦,https://github.com/trinker/pacman , 需要学习的函数很少,如下所示:
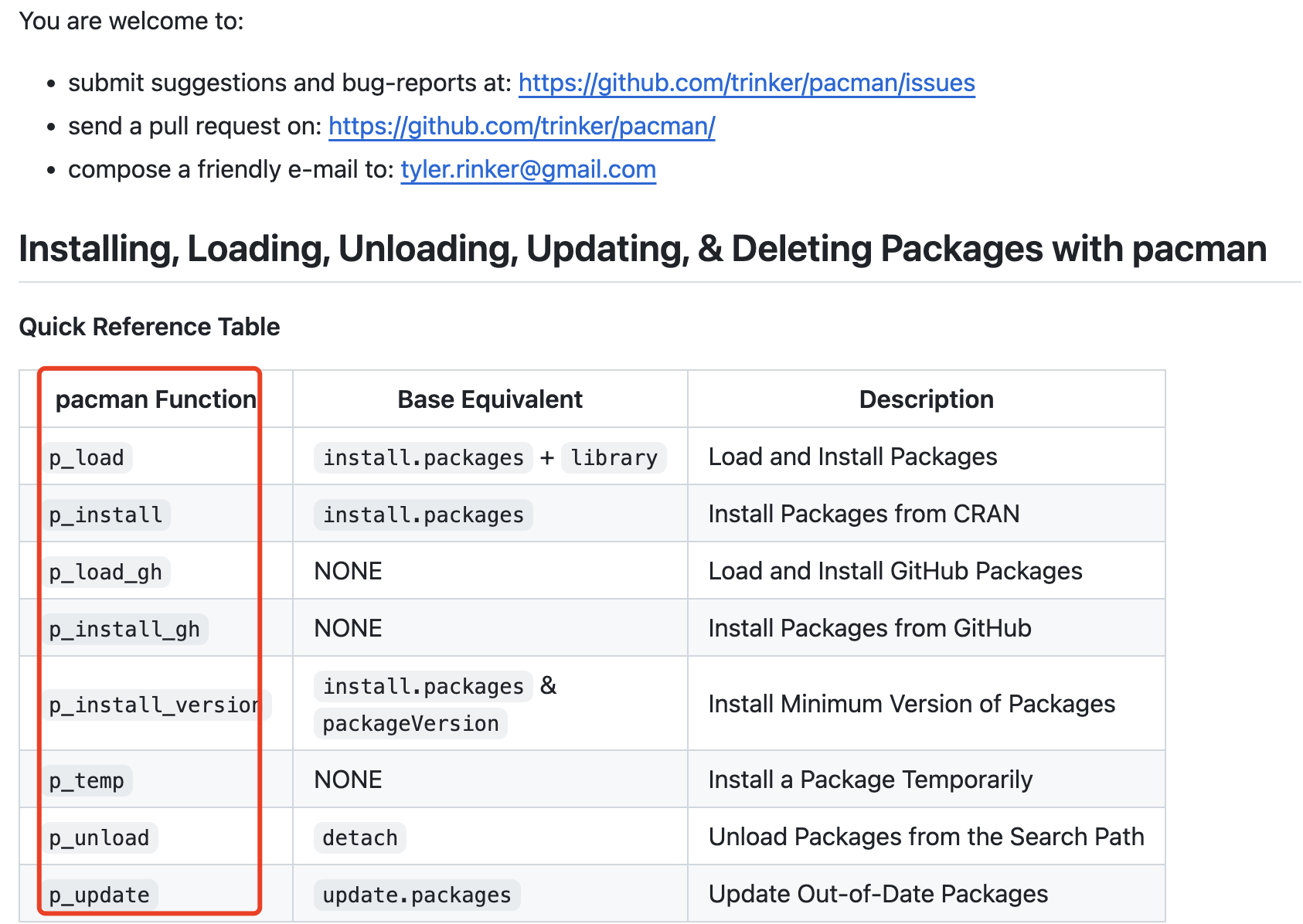
首先是可以批量安装或者加载大量的包
#install.packages('pacman')
library(pacman)
pacman::p_load(char = cran.packages)
pacman::p_load(char = bioconductor.packages)
只需要前面的代码设置好 cran.packages 和 bioconductor.packages 这两个变量即可,它们里面的每个元素就是我们需要的安装或者加载大量包。当然了,如果是从github安装,就需要一点特殊的操作,就是给出来需要安装的r包的github用户名,如下所示:
pak::pkg_install('aertslab/SCENIC')
p_install_gh(c( 'aertslab/SCENIC' ))
p_load_gh( 'aertslab/SCENIC' )
还可以使用p_install_version函数去安装指定的版本的包,以及使用p_unload去载出指定的r包。
查看任意包的内置数据集和函数列表
就是 p_data 函数和 p_functions函数
> pacman::p_data(ggplot2)
Data Description
1 diamonds Prices of over 50,000 round cut diamonds
2 economics US economic time series
3 economics_long US economic time series
4 faithfuld 2d density estimate of Old Faithful data
5 luv_colours 'colors()' in Luv space
6 midwest Midwest demographics
7 mpg Fuel economy data from 1999 to 2008 for 38 popular models of cars
8 msleep An updated and expanded version of the mammals sleep dataset
9 presidential Terms of 12 presidents from Eisenhower to Trump
10 seals Vector field of seal movements
11 txhousing Housing sales in TX
> pacman::p_functions(ggplot2)
[1] ".data" ".pt"
[3] ".stroke" "%+%"
[5] "%+replace%" "aes"
[7] "aes_" "aes_all"
[9] "aes_auto" "aes_q"
其实就相当于你去看包的 官方文档而已:
p_help(ggplot2)
p_help(ggplot2, web = FALSE)
p_help(ggplot2, build.pdf = TRUE)
p_vignette()
p_vign(ggplot2)
如下所示:
-
Vignettes found by
utils::browseVignettes(package = pack) -
Vignettes in package
ggplot2 - Extending ggplot2 - HTML source R code
- Using ggplot2 in packages - HTML source R code
查看任意包引用详情
这个好像是用处有点弱,因为绝大部分情况下大家啊好像是并不会直接去引用某个r包,除非这个r包确实是有对应的文章。
> p_cite(ggplot2)
To cite ggplot2 in publications, please use
H. Wickham. ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New
York, 2016.
A BibTeX entry for LaTeX users is
@Book{,
author = {Hadley Wickham},
title = {ggplot2: Elegant Graphics for Data Analysis},
publisher = {Springer-Verlag New York},
year = {2016},
isbn = {978-3-319-24277-4},
url = {https://ggplot2.tidyverse.org},
}