看到了交流群有人分享了这个发表于2024的3月份在Nature Medicine 杂志的文章,标题是:《Single-cell transcriptomic analyses reveal distinct immune cell contributions to epithelial barrier dysfunction in checkpoint inhibitor colitis》,内容简介如下所示:

交流群的小伙伴表示它这个文章的数据集里面的样品数量和细胞数量的关系很奇怪,数据集是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE206301 ,可以看到样品数量还是很夸张的,而且这个文章是发表在2021的预印本的研究,也就是说早在2020左右就已经完成了152个样品的单细胞转录组,那个时候起码还得三四万一个样品,这就是500万人民币的经费在燃烧了。
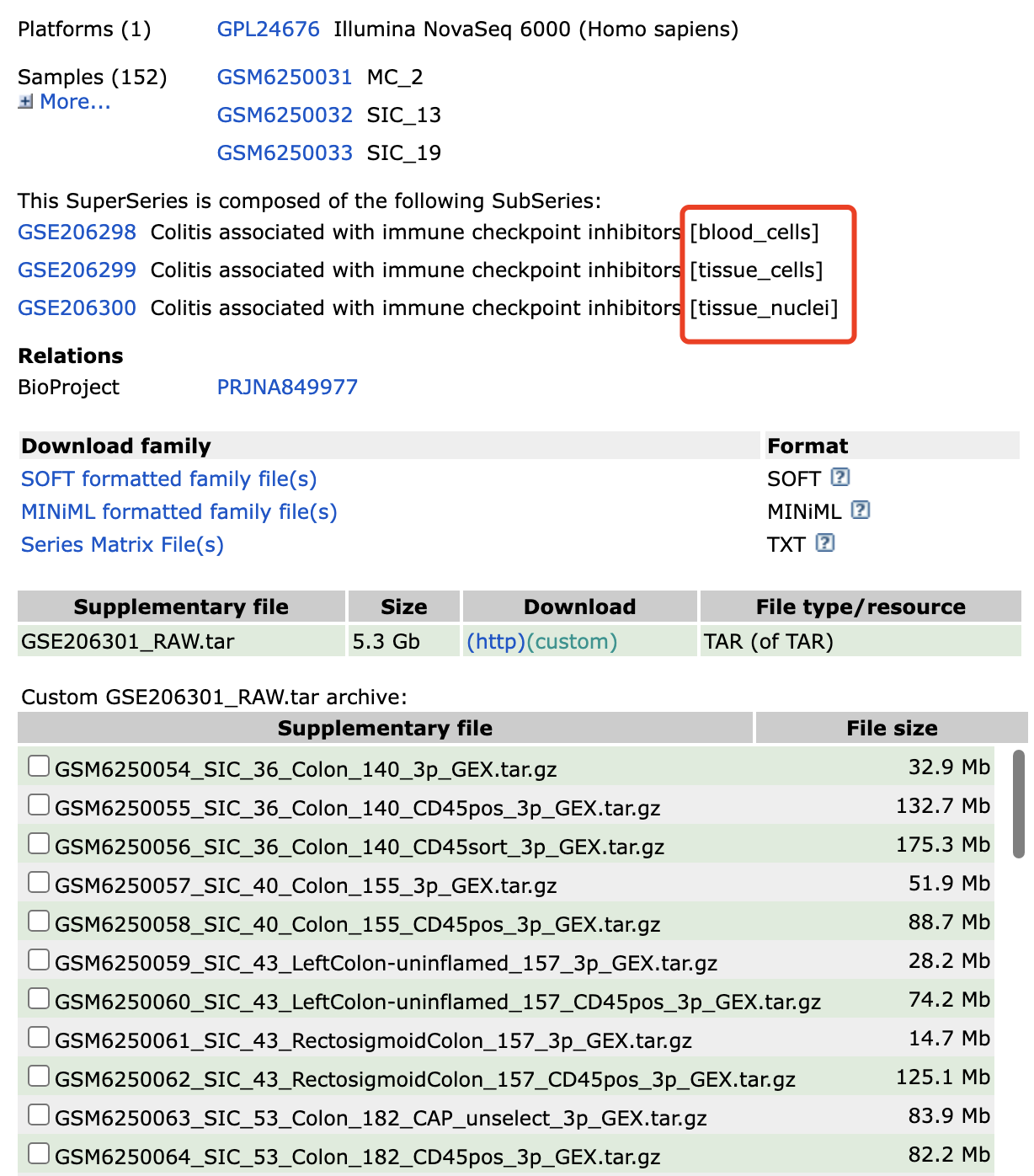
但是,如果我们下载它提供的每个样品的表达量矩阵文件和cellranger报表,就会发现每个样品质量都惨不忍睹!
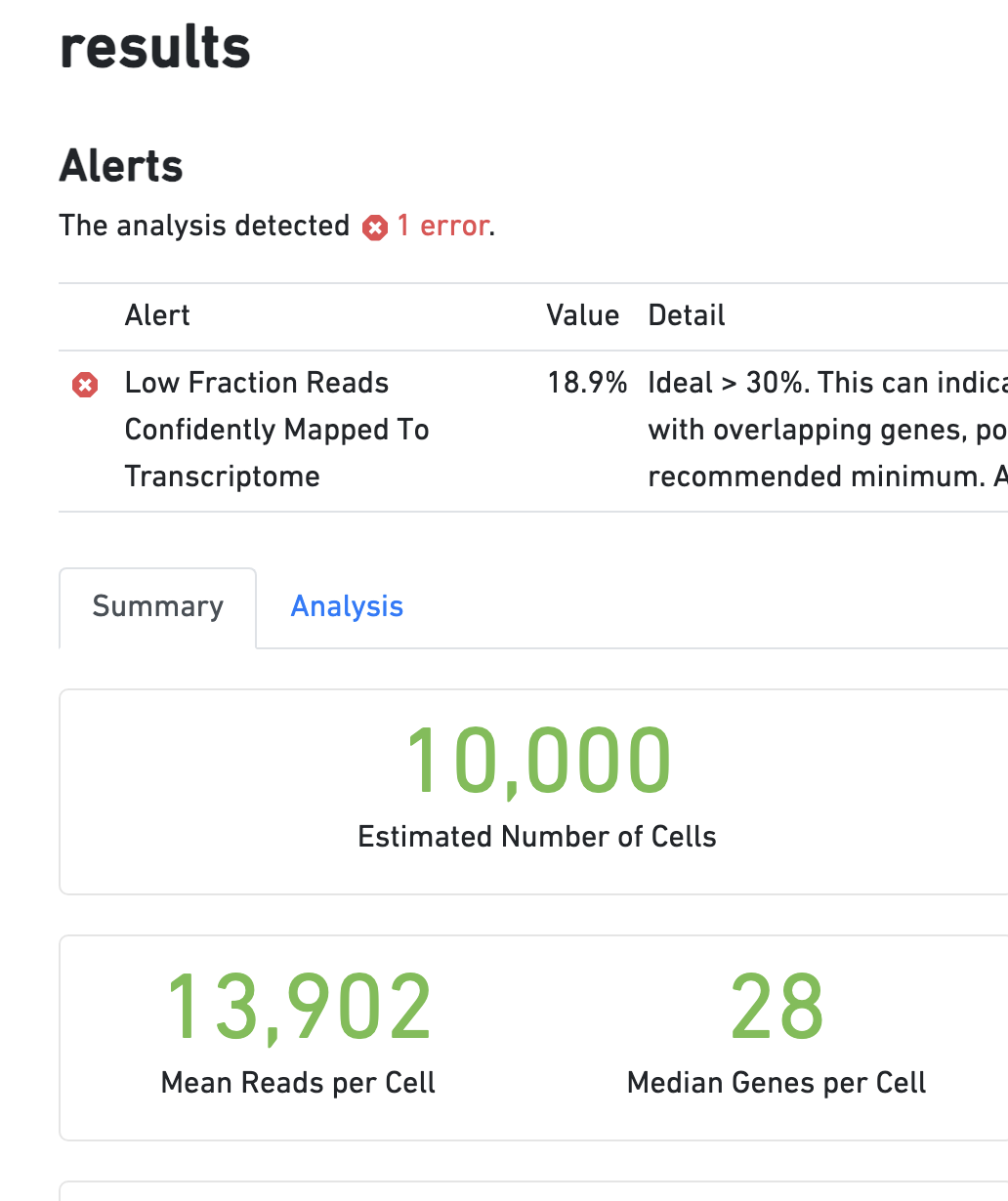
我汇总了这个文章里面的单细胞核转录组的34个10x技术单细胞转录组的报表,如下所示:
10,000 13,902 28 139,020,938
10,000 14,920 30 149,202,916
10,000 14,648 154 146,486,229
10,000 15,325 19 153,253,107
10,000 10,418 20 104,185,854
10,000 11,201 10 112,013,839
10,000 12,212 62 122,125,808
10,000 14,790 87 147,908,590
10,000 16,043 409 160,439,328
10,000 12,464 32 124,640,796
10,000 13,753 52 137,532,673
10,000 10,160 38 101,604,274
10,000 14,190 32 141,907,328
10,000 25,716 50 257,167,328
10,000 9,181 28 91,813,272
10,000 8,759 59 87,594,642
10,000 6,941 24 69,417,191
10,000 8,725 38 87,250,215
10,000 15,606 117 156,060,575
10,000 13,403 346 134,037,076
10,000 15,876 143 158,764,477
10,000 14,423 133 144,234,478
10,000 14,683 26 146,838,589
10,000 14,239 75 142,393,186
10,000 15,101 186 151,010,490
10,000 17,111 198 171,115,614
10,000 12,556 13 125,561,916
10,000 14,662 65 146,626,833
10,000 15,314 385 153,148,910
10,000 18,057 572 180,578,674
10,000 9,380 23 93,805,038
10,000 13,772 51 137,729,978
10,000 15,156 181 151,562,594
10,000 15,732 558 157,322,231
可以看到,基本上大量的样品的细胞的平均检测到的基因数量都少得可怜,可以说是质量非常差了。这就是为什么可以看到文章里面的降维聚类分群结果就很奇怪:
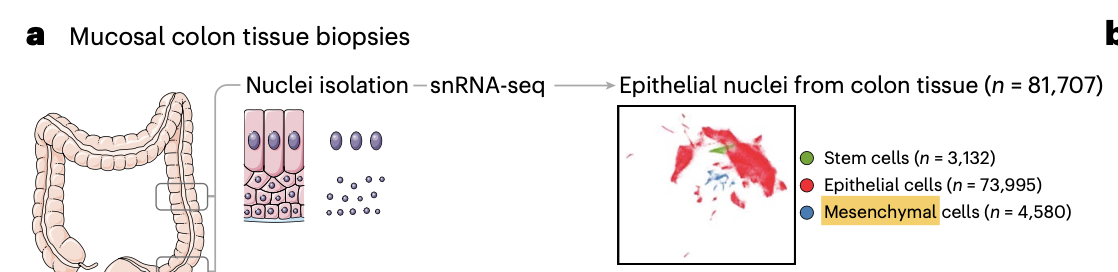
其实研究者并没有在这个单细胞核转录组项目里面进行细胞筛选,所以其实如果你读取它们给出来的表达量矩阵文件后进行降维聚类分群,是可以看到各种不同的免疫细胞的, 如下所示:

我本来呢因为是这些样品质量不好说因为研究者们没有hold住这个单细胞核转录组,但是我下载另外的两个普通单细胞转录组,发现质量也是惨不忍睹, 都需要进行严格的过滤啦。基本上跟前面分享的:好狠的心啊,直接就删除8成的单细胞! 有异曲同工之妙。
主要的问题就是检测到的基因数量不太够
原则上现在大家做10x技术流的单细胞转录组的时候,平均基因检出应该是可以到1000到2000,它这个项目还不到十分之一,所以删除了80%的细胞也不足为奇。而且,如果进行了严格的质量控制后的降维聚类分群,居然效果还不错。起码分群是合理的,不过平均基因检出这么少,其实影响后面的差异分析富集分析等等。