我发现初学者在入门单细胞转录组数据分析的时候会有很多“细枝末节”的过度纠结,比如过分的追求得到文献那样的分群。
学员给我反馈了一个2018的人类肝脏单细胞图谱研究:《Single cell RNA sequencing of human liver reveals distinct intrahepatic macrophage populations》,如下所示的单细胞转录组降维聚类分群图谱可以看到仅仅是肝细胞就可以分成6个群 :

大家可以下载这个100多M的文件(GSE115469_Data.csv.gz),而且可以载入作者自己的降维聚类分群结果,然后走Seurat的单细胞转录组数据分析流程:
ct=data.table::fread('GSE115469_Data.csv.gz',data.table = F)
ct[1:4,1:4]
rownames(ct)=ct$V1
ct=ct[,-1]
phe=data.table::fread('GSE115469_CellClusterType.txt.gz',data.table = F)
rownames(phe)=phe$CellName
phe=phe[,-1]
sce.all=CreateSeuratObject(counts = ct,meta.data = phe)
as.data.frame(sce.all@assays$RNA$counts[1:10, 1:2])
head(sce.all@meta.data, 10)
table(sce.all$orig.ident)
但是如果是大家使用的是我们给大家的单细胞多样品的Seurat的单细胞转录组数据分析流程代码,往往是会得到如下所示的第一层次降维聚类分群结果,而且我把作者的单细胞亚群命名结果映射上去了 :
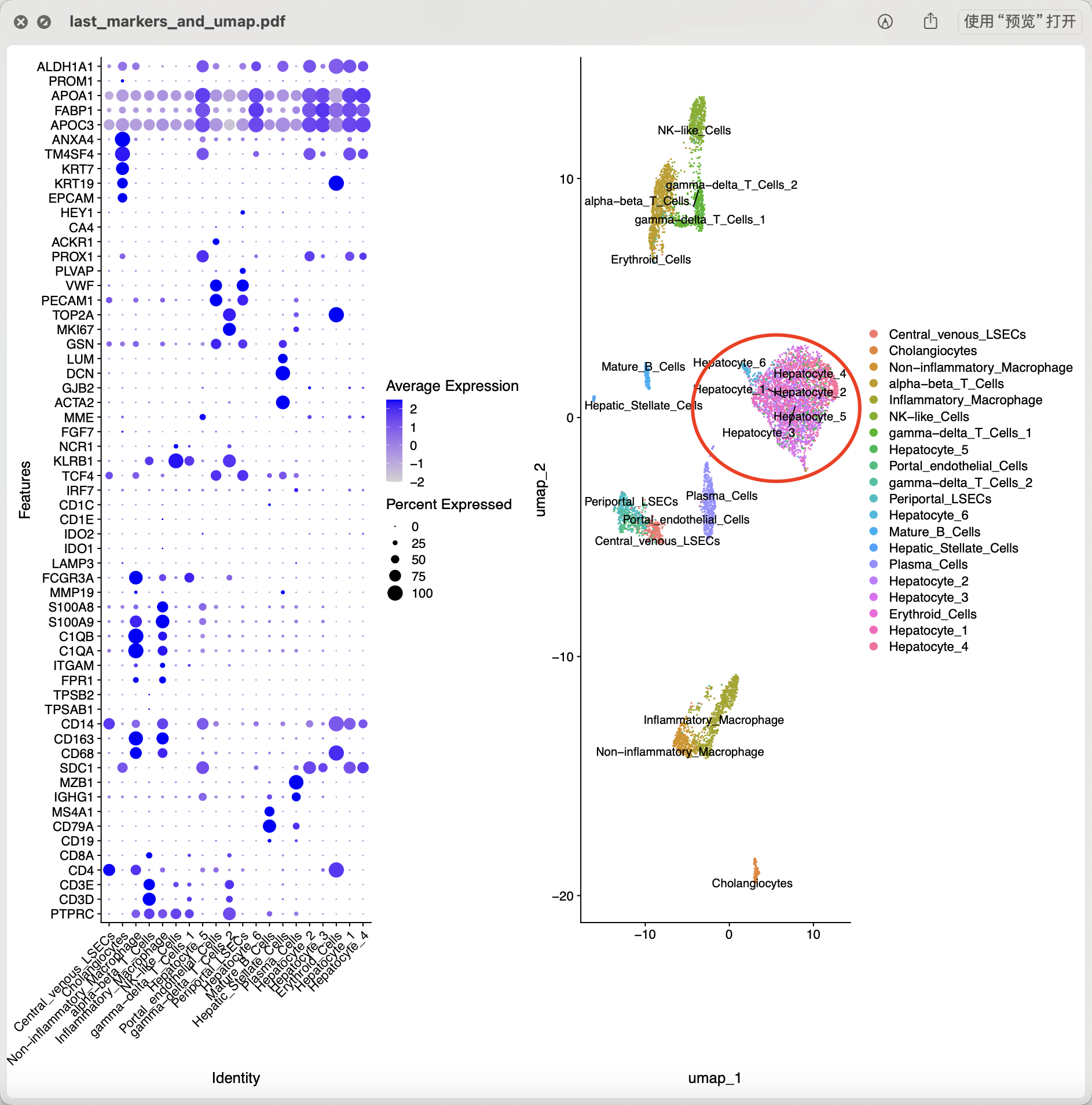
可以看到,其实肝细胞根本就没办法区分成为文章提到的6个亚群,如果仔细检查其实6个亚群的肝细胞就是不同个体的差异:
> table(sce.all$CellType,sce.all$orig.ident)
P1TLH P2TLH P3TLH P4TLH P5TLH
alpha-beta_T_Cells 275 1 2 305 378
Central_venous_LSECs 191 31 8 49 27
Cholangiocytes 8 23 41 13 34
Erythroid_Cells 1 2 9 15 66
gamma-delta_T_Cells_1 67 0 0 111 286
gamma-delta_T_Cells_2 15 5 1 81 3
Hepatic_Stellate_Cells 8 0 24 2 3
Hepatocyte_1 0 0 1004 1 1
Hepatocyte_2 1 0 908 0 0
Hepatocyte_3 0 629 0 0 0
Hepatocyte_4 0 0 603 0 0
Hepatocyte_5 90 0 0 12 100
Hepatocyte_6 5 0 106 30 11
Inflammatory_Macrophage 186 34 214 165 214
Mature_B_Cells 7 7 0 34 81
NK-like_Cells 92 0 0 199 197
Non-inflammatory_Macrophage 97 89 30 129 34
Periportal_LSECs 15 49 61 68 134
Plasma_Cells 4 344 29 77 57
Portal_endothelial_Cells 6 5 87 34 79
因为这个文章比较早期了,那个时候并不流行单细胞转录组的多个样品的整合,比如我们的harmony流程,所以它个体差异会被保留。
不过,比较有意思的是为什么其它单细胞亚群就没有如此强烈的个体异质性呢,这篇文章做的又不是肝细胞癌症样品的单细胞转录组。
学徒作业
完成这个GSE115469数据集的降维聚类分群,然后进行代谢基因集打分,看看是不是因为不同个体的肝细胞的代谢比较旺盛所以体现出来了异质性。