看到了一个新鲜出炉的单细胞数据挖掘文章,标题是:《Single-cell analysis of tumor microenvironment and cell adhesion reveals that interleukin-1 beta promotes cancer cell proliferation in breast cancer》,研究者重新处理了GSE176078这个数据集,里面是26个乳腺癌患者的单细胞表达量矩阵 (five HER2+, 12 ER+/PR+, and nine TNBC samples) 。
很简单的降维聚类分群后,可以看到是如下所示是的主要单细胞亚群 :
- 6500 CAF cells (markers: COL1A1, COL3A1, and DCN),
- 7600 endothelial cells (markers: CLDN5 and CDH5),
- 28200 epithelial cells (markers: EPCAM, KRT8, and KRT18),
- 9500 myeloid cells (markers: CD68, CD163, CD14, and LYZ),
- 3200 B cells (markers: CD79A, CD19, and MS4A1),
- 3500 plasmablast cells (markers: CD79A, MZB1, CD38, and IGHG1),
- 35200 T cells (markers: CD3D, CD3E, and CD8A)
这个是比较容易的:

而且很明显,第一层次降维聚类分群其实是没办法区分 28200 epithelial cells (markers: EPCAM, KRT8, and KRT18), 里面的 恶性与否的上皮细胞,这个时候需要走inferCNV等流程对上皮细胞进行恶性程度的判断。
然后,作者找出来了这26个乳腺癌患者的单细胞表达量矩阵里面的恶性的上皮细胞后,根据病人的临床分组 (five HER2+, 12 ER+/PR+, and nine TNBC samples) 做差异分析,居然得到了如下所示的一个看起来有点奇怪的火山图:
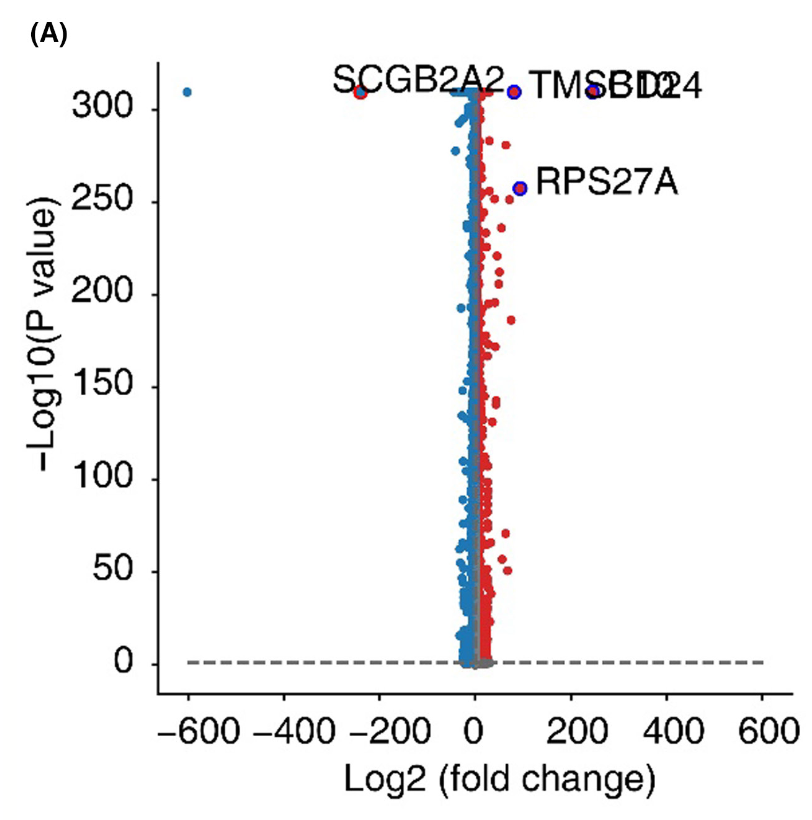
如果是这个火山图是基于bulk表达量矩阵,无论是表达量芯片还是转录组测序,的差异分析后的可视化,很明显就有问题的。一般来说log2FC绝大部分都是5以内,大于10的都很少很少了。但是上面的差异分析结果来源于恶性上皮细胞的单细胞表达量矩阵分组,所以有可能是合理的,仅仅是火山图的展示方式不太好了。学徒作业
重新重新处理了GSE176078这个数据集,里面是26个乳腺癌患者的单细胞表达量矩阵,然后对它进行严格的质量控制后,做出来第一层次降维聚类分群后,提取里面的上皮细胞进行细分亚群后,鉴定里面的恶性情况,然后取里面的恶性上皮细胞根据tnbc与否进行二分组然后进行差异分析哈!
如果你想完成上面的学徒作业,最好是有自己的计算机资源哦,比如我们的2024的共享服务器交个朋友福利价仍然是800,而且还需要有基本的生物信息学基础,也可以看看我们的生物信息学马拉松授课(买一得五) ,你的生物信息学入门课。
如果你有了生物信息学基础,仅仅是感兴趣单细胞的细致分析,就看看SBC的 Nature年度技术 | 单细胞及空间多组学实验技术与生信分析培训(暑期班)招生火热进行中