看到了一个简单的单细胞转录组数据挖掘文章:《Single-cell analysis revealed that MTIF2 could promote hepatocellular carcinoma progression through modulating the ROS pathway》,这个研究就是针对GSE242889数据集进行了一个简单的降维聚类分群,然后进行合理的单细胞亚群的生物学命名,如下所示:
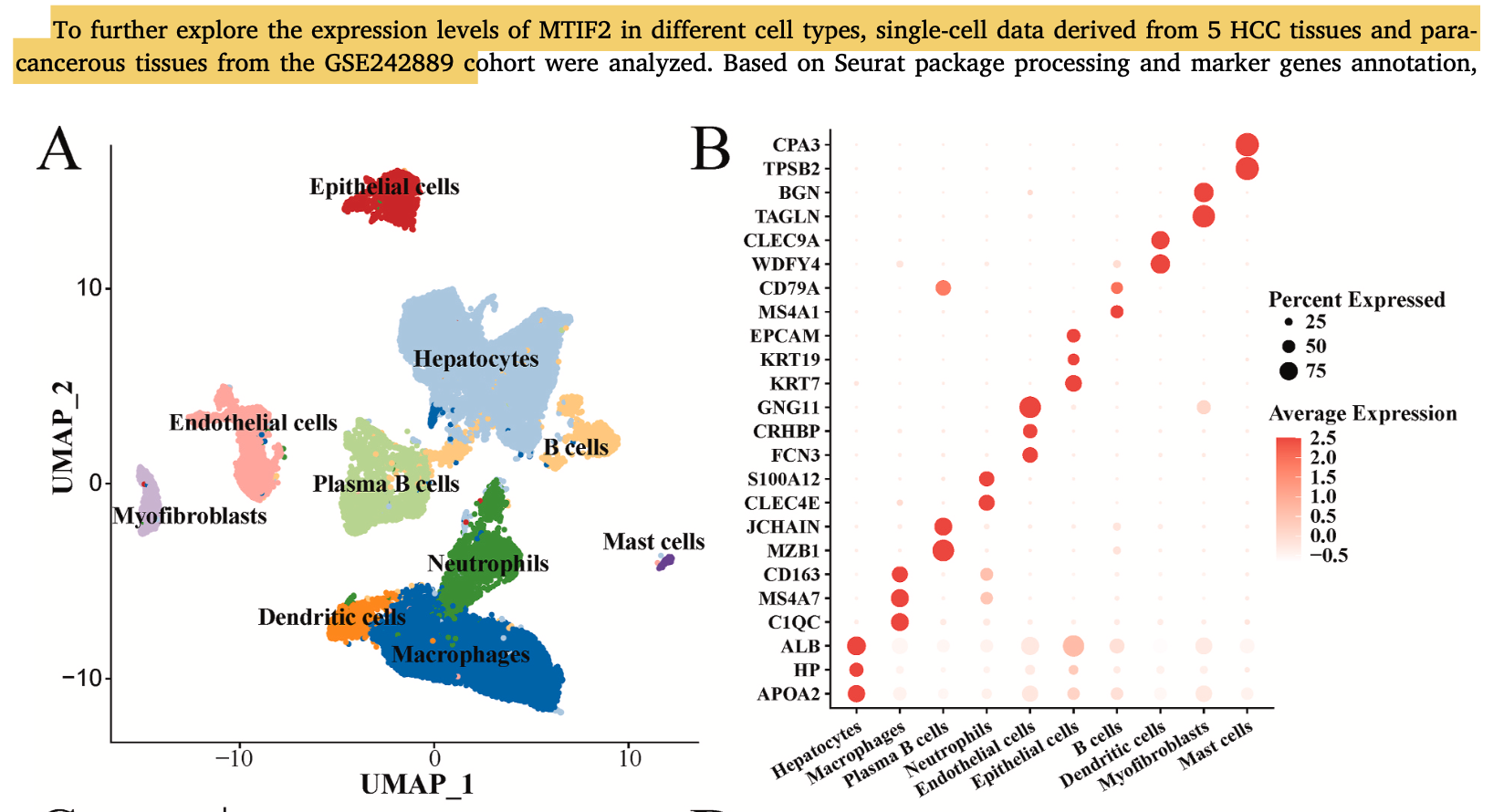
很容易读取GSE242889数据集,因为它是标准的10x单细胞转录组,而且是比较早期的。
然后因为这个单细胞转录组数据集是有分组的, 肝癌的癌症样品和癌旁组织,就可以看目标基因在具体的单细胞亚群的表达量高低情况,这个时候不需要进行差异分析,因为目标基因是确定的, 如下所示:
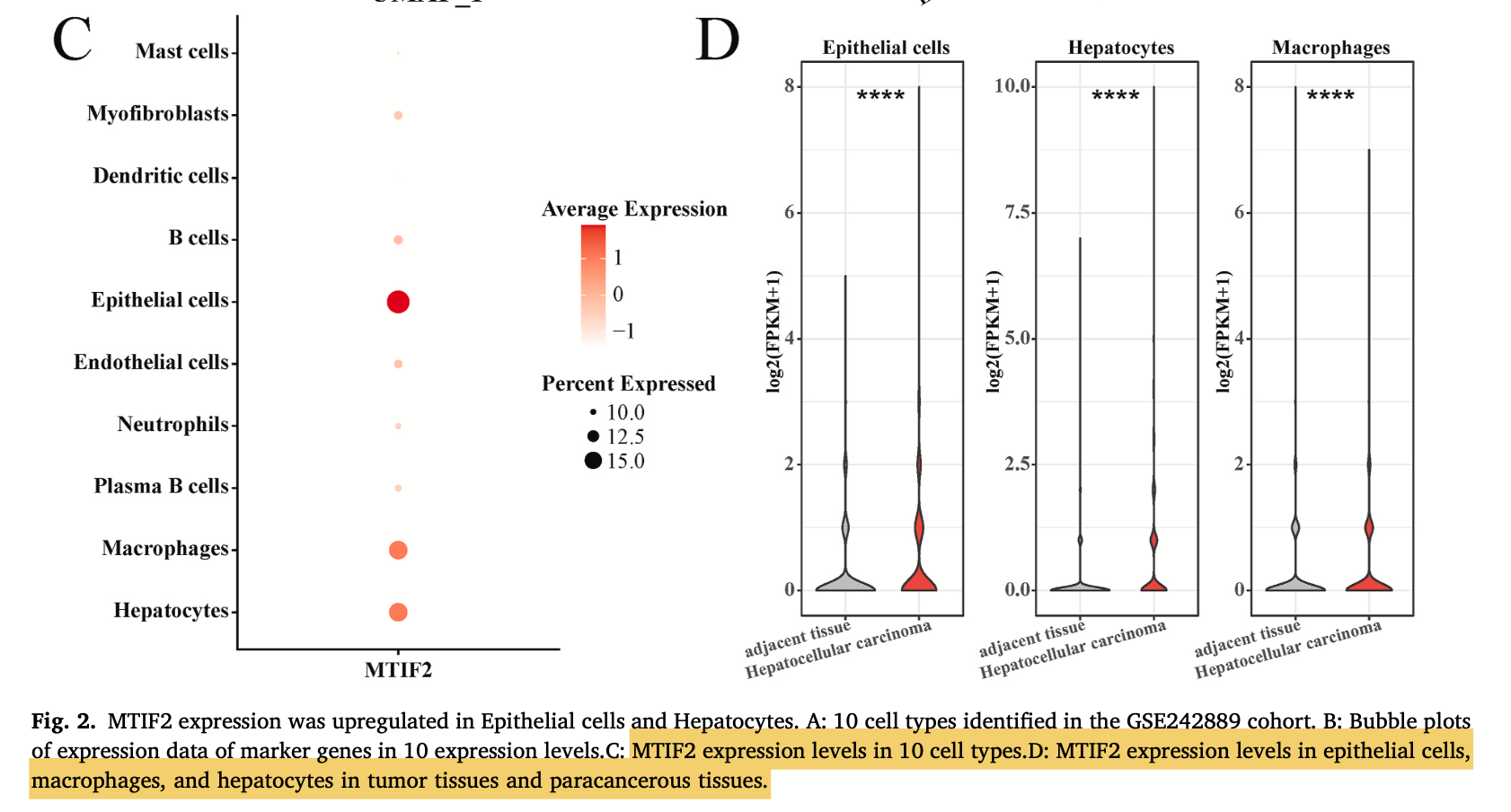
但是,我怀疑上面的这个分析是有问题,因为上面的样品的两个分组的, 肝癌的癌症样品和癌旁组织,里面的这3个单细胞亚群的细胞数量是不一样的,而且上面压根就不需要是FPKM,更别说是log2(FPKM+1) 这样的表达量格式了。
我尝试了一下简单的降维聚类分群和命名,然后看了看如下所示的细胞数量情况 (其中 文章里面指出来了 NT 是 non-tumor; ):
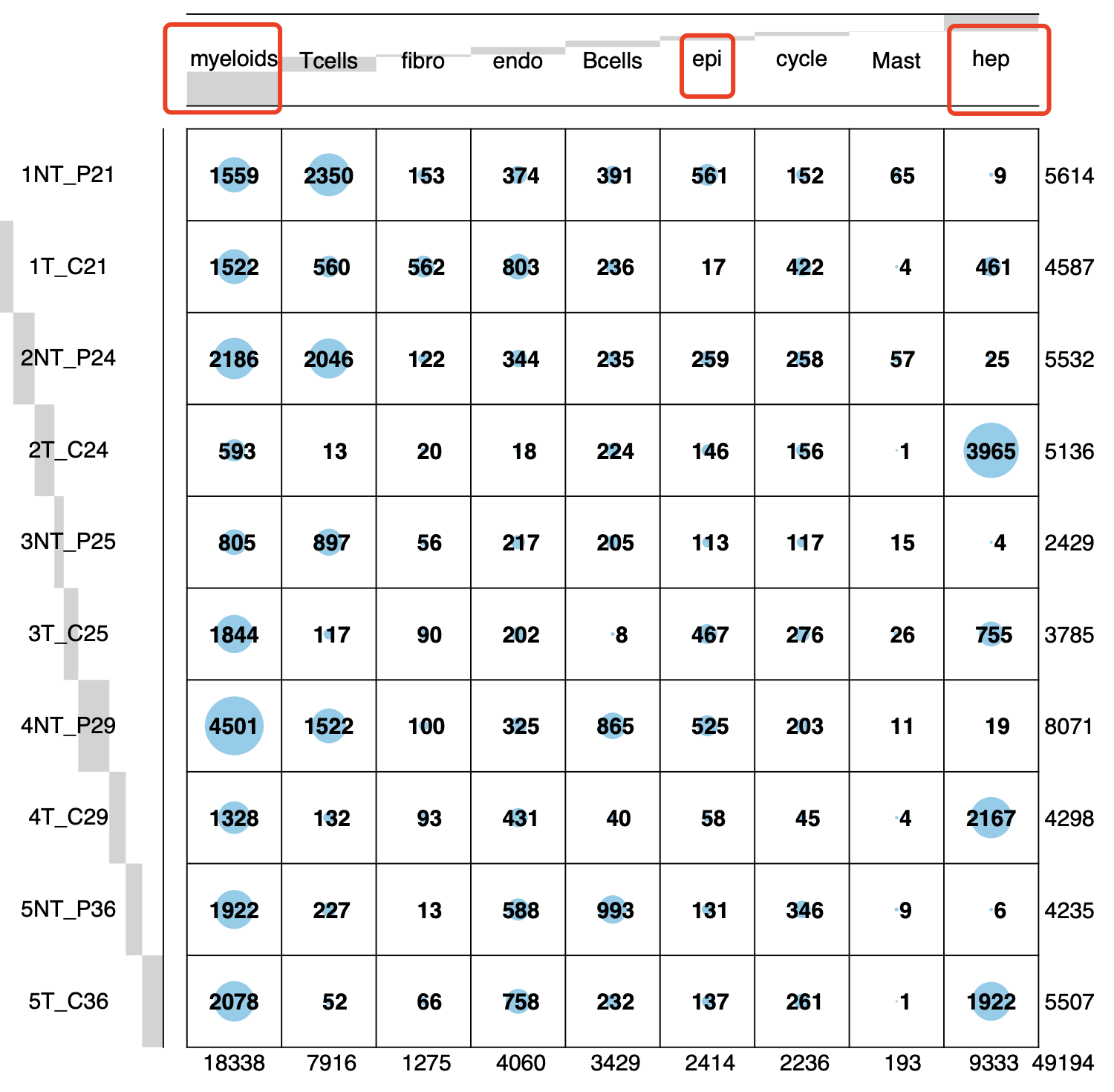
让我感到困惑的是肝细胞因为是正常的细胞,但是它绝大部分都存在于癌组织,在癌旁里面很少????
另外就是上皮细胞是有可能是会恶化的,所以它在 肝癌的癌症样品和癌旁组织都是有的!
看看 MTIF2 是否有细胞亚群特异性
可以看到确实是这个MTIF2基因在肝细胞里面的三分之一的阳性,但是并不能说它在巨噬细胞和上皮细胞里面的特殊性 :
> t(table(MTIF2,sce.all$celltype))
MTIF2
FALSE TRUE
Bcells 3069 360
cycle 1823 413
endo 3612 448
epi 1971 443
fibro 1131 144
hep 6294 3039
Mast 186 7
myeloids 15817 2521
Tcells 7413 503
另外就是看看这个MTIF2基因在肝细胞在上面的样品的两个分组的, 肝癌的癌症样品和癌旁组织,是否有差异呢?我也是简单的统计一下:
FALSE TRUE
1NT_P21 7 2
1T_C21 297 164
2NT_P24 20 5
2T_C24 2385 1580
3NT_P25 4 0
3T_C25 480 275
4NT_P29 9 10
4T_C29 1755 412
5NT_P36 3 3
5T_C36 1334 588
就很难说清楚,虽然是这个这个MTIF2基因在肝细胞的特异性高表达是事实,但是并不能说它在肝癌病人的癌症样品和癌旁组织是有差异的。。。
学徒作业
大家试试看,处理GSE242889数据集,搞清楚里面的上皮细胞和肝细胞的恶性情况,还有它是否有MTIF2基因的特异性!