学员在掌握了我们的授课的GEO数据挖掘之后通常是会试试看自己感兴趣的科研领域的表达量芯片或者转录组测序公开数据集,然后就各自碰壁,因为绝大部分文献在公开自己的数据的时候往往是会埋一些不大不小的坑。
比如学员就反馈了2020的一个美国纽约的哥伦比亚大学的阿兹海默症研究文章:《T Cell Responses to Neural Autoantigens Are Similar in Alzheimer’s Disease Patients and Age-Matched Healthy Control》, 对应的数据集是GSE153104,可以看到研究者关注的应该是Alzheimer’s disease (AD),和healthy controls (HC).的转录水平的变化,而且还具体到了不同的细胞亚群:
- PBMCs (HC n=28 and AD n=27),
- CD4 memory (HC n=28 and AD n=27)
- CD8 memory (HC n=30 and AD n=26) T cells
这是一个大队列的转录组了,166个转录组测序在2019之前在美帝那边起码耗费二十万人民币经费。但是呢,我们很容易读取作者给出来的表达量矩阵文件进行简单的质量控制,如下所示:
我在生信技能树的教程:《你确定你的差异基因找对了吗?》提到过,必须要对你的转录水平的全局表达矩阵做好质量控制,最好是看到标准3张图: - 左边的热图,说明我们实验的两个分组,normal和npc的很多基因表达量是有明显差异的
- 中间的PCA图,说明我们的normal和npc两个分组非常明显的差异
- 右边的层次聚类也是如此,说明我们的normal和npc两个分组非常明显的差异
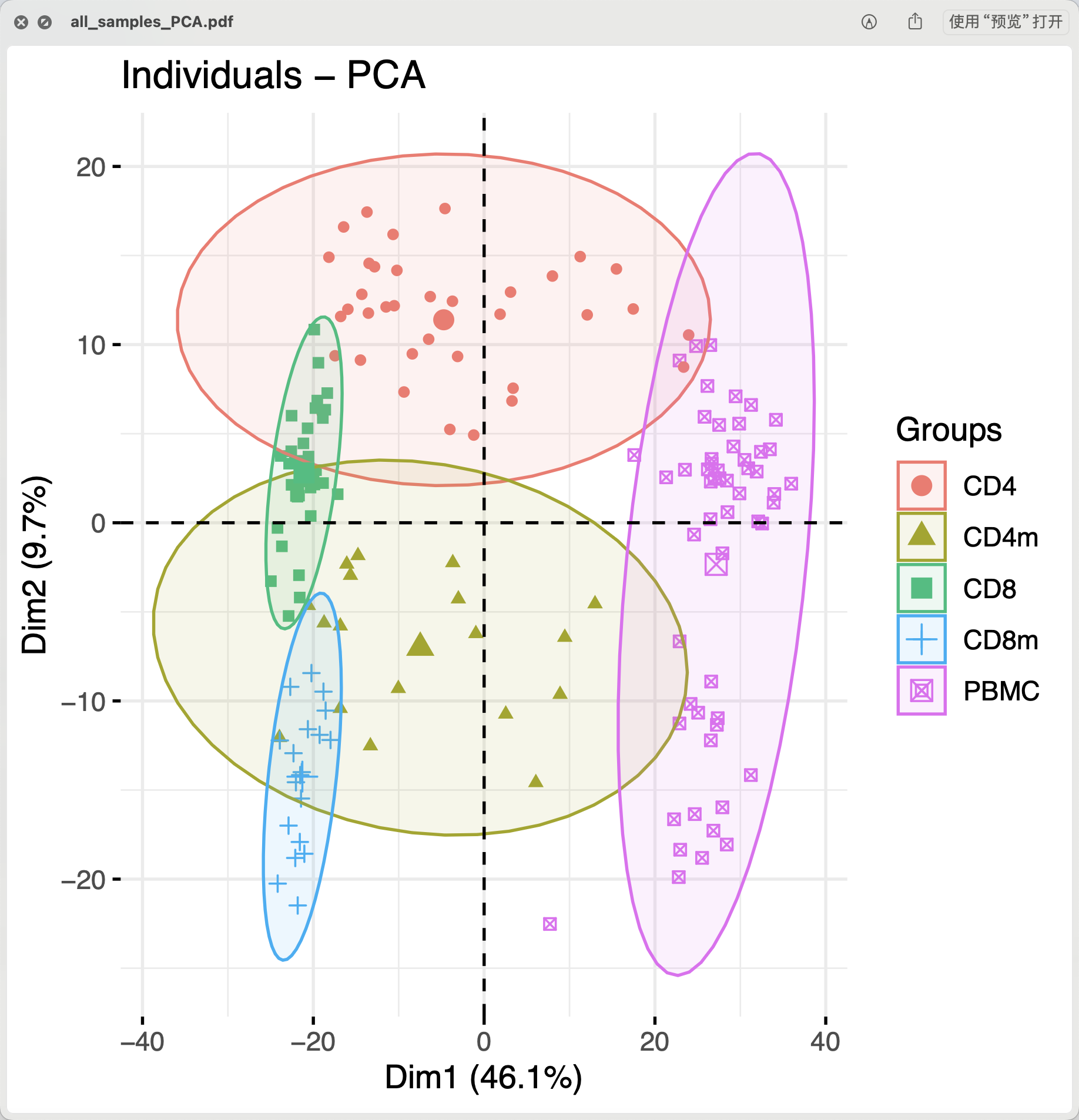
如果分组在3张图里面体现不出来,实际上后续差异分析是有风险的。这个时候需要根据你自己不合格的3张图,仔细探索哪些样本是离群点,自行查询中间过程可能的问题所在,或者检查是否有其它混杂因素,都是会影响我们的差异分析结果的生物学解释。CD4 CD4m CD8 CD8m PBMC Alzheimer's disease 18 9 17 9 27 Healthy control 19 9 20 10 28上面的PCA图就可以看到,其实作者给出来的表达量矩阵主要的差异完全是取样材料的差异,压根就很难定位到究者关注的应该是Alzheimer’s disease (AD),和healthy controls (HC).的转录水平的变化。研究者们也做了3次差异分析,如下所示的火山图 :
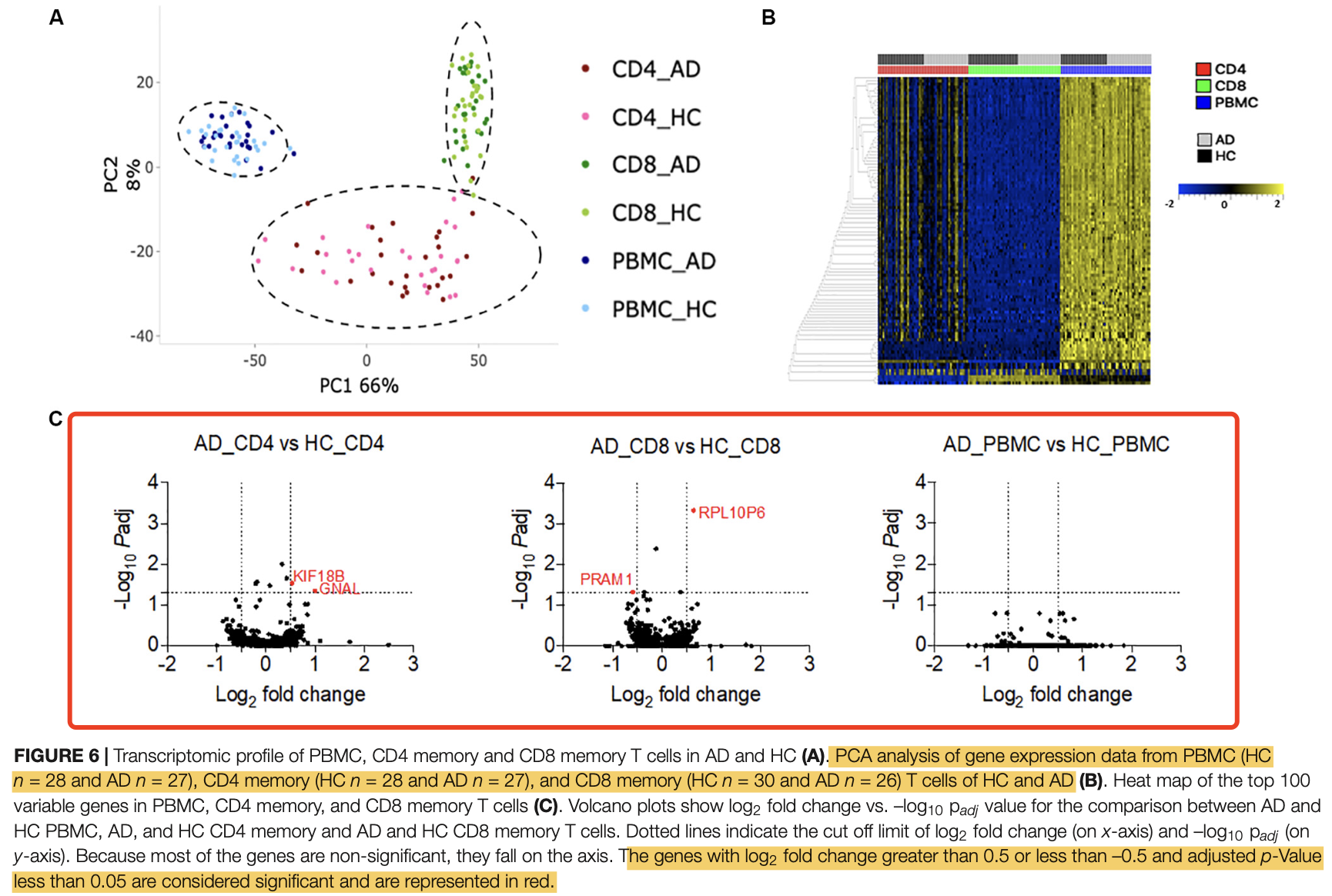
可以看到确实是符合统计学显著的上下调差异基因数量非常少,都不需要我们来复现这个。
而且不仅仅是这些不同细胞亚群在Alzheimer’s disease (AD),和healthy controls (HC).的两个分组是没有全局的转录水平的变化,而且如果是取这两个分组样品进行流式细胞,也基本上是阴性结果:
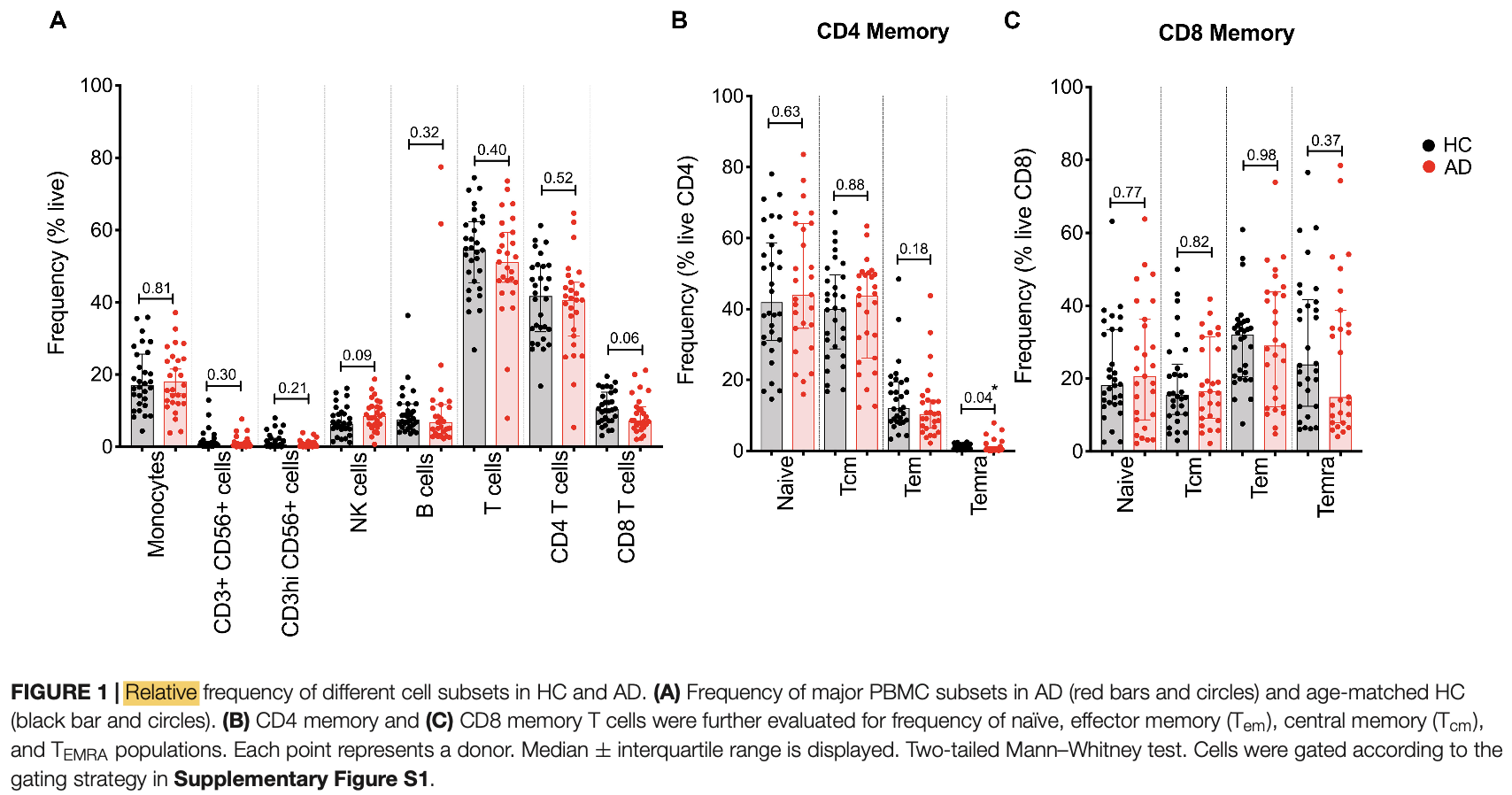
流式细胞仪甚至是更贵一点,但是因为无论是阿兹海默症患者,还是正常志愿者,都是直接抽血然后去做实验。Whole blood was collected in EDTA vacutainers and PBMCs were isolated by density gradient centrifugation with Ficoll-Paque plus
这样其实就天然不太可能有差异,前面我在生信技能树推文:你确定你的差异基因找对了吗? 提出了文章的转录组数据的60个样品并没有按照毒品上瘾与否这个表型来区分,而是不同人之间的异质性非常高,这个时候我提出来了一个解决方案,就是理论上就可以把人当做是一个批次效应,使用sva包的combat函数,把这样的效应去除一下,接着再找差异。
类似的, control组以及毒品组,基本上是无法区分开来的,然后呢,这个文章Alzheimer’s disease (AD),和healthy controls (HC).的两个分组是没有全局的转录水平的变化。什么是合理的实验设计看疾病和正常对照的差异
在转录组研究中,疾病状态和正常对照之间的比较确实是一种常见的实验设计。当疾病主要影响特定组织或器官时(如癌症),通常首选的是直接从患病组织(例如肿瘤组织)中提取样本进行分析,因为这样可以更直接地观察到与疾病相关的分子变化。然而,如果研究设计仅限于从患者和健康对照者抽取血液样本,那么在血液中观察到的转录组差异可能不如直接从病变组织中获得的样本那么显著或特异。血液样本中的基因表达差异可能反映了:
- 全身性反应:某些疾病可能会引起全身性的炎症反应或其他系统性变化,这些变化可以在血液细胞的基因表达中反映出来。
- 疾病微环境的影响:肿瘤患者血液内的细胞可能受到肿瘤微环境释放的信号的影响,导致基因表达的变化。
- 治疗效应:如果患者已经接受了治疗,血液样本中的基因表达差异可能部分反映了治疗的影响。
- 疾病进展阶段:在疾病早期,血液样本中的基因表达差异可能不如晚期疾病那样明显。
- 个体差异:不同个体对疾病和治疗的反应存在差异,这可能会影响血液样本中的基因表达模式。
- 血液细胞组成变化:疾病状态可能会影响血液中不同类型细胞的比例,从而影响整体的基因表达谱。
尽管从血液样本中获得的转录组数据可能包含有关疾病状态的信息,但这些信息可能不如直接从病变组织中获得的数据那样具体和直接。此外,血液样本中的基因表达差异可能更难以解释,因为它们可能受到多种因素的影响,包括疾病本身的影响、治疗效应、个体的生活方式和遗传因素等。
因此,如果研究目的是要精确地描述疾病特有的分子变化,那么首选应该是从病变组织中获取样本。然而,血液样本具有易于获取和较少侵入性的优点,可以用于初步筛查、监测疾病进展或治疗反应等目的。在分析血液样本的转录组数据时,研究人员需要谨慎解释结果,并考虑到可能影响基因表达的其他因素。同理,精神疾病和神经退行性疾病的患者应该是如何取样呢
首先,精神疾病和神经退行性疾病的患者大概率上并不需要做大脑区域的手术,就没有病变部位的组织。以下是一些常见的精神疾病和神经退行性疾病的中英文对照列表:
精神疾病: - 抑郁症 - Major Depressive Disorder (MDD)
- 双相情感障碍 - Bipolar Affective Disorder
- 精神分裂症 - Schizophrenia
- 焦虑症 - Anxiety Disorders
- 强迫症 - Obsessive-Compulsive Disorder (OCD)
- 创伤后应激障碍 - Post-Traumatic Stress Disorder (PTSD)
- 边缘型人格障碍 - Borderline Personality Disorder (BPD)
- 注意力缺陷多动障碍 - Attention Deficit Hyperactivity Disorder (ADHD)
- 饮食障碍 - Eating Disorders (如: 神经性厌食症 Anorexia Nervosa, 神经性贪食症 Bulimia Nervosa)
- 物质使用障碍 - Substance Use Disorders (如: 酒精使用障碍 Alcohol Use Disorder)
神经退行性疾病: - 阿尔茨海默病 - Alzheimer’s Disease (AD)
- 帕金森病 - Parkinson’s Disease (PD)
- 亨廷顿病 - Huntington’s Disease (HD)
- 肌萎缩侧索硬化症 - Amyotrophic Lateral Sclerosis (ALS)
- 额颞叶变性 - Frontotemporal Lobar Degeneration (FTLD)
- 路易体痴呆 - Dementia with Lewy Bodies (DLB)
- 多发性硬化症 - Multiple Sclerosis (MS)
- 脊髓小脑变性 - Spinocerebellar Degeneration (SCD)
- 神经性系统性淀粉样变性 - Prion Diseases (如: 克雅氏病 Creutzfeldt-Jakob Disease, CJD)
- 神经纤维瘤病 - Neurofibromatosis
请注意,这个列表并不全面,精神疾病和神经退行性疾病的种类繁多,每种疾病都有其特定的临床表现、病理特征和治疗方法。
针对酒精成瘾、精神疾病、神经退行性疾病等状况进行血液样本的转录组测序,并与正常对照组的血液样本进行比较,可以揭示与这些疾病相关的基因表达变化。然而,不同分组的转录组全局表达量的差异程度可能会有显著差异,这主要取决于以下几个因素: - 疾病严重程度:疾病或成瘾的严重程度可能会影响基因表达的差异性。例如,重度酒精成瘾患者可能表现出与轻度患者不同的基因表达模式。
- 疾病阶段:不同疾病阶段的基因表达谱可能有所不同。例如,在精神疾病的早期阶段可能观察到的基因表达变化与慢性阶段或治疗后的表达变化不同。
- 疾病类型:不同类型的精神疾病或神经退行性疾病可能影响不同的生物学途径和基因集,导致基因表达的差异性。
- 样本选择:血液样本的类型(如全血、PBMCs等)和采集、处理方法可能会影响转录组数据的质量。
- 对照组的选择:对照组的健康状况、年龄、性别和遗传背景等因素需要与病例组匹配,以减少混杂因素的影响。
- 环境和生活方式因素:吸烟、饮食、睡眠和压力等生活方式因素也可能影响基因表达,并可能在病例组和对照组之间产生差异。
- 药物使用:酒精成瘾患者或其他精神疾病患者可能正在接受药物治疗,这可能会影响基因表达模式。
- 个体差异:即使在相同的疾病条件下,不同个体之间也可能存在显著的基因表达差异。
- 数据的统计分析:转录组数据的分析方法,包括差异表达分析、通路分析和多变量分析等,也会影响对全局表达差异的解释。
因此,虽然可以预期在这些疾病状态下会观察到与正常对照组相比的基因表达差异,但具体的差异程度需要通过实验和统计分析来确定。通过这些分析,研究人员可以识别与特定疾病相关的生物标志物,并进一步了解疾病的分子机制,为诊断、治疗和疾病管理提供信息。大家试试看
我简单的搜索了一些阿兹海默症的数据集,如下所示:
- 数据集:从GSE157827,是12名阿尔茨海默病(AD)患者和9名正常对照(Normal)样本的死后人脑前额叶皮质的单细胞表达谱数
- 2021-GSE168522-AD疾病的PBMC单细胞
- 2021-GSE181279-AD疾病的PBMC单细胞
给大家作为学徒作业吧,看看同样的pbmc,单细胞水平和普通转录组数据里面的,Alzheimer’s disease (AD),和healthy controls (HC).的两个分组的差异,是否有一致性。另外如果是取样直接就病变部位,是否是差异更加的明显呢?