customTrack,我这里翻译为自定义的测序片段示踪文件,可以追踪我们的reads到底比对到了参加基因组的什么区域,或者追踪参考基因组的各个区域的覆盖度,测序深度!翻译自:http://genome.ucsc.edu/goldenPath/help/customTrack.html 这个非常有用!!!
UCSC提供的Genome Browser工具非常好用,可以很方便的浏览我们的测序数据在参考基因组的比对情况,由于定义好了一系列track的文件格式,用户可以非常方便的上传自己的track文件,但是如果用户超过48小时没有浏览自己的数据,UCSC会默认删除掉这些数据,除非用户已经保存在session里面。或者用户可以分享这些自定义的reads示踪文件customTrack。
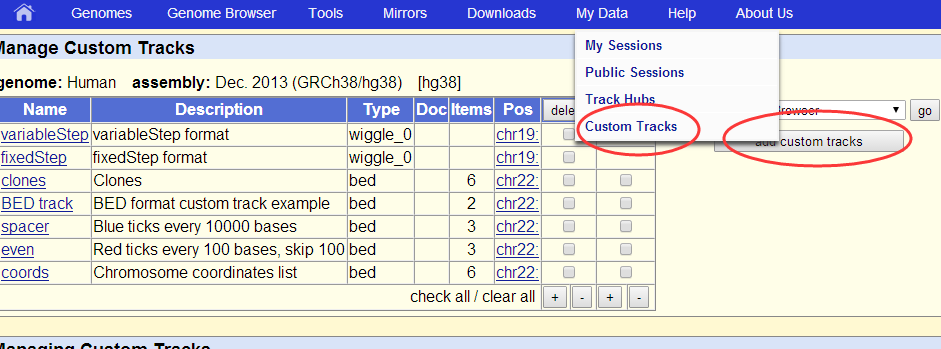
UCSC已经提供了一系列customTrack的例子:click the Custom Tracks link
这些自定义的Track文件保密性非常好,如果用户感兴趣,可以按照以下4个步骤来操作:
Step 1. Format the data set
我们支持非常多的Track文件格式,尤其是标准的GFF文件,还包括:bedGraph, GTF, PSL, BED, bigBed, WIG, bigGenePred, bigMaf, bigChain, bigPsl, bigWig, BAM,CRAM, VCF, MAF, BED detail, Personal Genome SNP, broadPeak, narrowPeak, and microarray (BED15).
染色体一定是chrN 类型的标记,大小写敏感!也支持多种或者多个annotation的track文件。
Step 2. Define the Genome Browser display characteristics
设置浏览器选项,是否在Genome Browser里面显示UCSC的其它数据类型,包括hide/dense/pack/squish/full各种选项,包括ENCODE计划等各种公共数据是否需要显示。Add one or more optional browser lines to the beginning of your formatted data file to configure the overall display of the Genome Browser when it initially shows your annotation data.
这个非常复杂,但是一般就定义有限的几个属性即可。
Step 3. Define the annotation track display characteristics
设置如何显示自己的数据,包括颜色,数据名,数据描述情况。Following the browser lines--and immediately preceding the formatted data--add a track line to define the display attributes for your annotation data set.
下面这幅图里面的一些track的颜色,形状,注释,都是可以设置的,设置规则需要自己详细读说明书啦。
Step 4. Display your annotation track in the Genome Browser
重点就是上传自己的文件,步骤是:
open the Genome Browser home page ,click the Genome Browser link in the top menu bar.
On the Gateway page that displays, select the genome and assembly on which your annotation data is based, then click the "add custom tracks" button.
On the Add Custom Tracks page, load the annotation track data or URL for your custom track into the upper text box and the track documentation (optional) into the lower text box, then click the Submit button. Tracks may be loaded by entering text, a URL, or a pathname on your local computer.
用户可以提交多种格式的自定义track文件
提交完毕之后,直接回到 Genome Browser 页面就可以看到了,这个工具不默认跳转。
Step 5. (Optional) Add details pages for individual track features
Step 6. (Optional) Share your annotation track with others
这是可选的步骤,自己去探索:read the section Sharing Your Annotation Track with Others.
我这里添加了一个UCSC也提供的一个wig文件:http://genome.ucsc.edu/goldenPath/help/examples/wiggleExample.txt 作为测试例子,显示如下: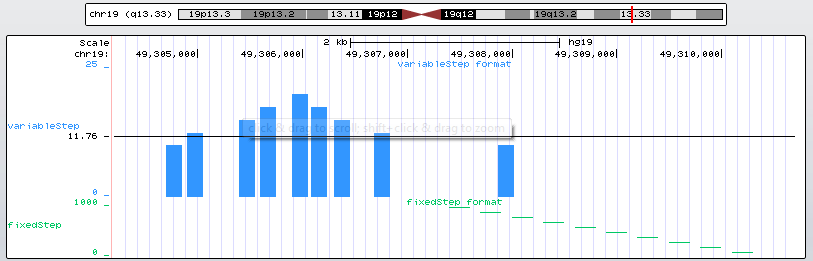



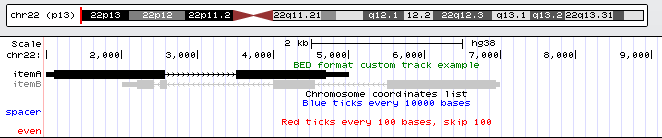
 看到下面的图片的链接,点进去就好啦
看到下面的图片的链接,点进去就好啦