在KRAS-TRP53基因驱动的LADC依赖于rRNA的合成依赖于Ect2基因
今天JC的时候,同学分享的是一个比较难懂的文章,我尝试着follow她,但失败了。文章发表于2017年1月的CELL,是 Ect2-Dependent rRNA Synthesis Is Required for KRAS-TRP53-Driven Lung Adenocarcinoma , 里面太多陌生的名词,如下:
-
nucleolar transcription factor upstream binding factor 1 (UBF1)
-
guanine nucleotide exchange factor (GEF) epithelial cell
-
transforming sequence 2 (Ect2) - oncogene
-
-
rDNA promoters
-
lung adenocarcinoma (LADC)
-
KRAS-TRP53-Driven Lung Adenocarcinoma
-
CD24+/ITGB4+/NOTCH3hi (3+) lung epithelial cells
-
Madin-Darby canine kidney (MDCK) epithelial cells
-
Lentiviral short hairpin RNA (shRNA)-mediated knockdown (KD)
-
mouse-specific Ect2 shRNAs
-
cytoplasmic Rho GTPases
-
Ect2 nuclear localization signal [NLS]
-
H358 and H23 cells
-
anti-rheumatoid agent auranofin (ANF)
太多了,我都懒得继续看了。
不过,这样的大文章已经不可避免的利用公共数据了,比如TCGA。对我来说,这反而是非常简单的了,我就来试着解读一下。
TCGA表达量看共表达情况
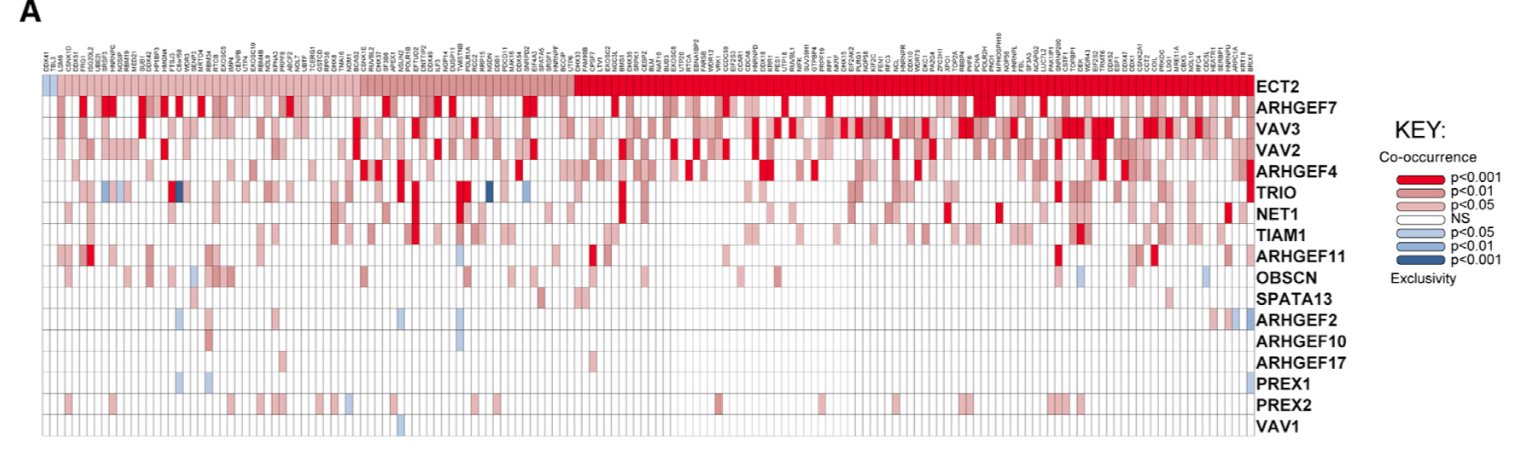
Co-occurrence analysis of the TCGA LADC dataset (n = 517) for expression of ribosomal processing genes and Rho family GEFs. Significant co-occurrences shown in red and exclusivity in blue.
如果要重复这个图,需要下载表达矩阵,然后要根据文献找到那 286 validated human ribosomal processing genes ,然后跟作者感兴趣的那十几个基因比较即可。
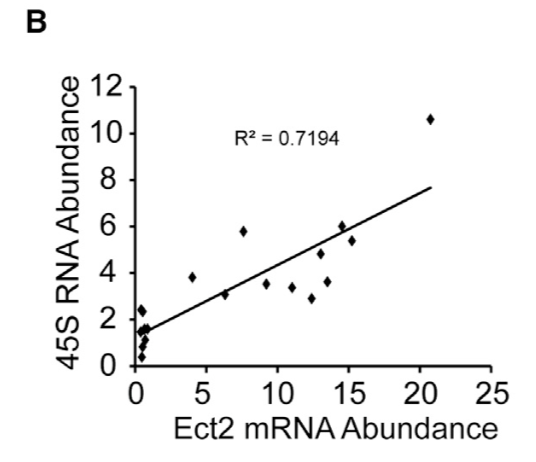
TCGA查询某两个基因的表达相关性
这里作者并没有选取全部的LADC数据,而是选取其中的有KRAS基因突变的样本,所以需要同步下载WES数据的MAF文件,查询突变的那19个样本ID,再去表达矩阵里面查询并且作图。
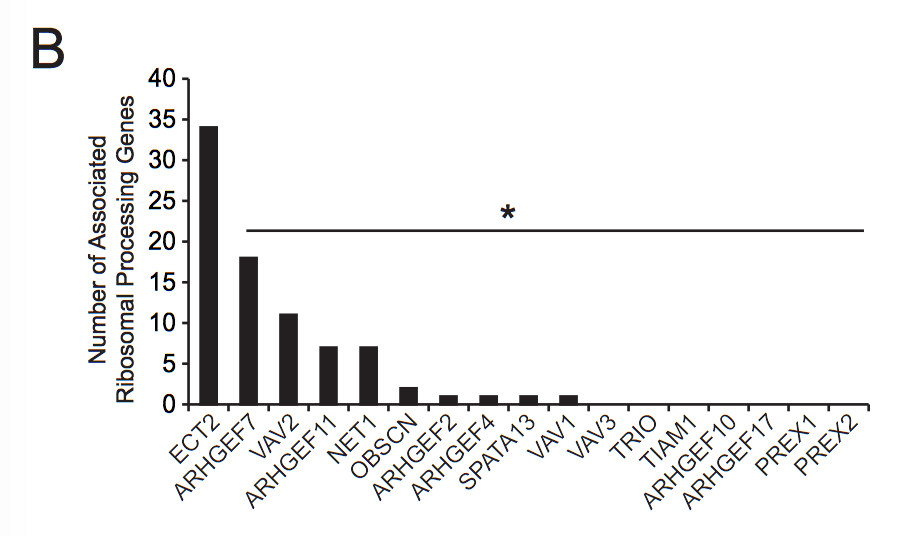
对共表达结果热图进行统计
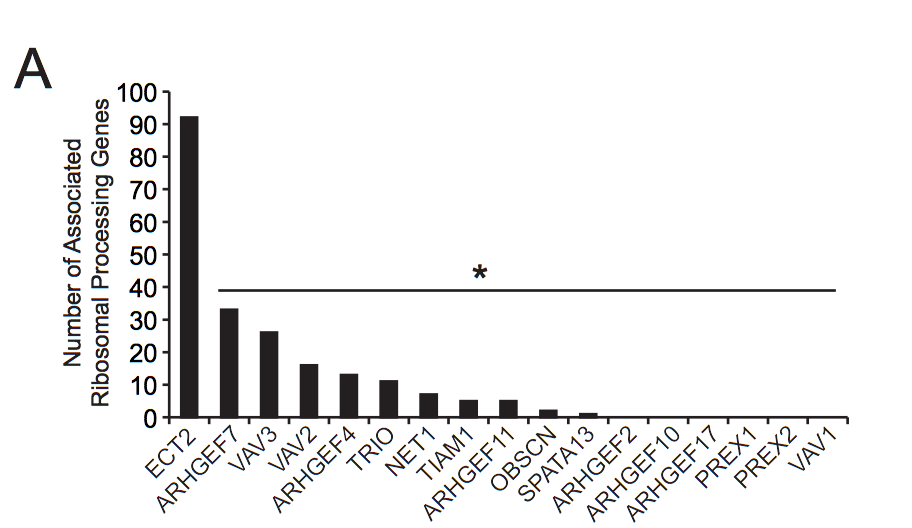
这个很简单,就是数一数上面的热图,对每个基因统计一下有多少个显著的 ribosomal processing genes 。
统计结果再过滤
只留下KRAS基因突变的样本,后再进行统计分析。
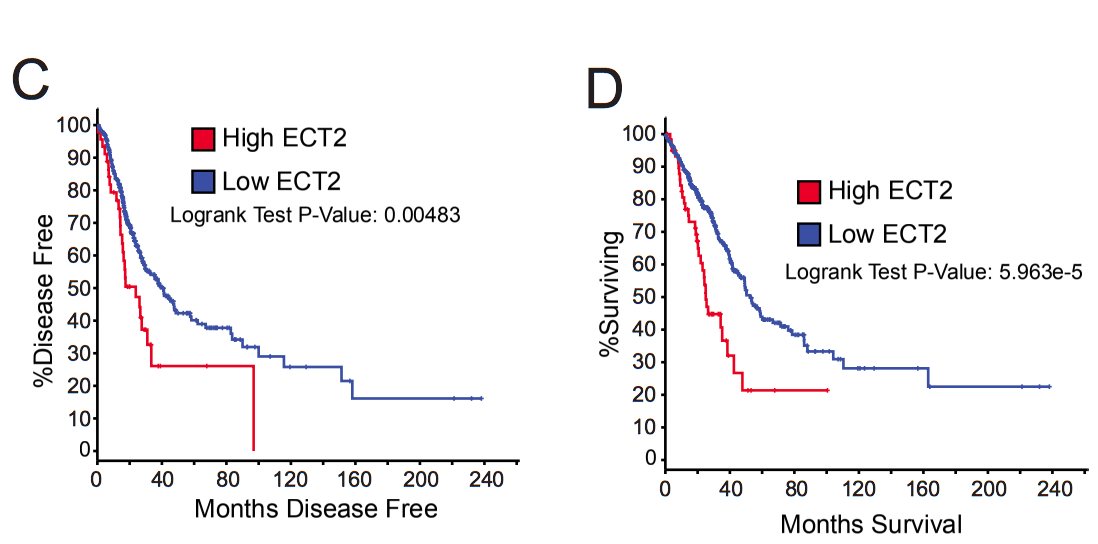
生存分析
这个有很多在线工具可以做了: