PDAC背景知识
胰腺癌生长快而且转移率高,是一种非常致命的恶性疾病。2016年美国癌症协会的数据显示,胰腺癌患者的5年生存率在7-8%之间。虽然手术可以根治这种疾病,但胰腺癌在确诊时只有10–20%是可切除的,其它患者只能接受化疗。
2月8日在Nature Communications上发表的GWAS研究纳入了21,536人,发现位于人类染色体1(位置1p36.33)、7(位置7p12)、8(位置8q21.11)、17(位置17q12)和18(位置18q21.32)上的新鉴定的遗传变异可增加胰腺癌风险。这些基因组中每个拷贝的存在都会增加胰腺癌风险15%-25%。
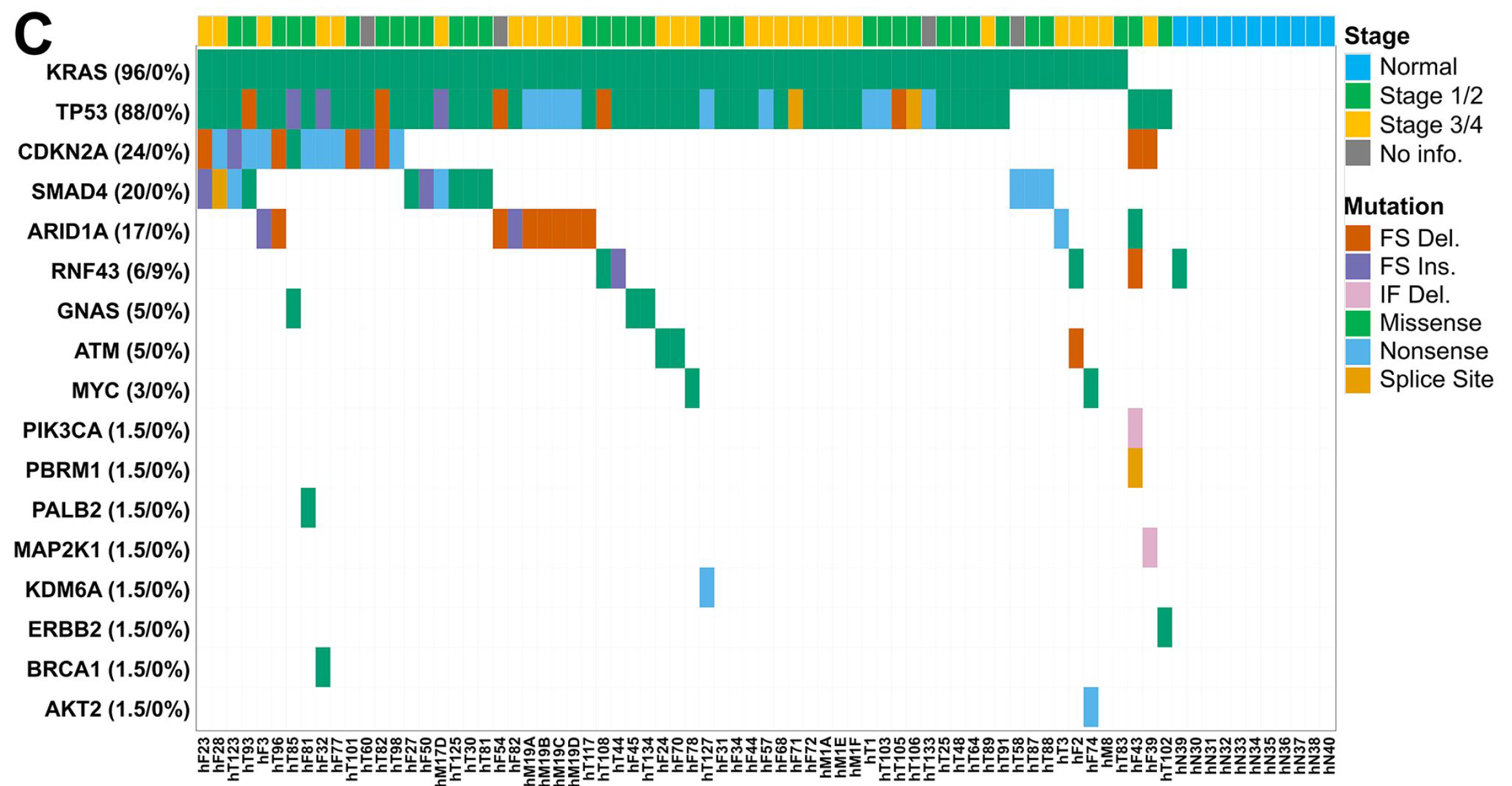
关于胰腺癌发生的缘起假说目前主要集中在两个学说,一个是最早由肿瘤遗传学传奇人物Alfred G Knudson提出的肿瘤的“二次打击学说”,其后由诸多胰腺癌研究领域的科学家完善并确定提出的“Kras-CDKN2A-P53-SMAD4”成次序改变和逐步积累的描述胰腺肿瘤由PanINs发展至PDAC的学说体系。自此,古老的基于病理形态的学科体系与新型的基于基因分子生物分型的学科体系变得不再各自孤立:近20年间的系列研究在胰腺癌中比较有代表性地强调了胰腺癌病理形态改变和分子生物学改变的平行相关性。而2017年的一项来自dana farber和哈弗医学院的最新研究更是在356例胰腺癌样本中较为全面和完整地的探索四个关键驱动基因改变与胰腺癌预后的相关性(四大驱动基因中,KRAS和CDKN2A与临床预后紧密相关;与此同时,驱动基因的突变个数越多,预后越差)
TCGA数据分析PDAC分成两个大类:
-
The Basal-like, Squamous or Quasi-mesenchymal, subtype identifies PDAC patients with poor prognosis and is characterized by basal markers such as cytokeratins.
-
The Classical or Pancreatic Progenitor subtype is characterized by differentiated ductal markers and identifies patients with a better prognosis.
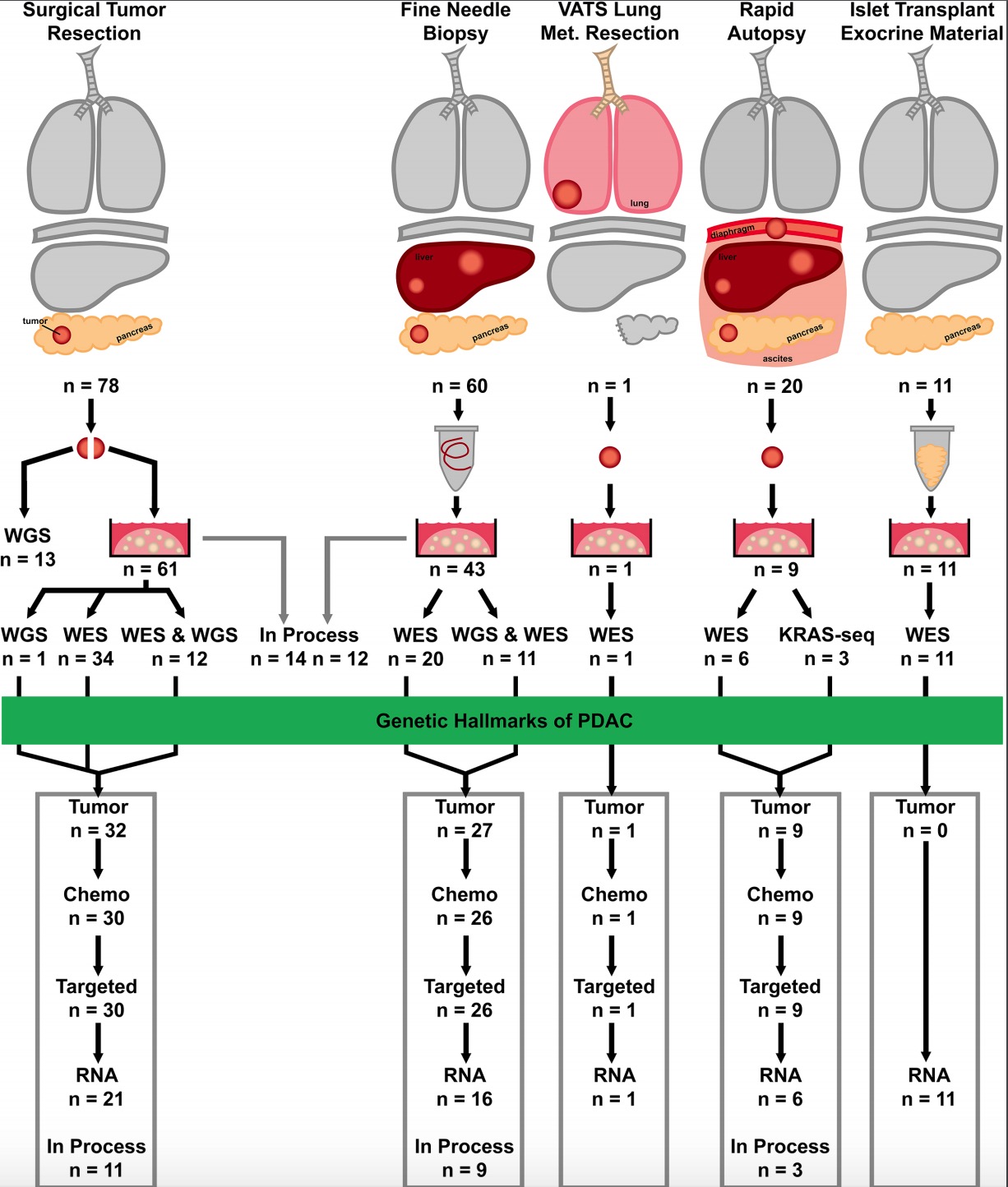
实验设计
本研究是从PDAC病人的肿瘤部分取样做3D培养:
总共是138个病人,里面取了 159个样品,包括 primary tumors (hT) metastases (hM)
- 78 个样本是specimens were isolated from surgical resections
- 60 个样本是fine needle biopsies of primary or metastatic lesions (hF)
- 20个样本是 metastatic disease following rapid autopsies
- 1个样本是a video-assisted thoracoscopic surgical (VATS) resection of a lung metastasis.
值得一提的是还有 11 human normal (hN) pancreatic ductal organoids were established from heathy normal pancreata,整体如下:
WGS数据部分
作者在这里对13个病人的原始肿瘤样品以及培养好的organoids样品配对测了WGS数据,主要是看somatic mutation的overlap情况,如下:
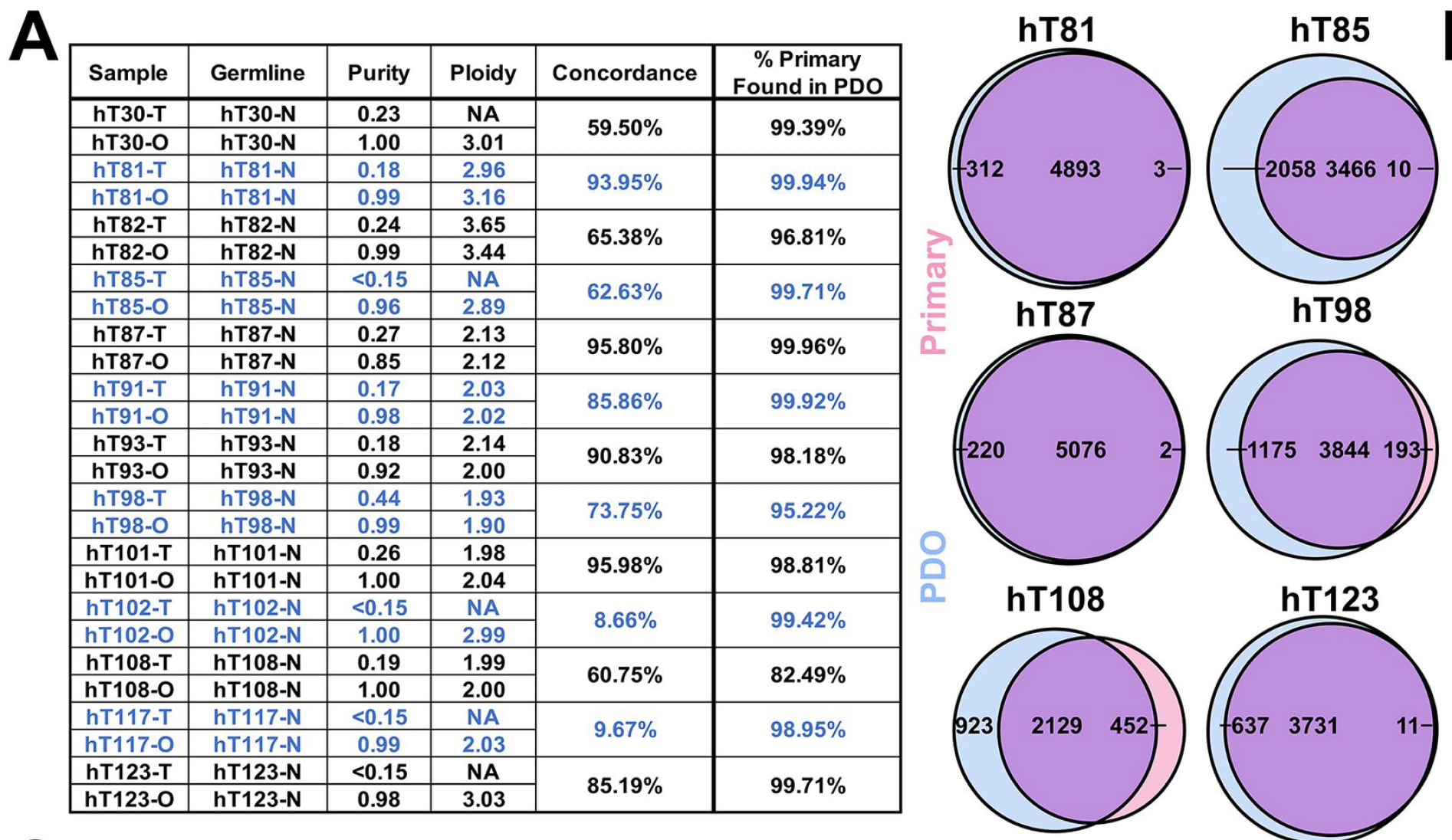
可以看到的是大部分病人的overlap情况非常棒,说明了 organoids可以比较完美的保留其来源的primary tumor的突变等肿瘤特征,虽然有两个overlap情况很差,但是作者找到了原因,是因为其primary tumor样品的肿瘤纯度太低了。WES数据部分
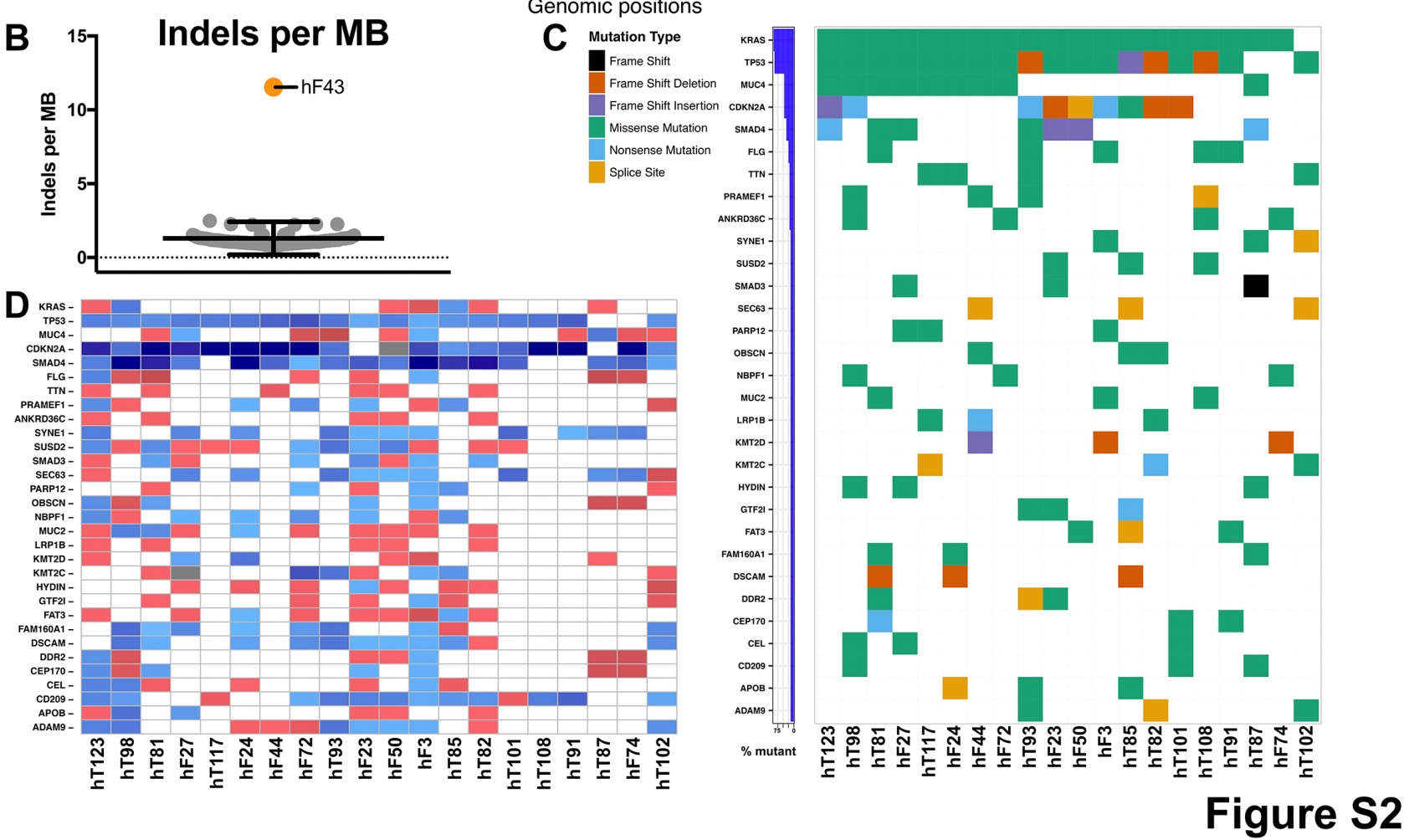
总共有66个PDAC的PDO都做了WES测序,但是作者没有公布测序数据也没有写清楚数据量,只是给了CNV分析结果,如下:

其中有两个PDO仍然是比较明显的2倍体,并不是癌症通常表现着的非整倍体。就是上图中的hT83和hF43,值得一提的是hF43的indel频率异常, 超出平均值的8倍,如下所示::
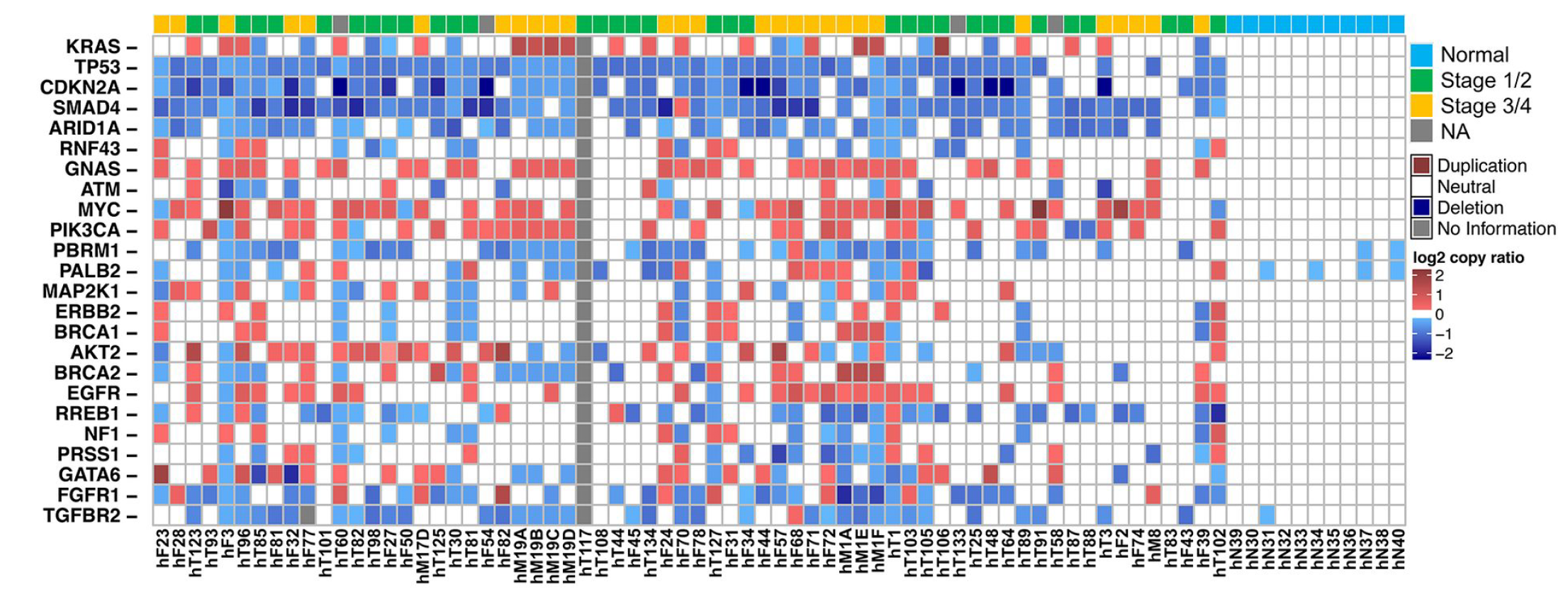
如果只看一些PADC相关的重要的基因的拷贝数变化,如图;
当然了,肿瘤外显子数据必不可少的的突变全景图如下:
RNA-seq数据部分
RNA sequencing was performed on 44 PDAC-confirmed PDOs and 11 hN organoid cultures.
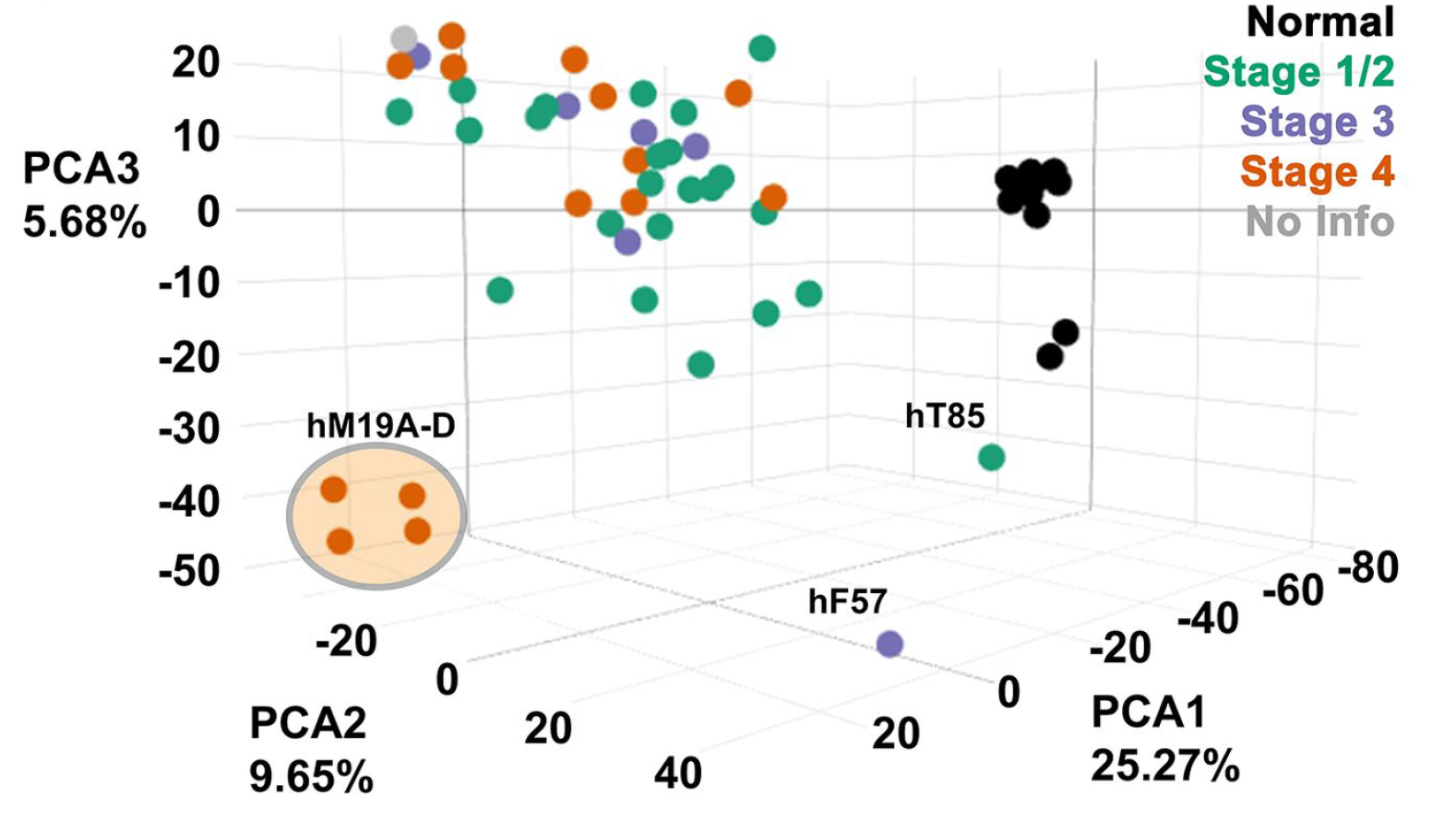
同样的,原始数据已经测序描述信息都没有,根据表达情况进行PCA可以比较明显的区分不同的PDO,如下:
表达矩阵最重要就是分类咯,首先看TCGA的那两个大类的marker基因的分类,如下;
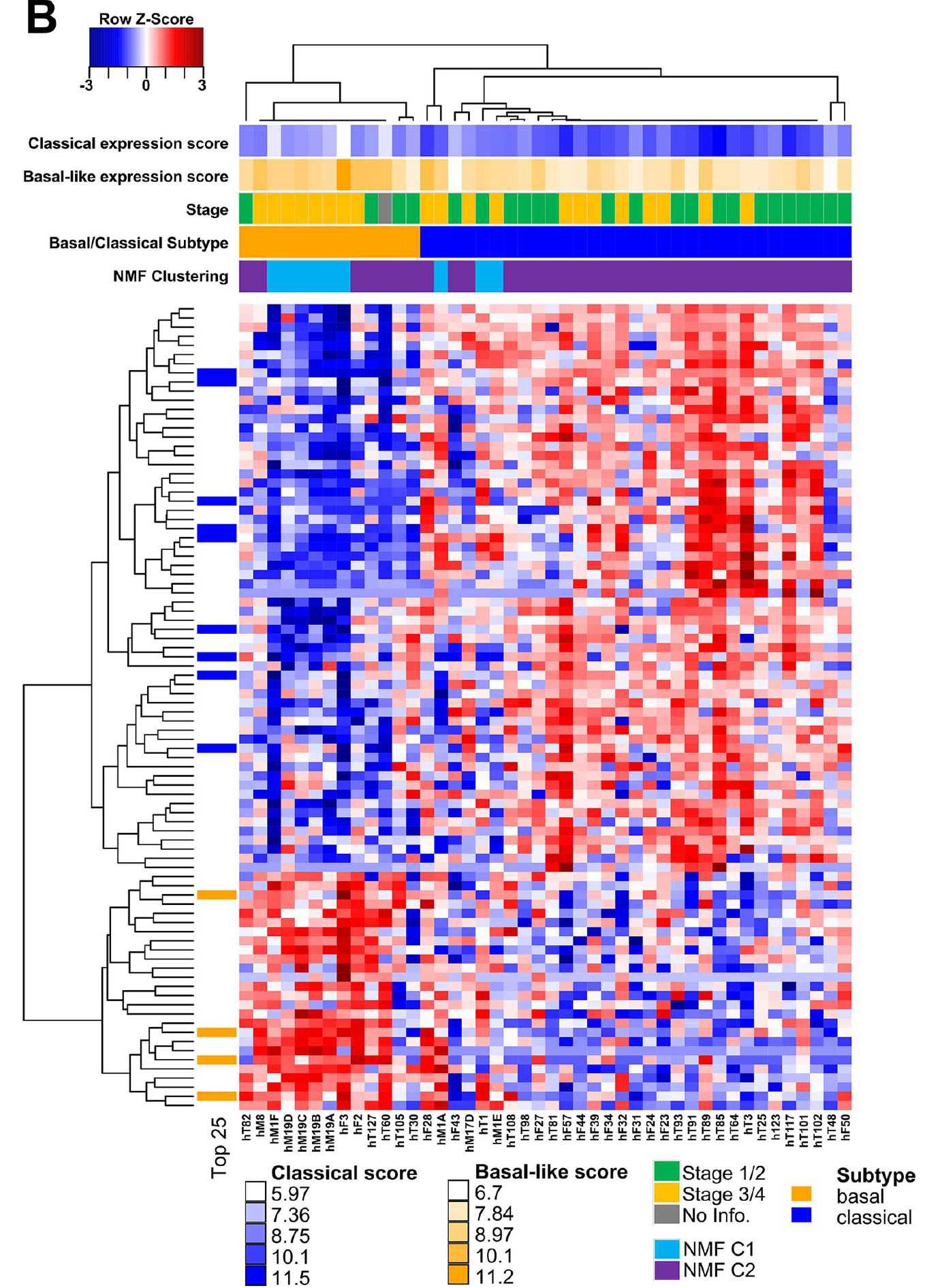
很清晰的分出来两类,然后看看NMF算法分类:

而且可以看到这两个分类虽然定义的基因集不一样,但是分类的overlap情况非常棒!!!
有了分类, 就可以找差异基因,定位到通路哦,GSEA或者富集分析均可:

作者还放了使用NMF进行聚类的细节问题,如下图:
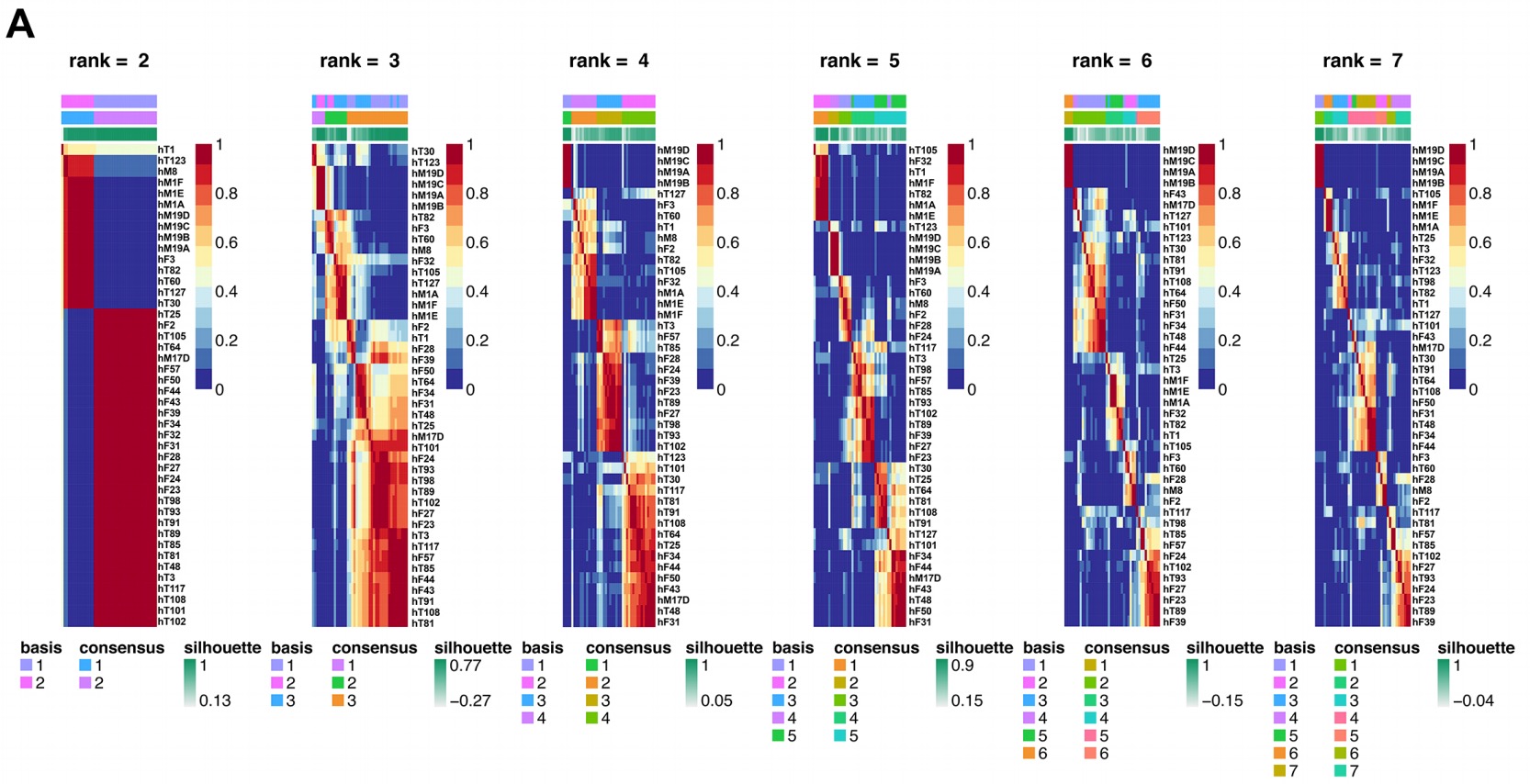
所以作者最后选取了2个分类作为最优分类,实际上异质性还是蛮大的,估计上单细胞可以看到更多的东西。药物处理部分
我本身对药物处理不是很感兴趣,就不深入解读了。
本文章利用了一些公共数据来辅佐其药物处理结果,包括: - tumors of PDAC patients on the COMPASS trial,来自于文章:Clin Cancer Res. 2018 Mar
- ICGC-CA转录组数据,95个接受新辅助化疗以及31个不处理的病人,来自于文章:JAMA Oncol. 2017 Jun