本次解读的文章是:Partitioning circadian transcription by SIRT6 leads to segregated control of cellular metabolism. Cell 2014 PMID: 25083875
全文基于设计完善的基因表达芯片实验数据展开。
最重要的结论是;We report that SIRT6 defines the circadian oscillation of a distinct group of hepatic genes, different from the ones under SIRT1 control.
组蛋白去乙酰化酶背景知识
1999年,科学家在最简单的真核生物酿酒酵母中发现了具有延长寿命的Sir2基因。后来,在小鼠等啮齿类动物中,Sir2的同源基因SIRT6同样被发现有此魔力(2012)。自此,SIRT6被科学界奉为经典的“长寿基因”,成为人类掌控衰老速度的一个热门突破口。
长寿基因SIRT6,是一种组蛋白去乙酰化酶,高表达该基因能明显延长雄性小鼠的寿命。SIRT6在代谢平衡,炎症,应激反应和基因组稳定性方面起到了至关重要的作用。SIRT6是DNA修复过程中的关键蛋白,细胞缺失SIRT6将不能修复多种类型的DNA损伤,包括DNA双链断裂(DSB)和碱基切除修复(BER)。SIRT6或者染色质重组蛋白SNF2H的缺失会影响重要的修复因子,如CtlP, Ku80, RPA, BRCA1和53BP1,的募集。
- 以色列古墓里安大学的Debra Toiber小组通过Nestin-Cre启动子构建了脑部特异性的SIRT6敲除小鼠。
- 麻省总医院(MGH)癌症中心报道Sirt6可通过抑制糖酵解途径抑制肿瘤细胞的生。Cell, 2012
- 芝加哥大学研究团队发现Sirt6可启动cox-2信号从而抑制AMPK信号,促进损伤皮肤细胞的存活和皮肤癌的发生发展。Cancer Res, 201
- 杭州师范大学鞠振宇教授团队对Sirt6基因敲除小鼠和正常小鼠的表达谱和组蛋白修饰谱进行分析,以探究Sirt6维持细胞干性的分子机制 Cell Stem Cell, 201600111-9)
其中,转录组芯片数据分析发现(Sirt6+/+)小鼠和(Sirt6-/-)小鼠的差异表达基因在Wnt信号通路(与衰老相关的重要通路)显著富集。诡异的是作者没有做重复,而且使用的是Affymetrix Mouse Genome 430 2.0 Array芯片数据。https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE73392
组蛋白去乙酰化酶调控基因表达、细胞周期、细胞分化,对于治疗癌症等疾病有着巨大潜力。目前在人体中共发现了18种HDAC,根据序列同源性不同被分为4种类型:
- 第Ⅰ类HDAC1~3,8,主要分布在细胞核内;
- 第Ⅱ类HDAC4~7,9,10;
- 第Ⅲ类为sirtuin蛋白家族,包括SIRT1~7
- 第Ⅳ类仅包含一个成员HDAC11.
去乙酰化酶(Sirt1-Sirt7)在哺乳动物中参与多个重要的细胞进程,如染色质沉默、细胞周期调控、细胞分化和代谢等。不同的去乙酰化酶调控相似的细胞进程,表明这些酶之间必然存在一种协调模式,然而去乙酰化酶家族成员间潜在的相互调控作用有待深入研究。
一般情况下,组蛋白的乙酰化有利于DNA与组蛋白八聚体的解离,核小体结构松弛,从而使各种转录因子和协同转录因子能与DNA结合位点特异性结合,激活基因的转录。
已知SIRT6针对H3K9和H3K56
表达芯片数据
都在 GSE57830 可以下载,使用的是 Affymetrix Mouse Gene 2.0 ST Array ,研究团队在6个昼夜周期时间点(0, 4, 8, 12, 16 and 20h)取样,分别是SIRT1,SIRT6突变以及WT小鼠,每个处理组合都有3个重复,所以是72个样本数据。
然后分析后把值得关注基因分成4组:
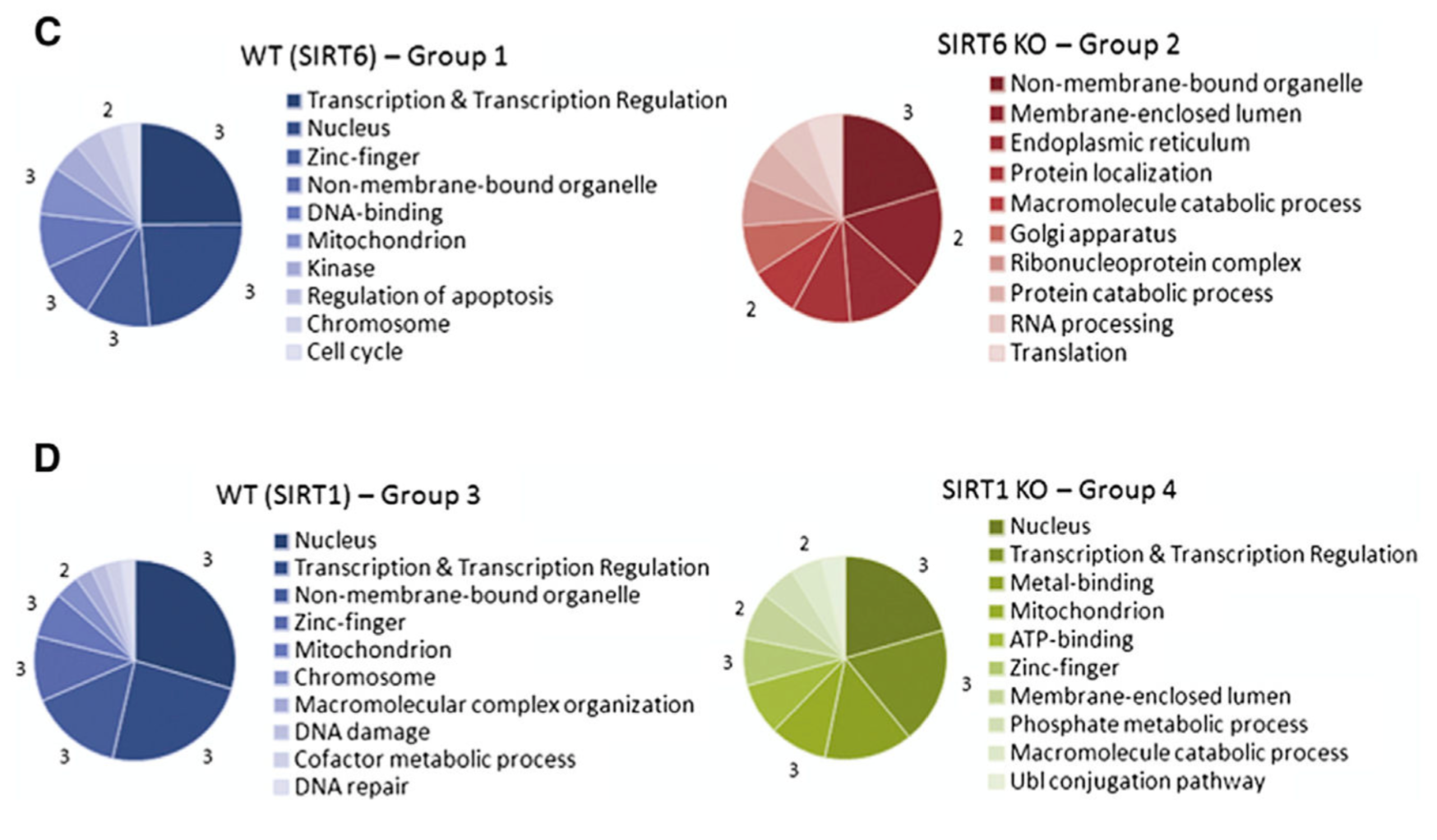
- group 1 represents genes that oscillate in WT (SIRT6) liver and whose oscillation is dampened/disrupted in SIRT6 knockout (KO) mice. (691 genes)
- Group 2 represents genes that oscillate in SIRT6 KO, but not in their corresponding WT littermates. (779 genes)
- Group 3 represents genes that oscillate in WT (SIRT1) liver and whose oscillation is dampened/disrupted in SIRT1 KO mice (SIRT1 KO). (703 genes)
- Group 4 respresents genes that oscillate in SIRT1 KO, but not in their corresponding WT littermates.(1126 genes)
有趣的是,作者整理并不是使用常规的差异基因寻找方法,因为分组太多,而是来自于 J Biol Rhythms. 2010 Oct;文章:JTK_CYCLE: an efficient nonparametric algorithm for detecting rhythmic components in genome-scale data sets 的统计方法。
p值小于0.01的基因如下图所示:

有了这些基因集,后续的各种数据库注释及生物学故事的完善就比较容易了。
依赖于SIRT1/6的共有周期节律相关基因有160个,如下所示

同样可以进行GO/KEGG数据库注释
Top ten GO terms (molecular function, biological process, and cellular component) were selected based on a 0.01 p value cutoff using DAVID.
使用R重复该数据处理流程
值得注意的是作者使用的JTK_Cycle统计方法,源代码的确是R写的,但是下载地址在:https://s3-us-west-2.amazonaws.com/oww-files-public/8/88/JTKversion3.zip
里面有测试数据,需要耗费一点时间来学。
3.1K Jul 13 2011 Example1_annot.txt
82K Jul 13 2011 Example1_data.txt
4.7K Jul 13 2011 Example2_annot.txt
43K Jul 13 2011 Example2_data.txt
309B Sep 15 2011 Example3_annot.txt
4.3K Sep 15 2011 Example3_data.txt
41K Sep 19 2015 Example4_data.txt
2.2K Sep 19 2015 Example5_data.txt
24K Sep 21 2015 JTK Users Guide (v3).docx
49K Sep 19 2015 JTK.Example1.rda
94K Sep 19 2015 JTK.Example1.txt
24K Sep 19 2015 JTK.Example2.rda
55K Sep 19 2015 JTK.Example2.txt
3.5K Sep 21 2015 JTK.Example3.rda
6.1K Sep 21 2015 JTK.Example3.txt
52K Sep 21 2015 JTK.Example4.txt
2.6K Sep 21 2015 JTK.Example5.txt
13K Sep 21 2015 JTK_CYCLEv3.1.R
886B Sep 21 2015 Run_JTK_CYCLE (Example1).R
1.1K Sep 21 2015 Run_JTK_CYCLE (Example2).R
956B Sep 21 2015 Run_JTK_CYCLE (Example3).R
1.9K Sep 21 2015 Run_JTK_CYCLE(Example4&5).R
然后本文章附带的数据,很容易通过GEO下载。
代码会同步更新在:https://github.com/jmzeng1314/GEO/tree/master/task3