时间序列表达矩阵揭示肿瘤转移的动态过程
肝癌很可怕,尤其是转移后,很多关于其转移前后对比的研究,但是缺乏中间过程数据,特别是转移临界点。
作者通过肝癌模型,在不同时间点取样做芯片转录组,试图分析 non-metastatic (or normal) and pre-metastatic (or critical) 这两种状态区别。顺利找到了临界点及其相关调控网络,而且还重点分析了其中一个网络的最重要的节点基因:CALML3
To discover early warning signals of pulmonary metastasis in HCC, we analysed time-series gene expression data in the spontaneous pulmonary metastasis mouse HCCLM3-RFP model with our novel dynamic network biomarker (DNB) method.
构建了 xenograft HCCLM3-RFP mice , 20只小鼠分成4组,即4个时间点(W2, W3, W4, W5)取样。
找差异基因
使用的是传统的T检验,对任意两组的组合找差异,最后合并成13,247 个差异基因列表,并且绘制热图如下:
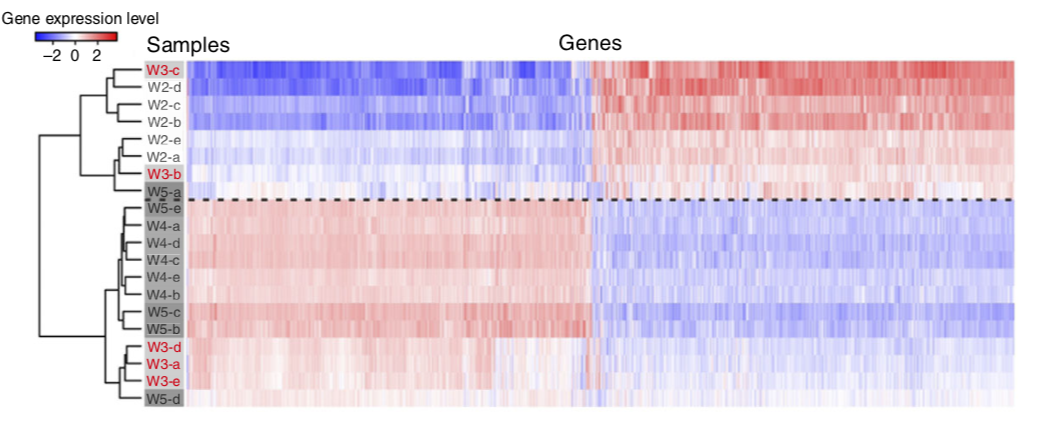
个人觉得这个分析有点悬,有时间的朋友可以下载他们公布的表达矩阵试试看,怎么样可以做成这样的热图。
时间序列基因集
使用的是R包Mfuzz把差异分析得到的13,247 个统计学显著差异基因根据表达模式分成了6组,如下:

这6组基因集特性是:
- Genes in Clusters 1 and 2 were continuously and monotonically downregulated and upregulated from the second to fourth weeks after orthotropic implantation, respectively.
- Genes in Clusters 3 and 4 were significantly downregulated and upregulated only during the second and third weeks, respectively,
- whereas genes in Clusters 5 and 6 were significantly downregulated and upregulated only during the third and fourth weeks, respectively.
所以作者合理的得出第3周这个时间点是 肿瘤转移的关键时刻。
而且顺理成章的可以对这些基因集做各种数据库的功能富集分析

临界点理论

关于 dynamical network biomarkers
同样的一个方法, dynamical network biomarkers ,使用了十几次 ,都是在探索各种生命活动的临界值。
而且只需要表达矩阵,性价比很高

挑出一个biomarker
这个时候需要理解作者的那个使用了十几次的 dynamical network biomarkers方法,才能理解为什么能定位到CALML3这个基因来做下游分析。