为了分析不同类型、组织起源肿瘤的共性、差异以及新课题。TCGA于2012年10月26日-27日在圣克鲁兹,加州举行的会议中发起了泛癌计划。参考:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6000284/ 为此我也录制了系列视频教程在:TCGA知识图谱视频教程(B站和YouTube直达)
发表在CNS正刊的泛癌研究不多,除了前面提到的 Nature. 2013 Oct 和 Cell. 2014 Aug ,接下来我们要介绍的是Nature. 2014 Jan , 引用也是近2000了,题目是:Discovery and saturation analysis of cancer genes across 21 tumour types. 主要关注的仍然是突变信息,仍然是寻找有统计学显著的癌症相关基因,创新点是分析了不同测序深度对找到的癌症相关基因的影响。
一个不容忽视的现象是TCGA计划的肿瘤外显子数据得到的突变位点突变频率通常是20%不到,而且非常多的肿瘤样本数据里面找不到任何重要基因的突变现象,
病人数量和癌症种类
这里研究者收集了TCGA计划的12种癌症和其它计划的14种癌症的4742个病人的N-T外显子测序数据分析结果,各个癌症的样本量及突变数量如下:

分析癌症相关基因
这里使用 MutSig软件,首先分癌症内部找癌症相关基因,发现只有22个基因在3个以上的癌症出现,能同时出现在3个癌症的也就10个基因而已。
合并癌症可以找到114个癌症相关基因,其中有30个是区分癌症的时候找不到的,区分癌症可以找到224个基因,其中140个是合并后找不到的,它们共有的基因就84个,这个时候应该是用一个韦恩图展示。
这总共的254个基因,就是作者宣传的 Cancer5000 set ,然后研究者做了统计学校正,缩小到219个基因,就是Cancer5000-S (for ‘stringent’) genes.
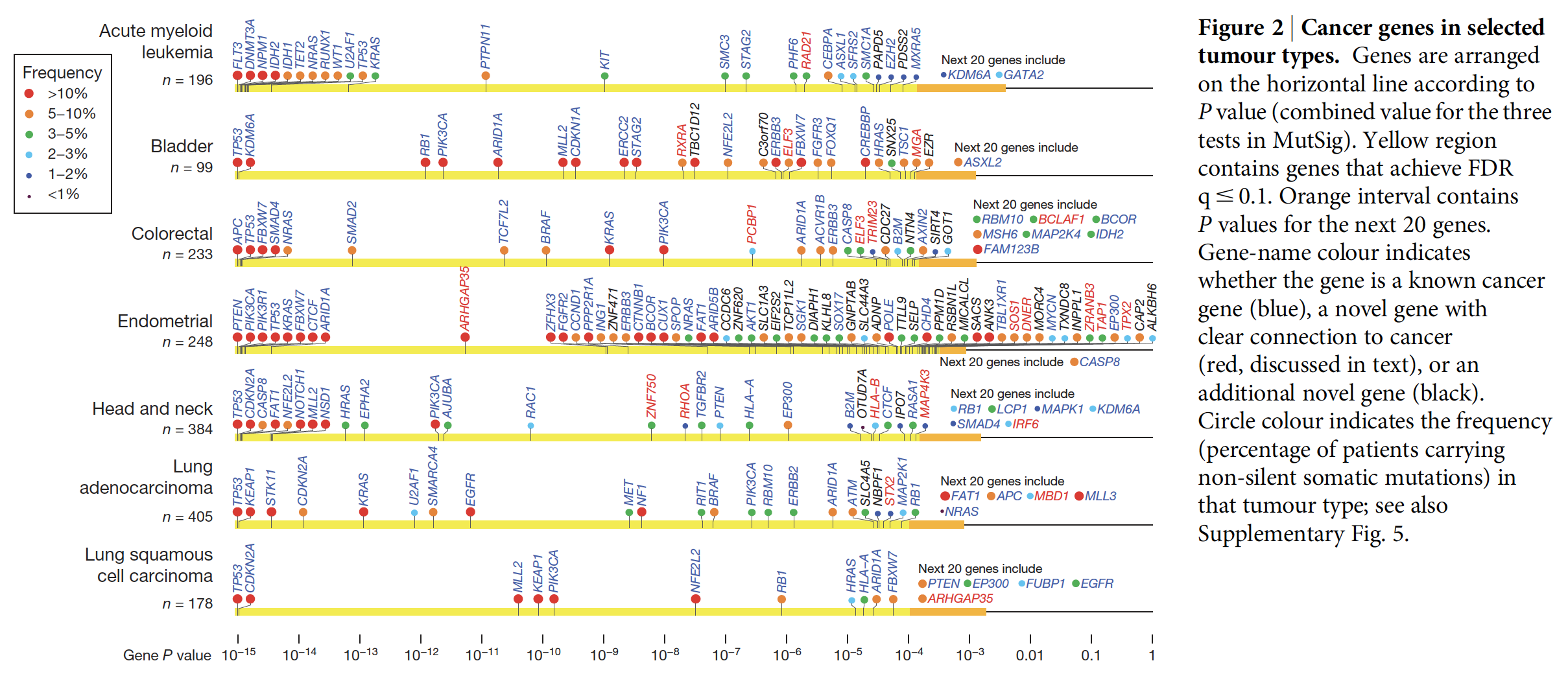
跟CGC比较
COSMIC数据库维护的CGC基因集,Cancer Gene Census - COSMIC 在这篇文章里面采用的是V65,也就130个基因,其中82个都在作者的 Cancer5000 set 里面。
有趣的是在 Cancer5000-S 这个219个基因里面,有81个不在CGC也不在公开发表的文章里面,作者认为是 ‘novel’ genes 进行细致的探索。即使这81个里面有40个可能是假阳性,也至少还是41是有意义的。
然后作者就一个个描述了这些基因,可怕!!!
测序饱和分析和功效检验
主要是对测序数据进行抽样,看看各个癌症需要收集多少个病人进行测序,才算是足够。
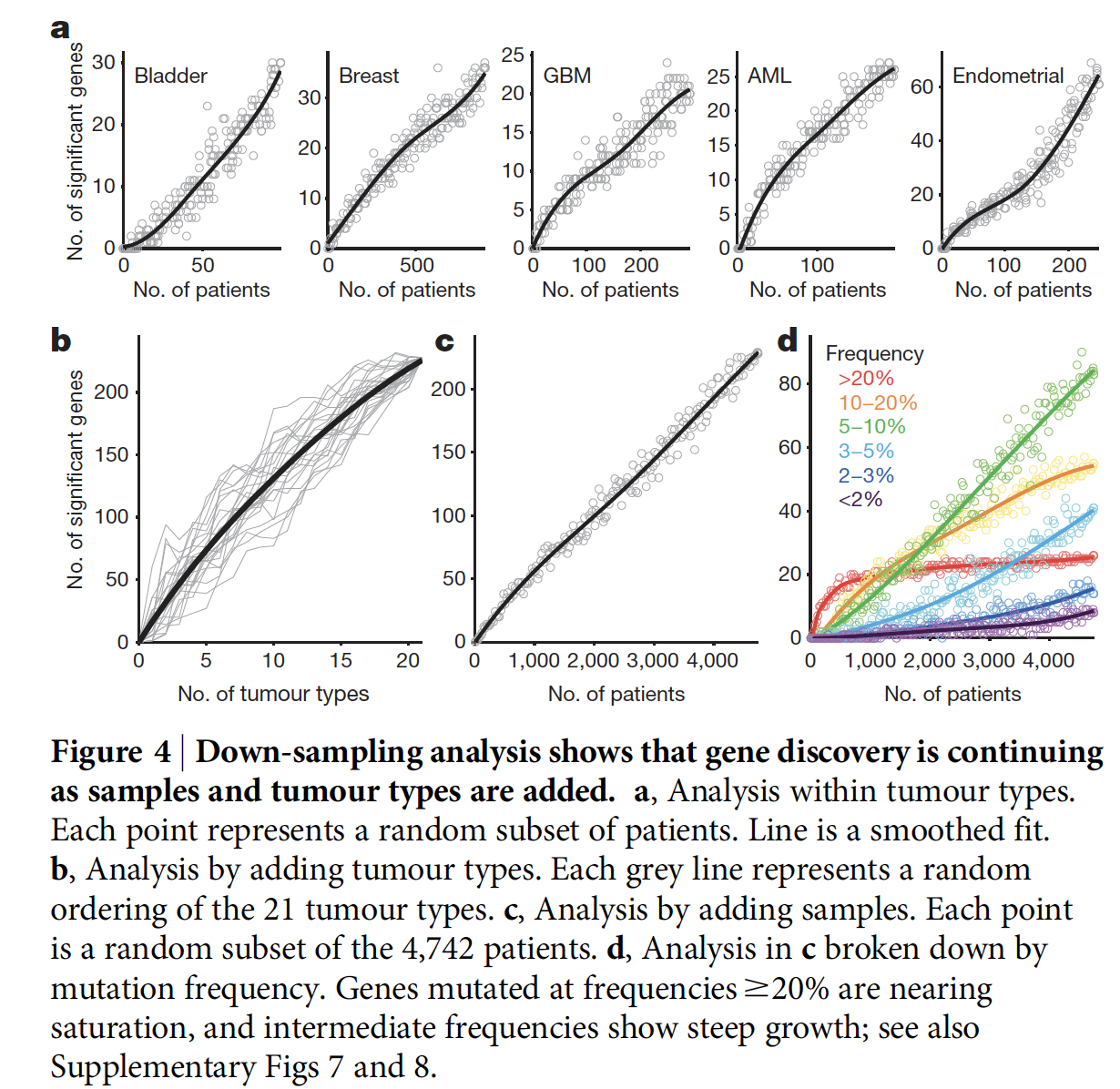
对部分VAF值非常低的突变来说,样本量大于10万(50种癌症,每个2000样本),才有可能全部覆盖。
Down-sampling of the full Cancer5000-S procedure (Fig. 4c): we repeated our procedure of constructing the Cancer5000-S list by applying the stringent procedure of correction for the approximately 400,000 hypothesis (18,388 genes 322 analyses), and computed how many genes remained significant at each smaller set size.
后记
发在CNS正刊的研究,分析的点都很多,而且涉及到的统计学算法比较难以理解。
本文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html