来自于文献:Identifying and Targeting Sporadic Oncogenic Genetic Aberrations in Mouse Models of Triple Negative Breast Cancer - BioProject 的数据:
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA398328 点击 RunSelector. Send results to Run selector 就可以下载那两个测序数据记录文件啦。
根据上面的两个文件就可以去下载完整测序数据,并且走完了WES和RNA-seq流程,原始数据fq的gz压缩包共1Tb大小,全部分析估计耗费10T空间,需要服务器。
数据分析流程
根据作者给出的ID号
下载:
cat srr.list |while read id;do (nohup ~/biosoft/sratoolkit/sratoolkit.2.8.2-1-centos_linux64/bin/prefetch $id -X 100G & );done
下载得到的sra文件,这里就不演示数据分析完整流程,大家可以自行去https://www.jianshu.com/p/a84cd44bac67 搜索代码,让我们看看这篇文章讲了什么生物学故事吧。
背景介绍
TNBC是最恶性的乳腺癌亚群,治疗手段有限,通常表现为TP53基因功能的变异,超80%的TNBC患者有着TP53基因点突变。
这里,很容易下载TCGA的somatic突变maf文件,自己导入R语言,根据PAM50分子分型分组绘制突变全景图,如下:
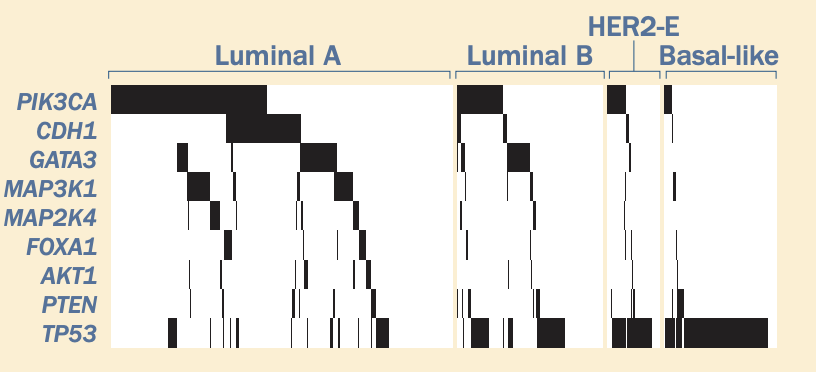
可以看到TP53基因的确在basal-like亚型超高频率突变,而basal-like亚型与TNBC大幅度重合,见:https://mp.weixin.qq.com/s/NP5LKEiyBq6M5hKGWpnxcA
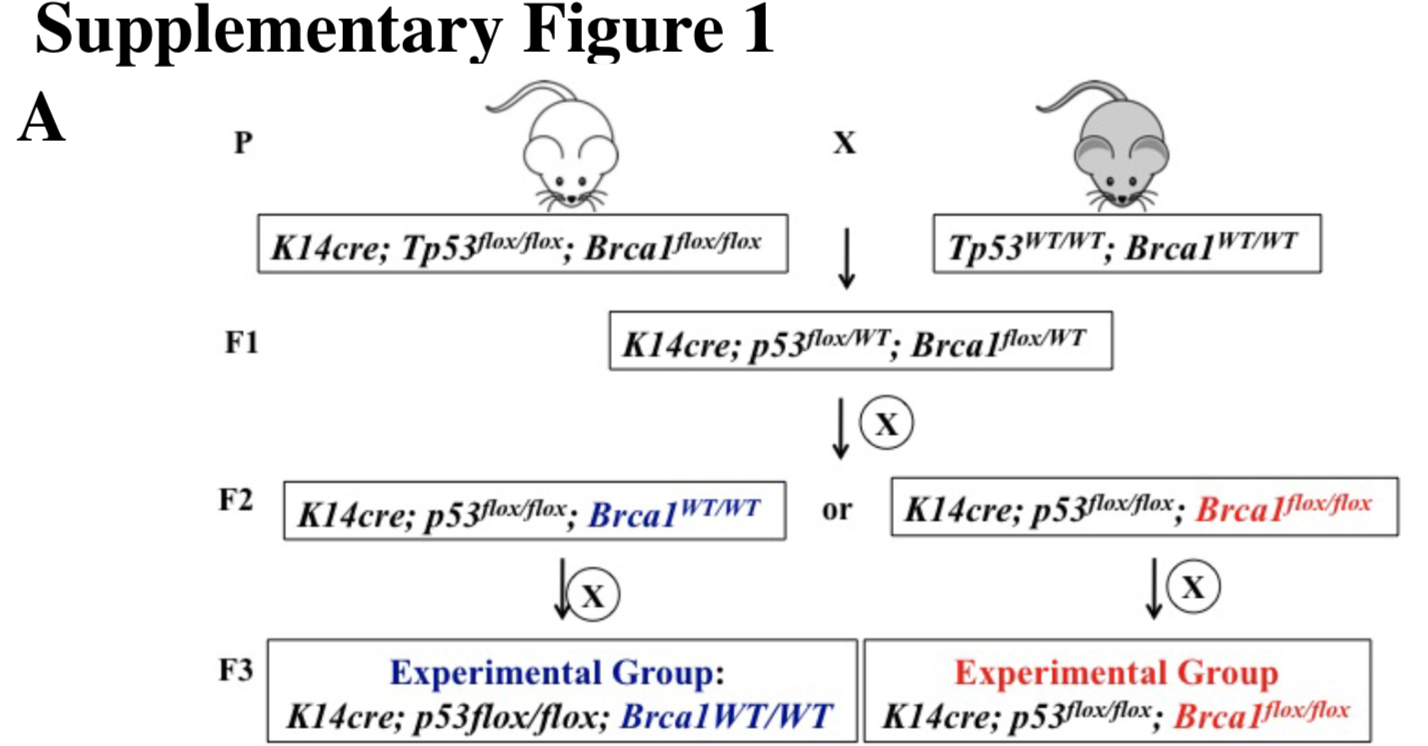
构建TNBC的基因工程小鼠
研究团队成功制备了Trp53缺失或Brca1基因缺陷的基因工程小鼠模型,并且通过WES和RNA-seq来说明他们的小鼠模型可以比较好的模拟人类的TNBC病人。
制备过程如下:
根据IHC结果可以看到PR并不都是阴性,不过并不影响这些小鼠大多属于TNBC,而且根据分子分型,可以看到大多是basal-like:
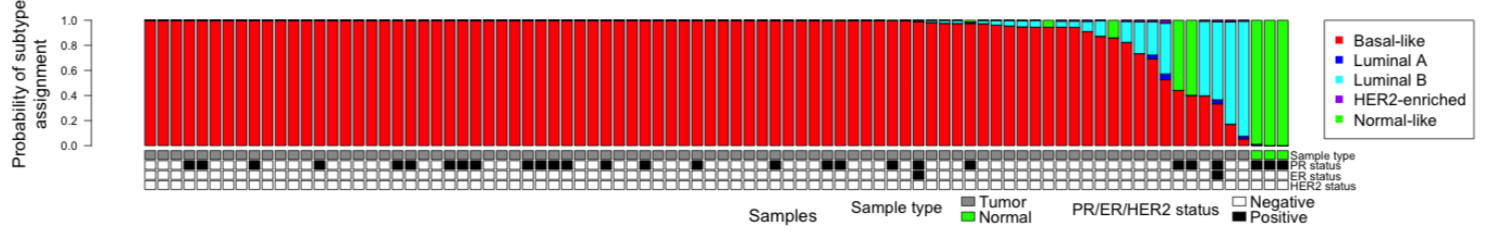
所以作者的重点是他们的实验小鼠模型的构建方法,可以比较好的模拟人类的TNBC病人。
Therefore, both immunohistological analysis and transcriptional profiling indicated that these mouse tumors, developed on a Tp53-deficient background with or without Brca1, provide reasonable models for human TNBC.
到这里,作者的故事已经完成一大半啦!点突变和拷贝数变异的异质性
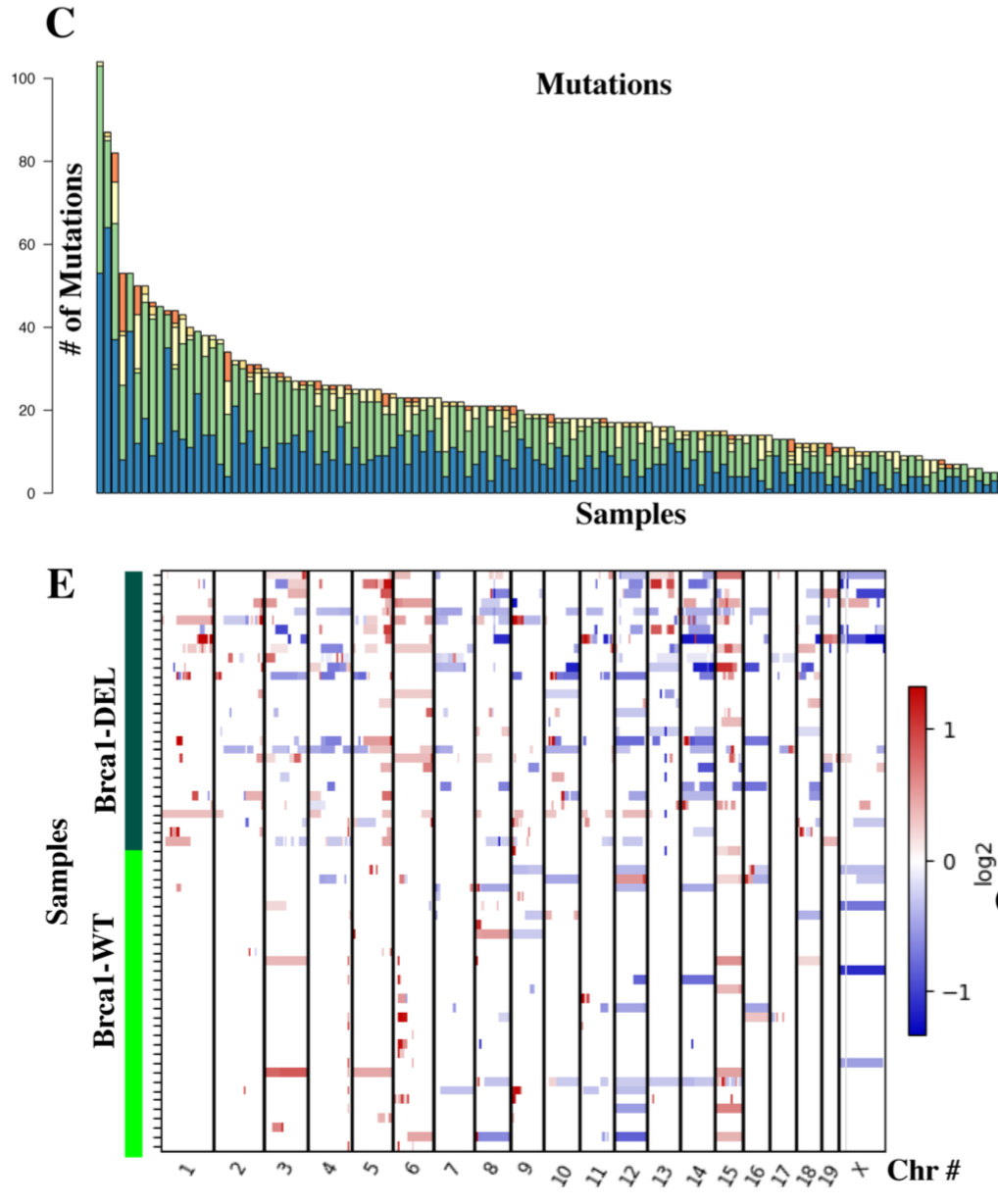
走完肿瘤外显子的标准分析流程,可以拿到点突变和拷贝数变异文件,简单绘图可视化可以看到,小鼠的somatic突变数量并不多,才 averaging 30 somatic mutations per tumor with 17 non-synonymous mutations , 比起TCGA数据库的BRCA的TNBC来说(average 127 somatic mutations per tumor with 107 being non-synonymous ) 值得思考,是不是小鼠模型有不完美的地方呢?
而且可以看到,是否有BRCA1基因突变,会极大的影响TNBC小鼠的拷贝数变异程度。
如下:
这里,可以看到小鼠之间的肿瘤异质性是非常大的, 每个小鼠的拷贝数变异模式都不尽相同。突变和表达的关系
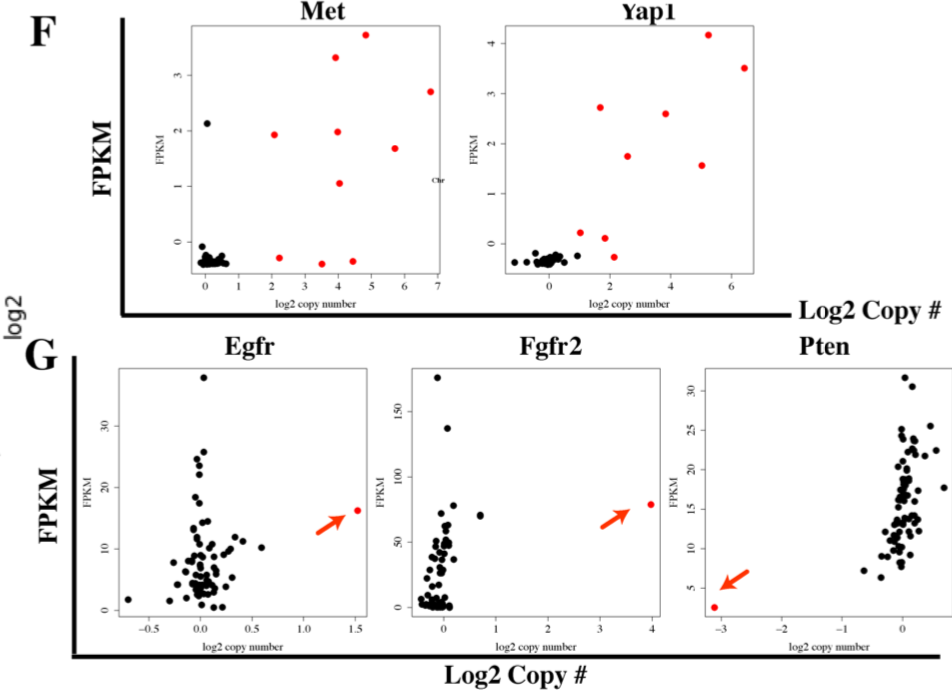
分析上面步骤的拷贝数变异结果,发现recurrent Met and Yap amplification in 20% and 22% of primary mouse tumors ,所以就继续探索CNV和RNA表达量的关系,如下;
融合基因情况
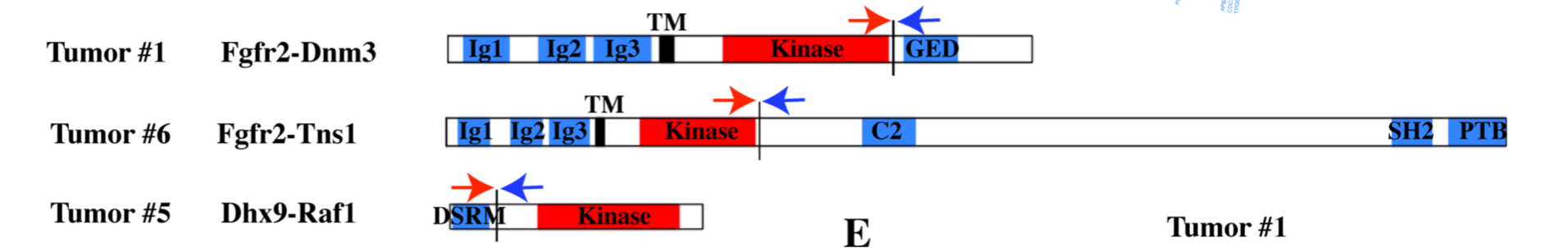
有趣的是研究者把RNA-seq数据的融合基因事件当做是重点来描述,使用的是FusionCatcher软件,而且还做了实验验证。
公共数据库挖掘
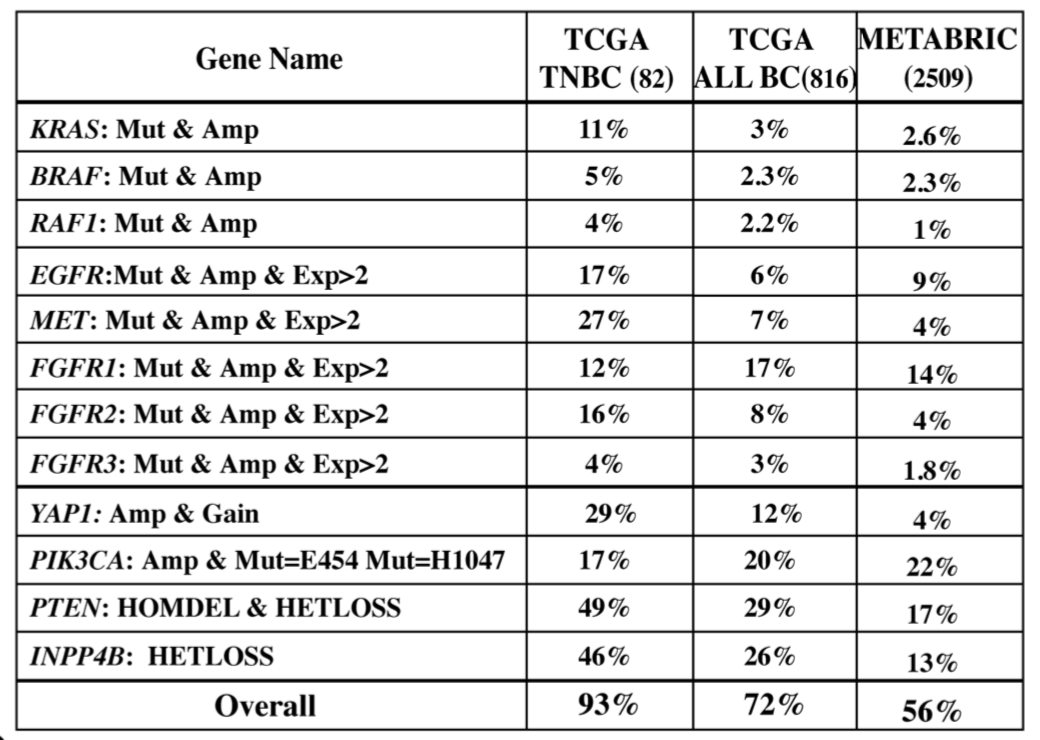
主要是比较一些能激活 MAPK and/or PI3K 通路重点事件在不同数据集的发生频率,如下:
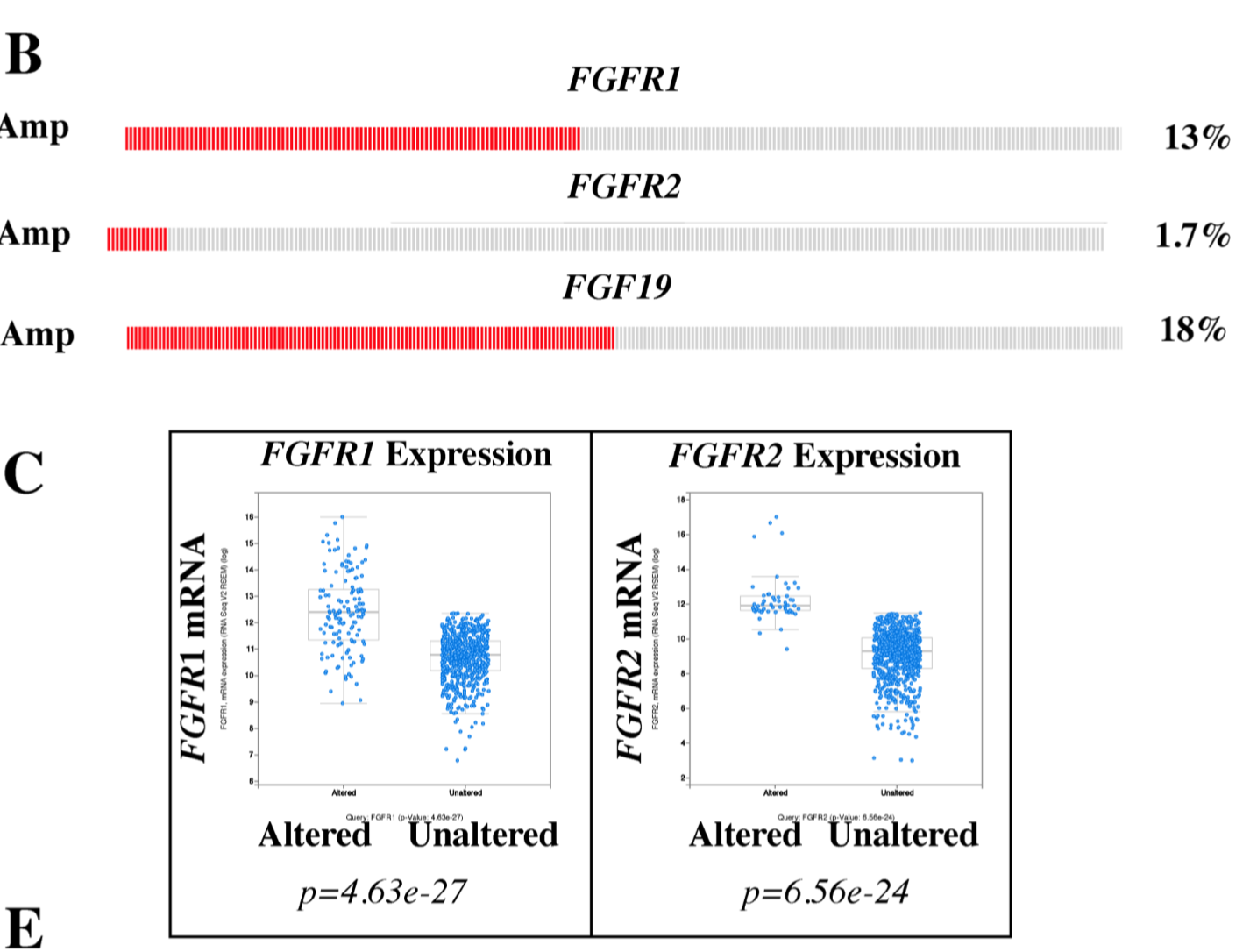
关于FGFR家族
作者分析自己团队构建的基因工程小鼠带有Tp53 deletion, with or without Brca1 deletion可以模拟TNBC病人,但是非常的diverse,共性在于 MAPK and/or PI3K 通路的激活,所以后续研究集中于修改药物测试,还有通路基因的探索上面。