WGCNA分析大家都不陌生了,我在生信技能树多次写教程分享WGCNA的实战细节:
- 一文看懂WGCNA 分析(2019更新版)
- 通过WGCNA作者的测试数据来学习
- 重复一篇WGCNA分析的文章(代码版)
- 重复一篇WGCNA分析的文章(解读版)(逆向收费读文献2019-19)
- 关键问题答疑:WGCNA的输入矩阵到底是什么格式
那些教程都是针对传统的bulk转录组测序的表达矩阵,其实单细胞转录组也是拿到表达矩阵,只不过是有一些特性,比如非常多的0值等等。那么有没有这样的研究尝试把WGCNA融入单细胞转录组数据分析呢?
答案是有的,Posted March 04, 2019. 丢在预印本的文章,题目是:Single-Cell RNA Sequencing Reveals Regulatory Mechanism for Trophoblast Cell-Fate Divergence in Human Peri-Implantation Embryo 就这样做了,让我们一起来看看吧。背景
To obtain transcriptomic profiles of human trophoblast cells during peri-implantation development, we harvested single cells from 19 embryos from day 6 to day 10, complement with 25 endometrial cells. Transcriptomes from 614 single cells were successfully profiled, with 0.7 million uniquely mapped reads and 24,011 detected transcripts per cell on average.数据都是在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE125616
主要样品是人类着床前胚胎的 Trophoblasts 进行单细胞转录组测序,其中516 embryonic cells 可以分成476 TE-, 14 EPI-and 26 PE-lineage cells. 最后的分析重点是 476 individual trophoblast cells isolated from 19 human embryos - cells of epiblast (EPI),
- primitive endoderm q (PE)
- trophectoderm (TE)
当然了,还有少量的endometrial cells,第一主成分就可以区分开来它们,如下:
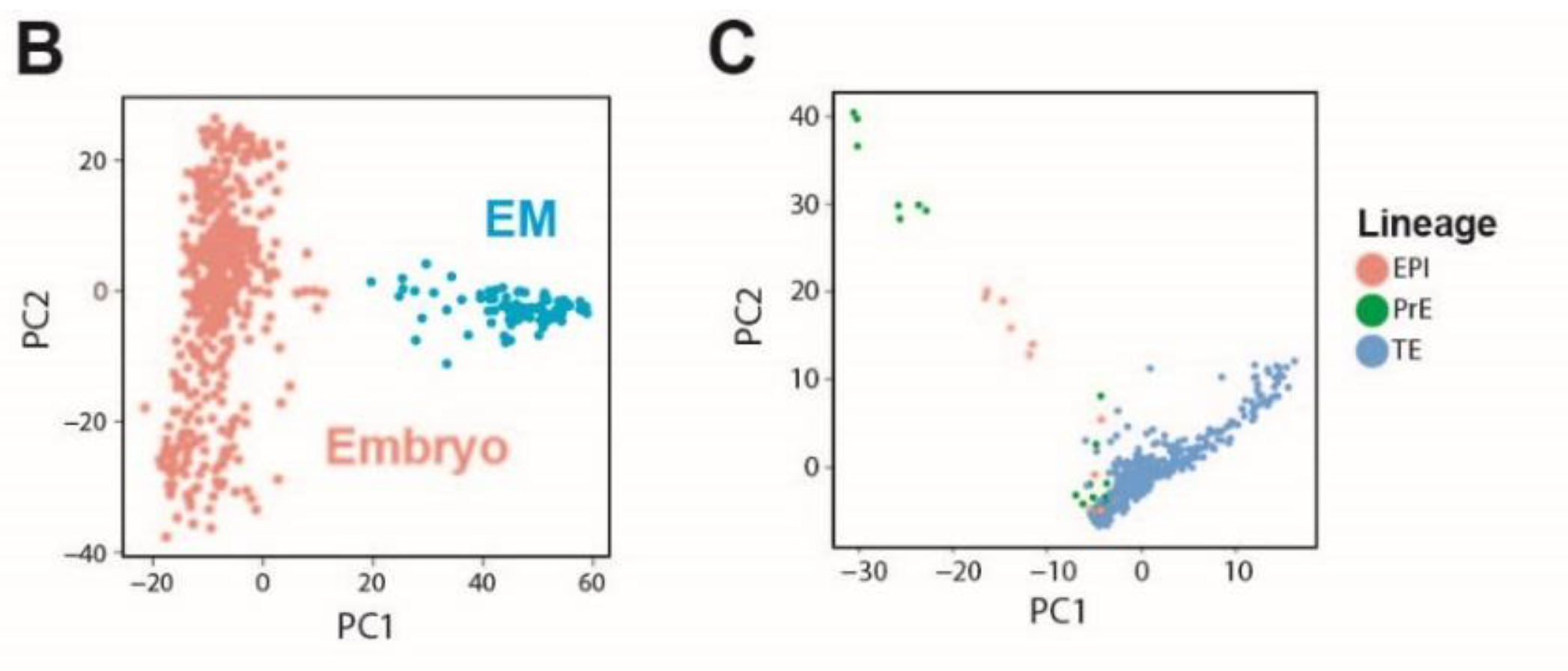
Embryonic cells were assigned into three lineages, namely TE, EPI and PE, based on their expression of 300 previous identified lineage marker genes. 需要相关生物学知识。
其中时间这个属性也是在PCA上面反映到:
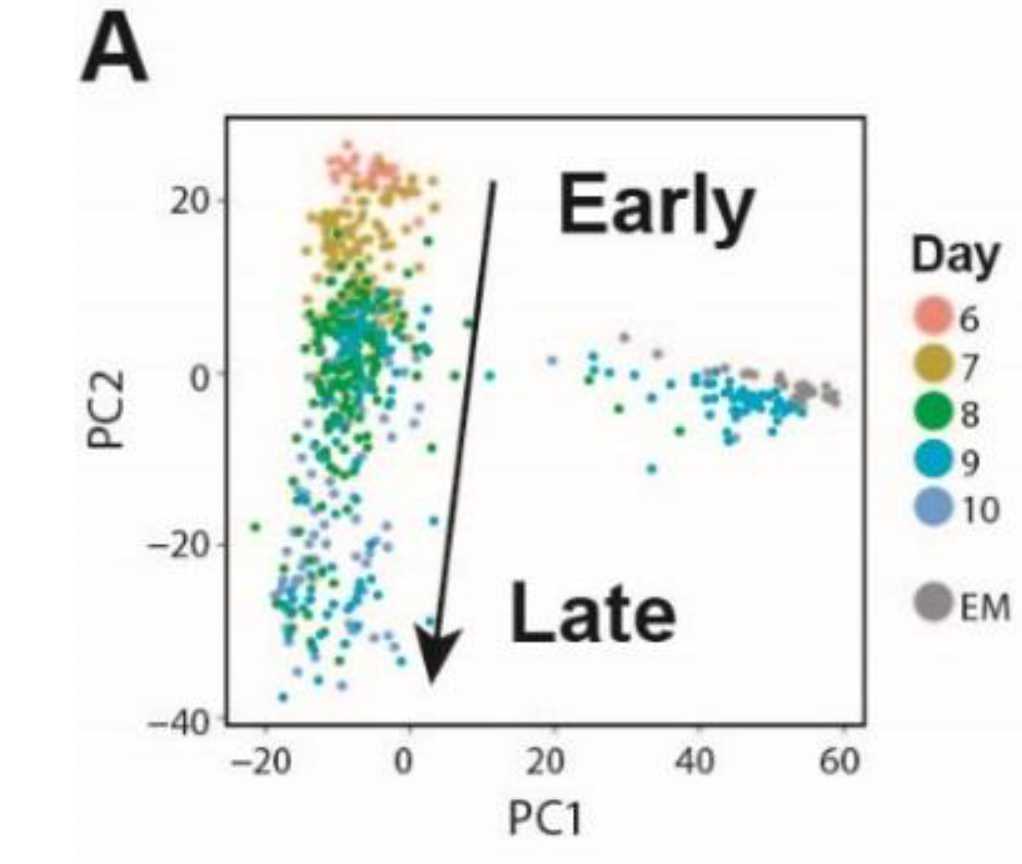
不管是时间这个属性天然对单细胞分组,还是整体的表达矩阵进入单细胞数据分析流程后分组, 都是可以看基因表达量情况的小提琴图等等。分析其实仍然是我们一直讲解的R包及基础流程,分别是: scater,monocle,Seurat,scran,M3Drop 需要熟练掌握它们的对象,:一些单细胞转录组R包的对象 流程也大同小异: - step1: 创建对象
- step2: 质量控制
- step3: 表达量的标准化和归一化
- step4: 去除干扰因素(多个样本整合)
- step5: 判断重要的基因
- step6: 多种降维算法
- step7: 可视化降维结果
- step8: 多种聚类算法
- step9: 聚类后找每个细胞亚群的标志基因
- step10: 继续分类
WGCNA步骤
To systematically investigate the genetic program dynamics, we performed Weighted Gene Co-expression Network Analysis (WGCNA) on 2,464 genes that were variably expressed in trophoblast cells between different developmental stages.
WGCNA identified eight gene modules, each of which contains a set of genes that tend to be coexpressed at a certain development stage!
可以看到WGCAN其实大家需要注意的是挑选基因,然后判断模块,最后关联起来性状即可!

研究者感兴趣的生物学组别
其实是:
- cytotrophoblast (CT),
- extravillous cytotrophoblast (EVT)
- syncytiotrophoblast (ST)
所以才会有如下图表:

让我意外的是,文章里面仅仅是提到了 Seurat 流程,没有monocle,但是却有lineage分析 !其实这个小鼠发育研究,跟我前面的视频课程非常类似,可以作为一个练习题,考核一下大家! - scRNA小鼠发育Smartseq2流程—前言及上游介绍
- 根据表达矩阵进行分群-1
- 根据表达矩阵进行分群-2
- 标记基因可视化
- 差异分析及功能注释(上)
- 差异分析及功能注释(下)
- 发育谱系推断及可视化
- 不同谱系的差异基因分类注释