实际上写完了这个全网最好的差异分析代码:免费的数据分析付费的成品代码 我就可以收工了,但是永远不能低估粉丝的疑惑数量,任何一个细节都会被拿出来剖析。
比如代码里面我挑选了top1000的sd基因绘制热图,然后就可以分辨出来自己处理的数据集里面的样本分组是否合理啦。其实这个热图差不多等价于PCA分析的图,被我称为表达矩阵下游分析标准3图!详见:你确定你的差异基因找对了吗? ,就是下面的3张图:
- 左边的热图,说明我们实验的两个分组,normal和npc的很多基因表达量是有明显差异的
- 中间的PCA图,说明我们的normal和npc两个分组非常明显的差异
- 右边的层次聚类也是如此,说明我们的normal和npc两个分组非常明显的差异
PS:如果你的转录组实验分析报告没有这三张图,就把我们生信技能树的这篇教程甩在他脸上,让他瞧瞧,学习下转录组数据分析。
为什么挑选top1000的sd基因绘制热图
我这个热图是为了说明本分组是否合理,就是看样本的距离,这个时候你如果需要理解距离,那么你需要学习非常多细节知识。不仅仅是一个函数那么简单:
- r 语言中使用 dist ( x, method = “ euclidean ”, diag = FALSE, upper = FALSE, p = 2 ) 来计算距离。其中x是样本矩阵或者数据框。method 表示计算哪种距离。method 的取值有:
- euclidean 欧几里德距离,就是平方再开方。
- maximum 切比雪夫距离
- manhattan 绝对值距离
- canberra Lance 距离
- minkowski 明科夫斯基距离,使用时要指定p值
- binary 定性变量距离.
- r 语言中使用 hclust (d, method = “complete”, members=NULL) 来进行层次聚类。其中 d 为距离矩阵。method 表示类的合并方法,有:
- single 最短距离法
- complete 最长距离法
- median 中间距离法
- mcquitty 相似法
- average 类平均法
- centroid 重心法
- ward 离差平方和法
- r 语言中主要使用 kmeans(x, centers, iter.max = 10, nstart = 1,algorithm =c(“Hartigan-Wong”, “Lloyd”,”Forgy”, “MacQueen”))来进行聚类。
- centers 是初始类的个数或者初始类的中心
- iter.max 是最大迭代次数
- nstart 是当 centers 是数字的时候,随机集合的个数
- algorithm 是算法,默认是第一个。
也就是说,看起来非常简单的3张图,背后是几十年的统计学知识的基础建设。
当然了,也不要气馁哦,反正你只需要会看图就好!再次强调:你确定你的差异基因找对了吗? 里面的3张图:
- 左边的热图,说明我们实验的两个分组,normal和npc的很多基因表达量是有明显差异的
- 中间的PCA图,说明我们的normal和npc两个分组非常明显的差异
- 右边的层次聚类也是如此,说明我们的normal和npc两个分组非常明显的差异
为什么选择top1000的sd基因绘制热图其实就是个人爱好,你可以探索top500,1000,2000,5000是否有区别。
比较不同的top基因聚类的差异
同样的是一个表达矩阵和分组,如下:
> dat[1:4,1:4]
GSM312896 GSM312897 GSM312898 GSM312899
ZZZ3 8.286337 8.427789 9.315658 8.601464
ZZEF1 8.097163 8.419503 8.843223 8.963619
ZYX 8.187251 7.919804 8.082291 7.094361
ZYG11B 8.718149 8.195136 8.478548 9.054829
> dim(dat)
[1] 18938 41
> table(group_list)
group_list
normal npc
10 31
然后检查不同top基因集的层次聚类情况
library(pheatmap)
cg=names(tail(sort(apply(dat,1,sd)),1000))
p1=pheatmap(dat[cg,])
cg=names(tail(sort(apply(dat,1,sd)),500))
p2=pheatmap(dat[cg,])
cg=names(tail(sort(apply(dat,1,sd)),2000))
p3=pheatmap(dat[cg,])
cg=names(tail(sort(apply(dat,1,sd)),5000))
p4=pheatmap(dat[cg,])
tmp=data.frame(top1000=cutree(p1$tree_col,2),
top500=cutree(p2$tree_col,2),
top2000=cutree(p3$tree_col,2),
top5000= cutree(p4$tree_col,2),
group_list=group_list)
这个时候,你会发现,好像不一样,我修改层次聚类的类别数量
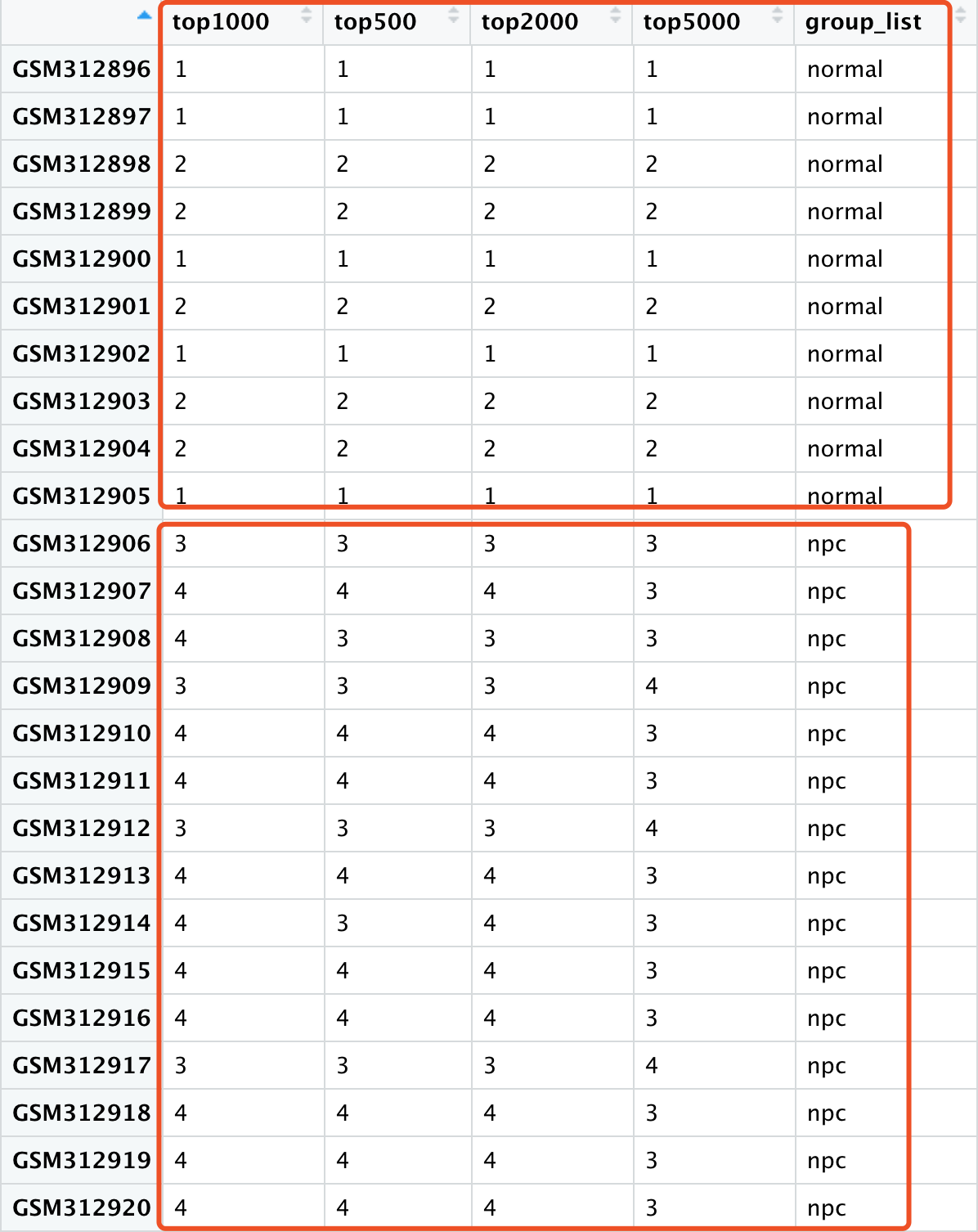
tmp=data.frame(top1000=cutree(p1$tree_col,4),
top500=cutree(p2$tree_col,4),
top2000=cutree(p3$tree_col,4),
top5000= cutree(p4$tree_col,4),
group_list=group_list)
然后你会发现,不管是top多少个基因,如果分成4类,那么1,2类都是normal,3,4类都是NPC,所以效果都挺好的。
其实基础知识点都在生信技能树了
- 生信技能树的2019年终总结 这里面记录着我博士期间抽空做的一点教程分享事业
- 2020学习主旋律,B站74小时免费教学视频为你领路 这里面记录着我为生物信息学教学行业做的一点贡献