2013年,《Nature Methods》杂志将单细胞测序列为年度技术。
2013年,《Science》杂志将单细胞测序列为年度最值得关注的六大领域榜首。
与此同时,随着测序成本的下降,越来越多的单细胞的文章发表在顶级的期刊上,这些都表明单细胞测序已经成为科研的热点。 但是并不意味着冲向这个领域就有CNS,哪怕花了几百万经费测大量单细胞转录组数据。尤其是某些癌症领域,已经有了几十个单细胞相关研究发表,如果确实从实验设计和数据角度来看都平淡无奇,那么很可能大量单细胞的转录组数据也不是很好发出去。
从摘要到预印本
比如,有一个研究团队(BGI-Shenzhen)做的是乳腺癌领域的三阴性乳腺癌的单细胞免疫微环境研究:
首先是2018摘要:https://cancerres.aacrjournals.org/content/78/13_Supplement/1763A 上面公布了研究者测了8个TNBC病人的七千多个单细胞,如下:
we performed deep single cell RNA sequencing to 7,066 immune cells collected from 8 TNBC patients using the MIRALCS platform. We identified 13 immune cell subsets according to their transcriptome features. We found that the frequency of identified T cells and macrophages varied greatly across patients.
结论居然仅仅是不同免疫细胞亚群在不同病人的比例差异很大!所以只能是一个摘要。
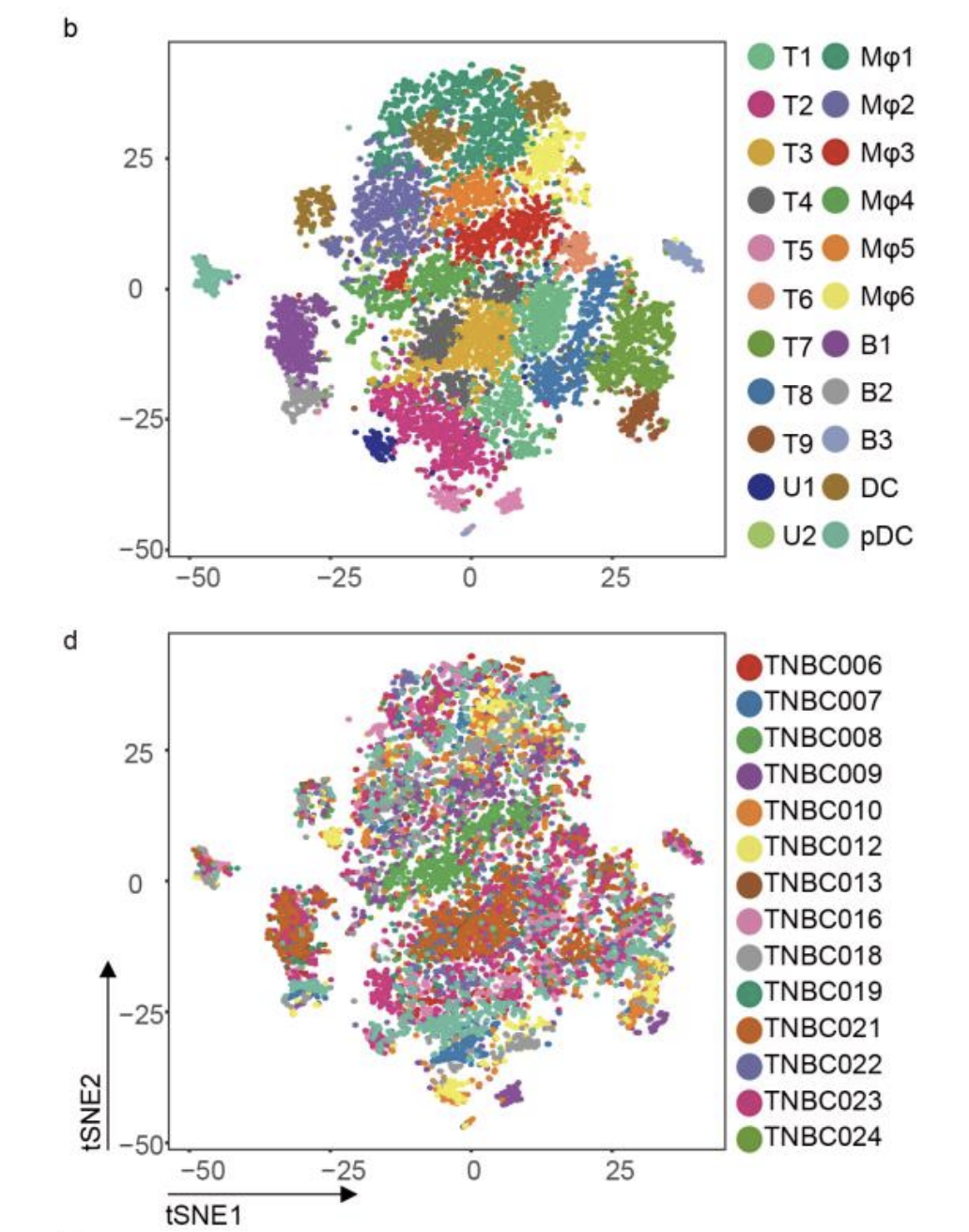
然后研究团队把之前的8个TNBC病人队列扩大到了14个病人,细胞数量也由之前的七千多到接近一万个。但仍然是丢在2019年5月的预印本:https://www.biorxiv.org/content/10.1101/566968v1
Here we report single-cell RNA sequencing results of 9683 tumor-infiltrated immune cells isolated from 14 treatment naïve TNBC tumors, where 22 immune cell subsets
这次的免疫细胞亚群更多了,如下:
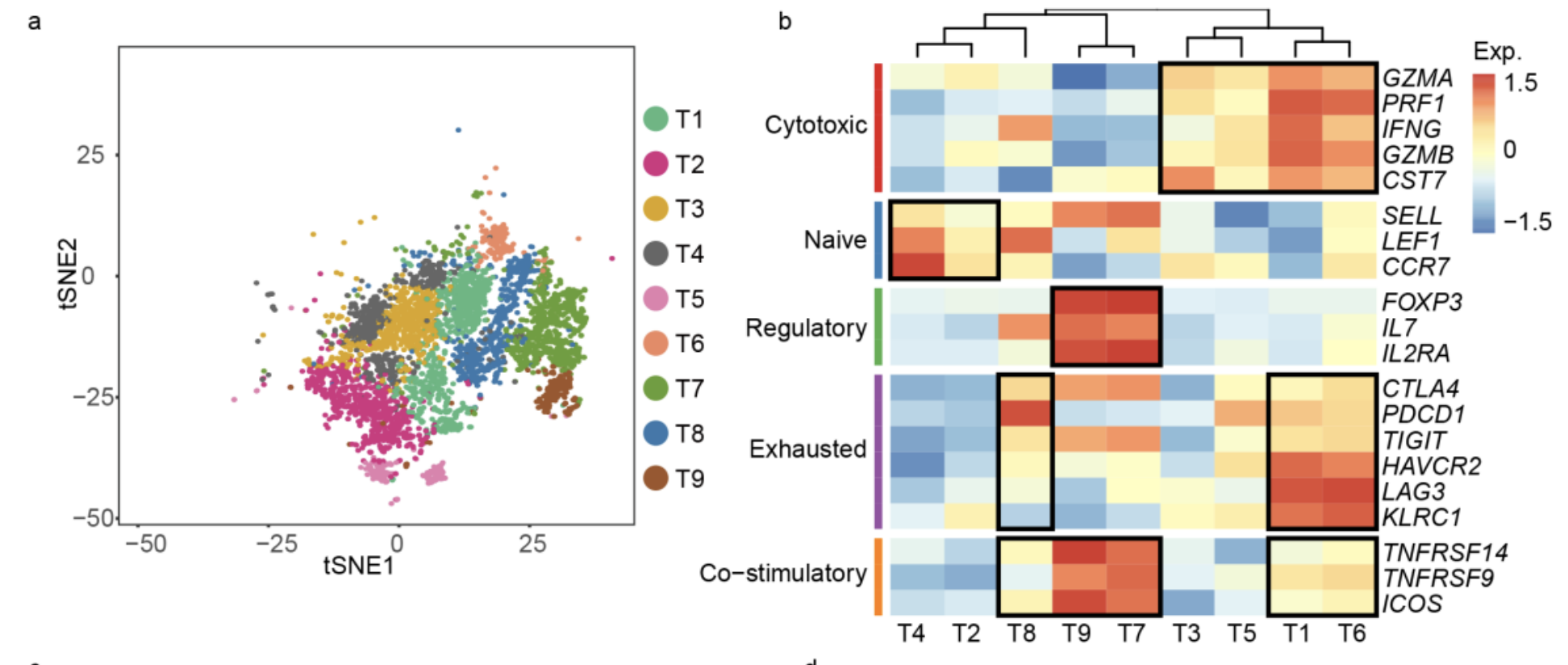
可以看到肿瘤病人的免疫细胞其实是没有病人特异性的,不同病人的免疫细胞会很好的混在一起,但是会依照免疫细胞的亚群来区分,仅仅是T细胞就有9个亚群,每个亚群特有的高表达量基因是:
而且有一个评价很有意思,感兴趣的可以直接去https://www.biorxiv.org/content/10.1101/566968v1围观哈。
仍然是停留在细胞分群以及亚群注释阶段,其实就是我们一直讲解的R包及基础流程,分别是: scater,monocle,Seurat,scran,M3Drop 需要熟练掌握它们的对象,:一些单细胞转录组R包的对象 流程也大同小异:
- step1: 创建对象
- step2: 质量控制
- step3: 表达量的标准化和归一化
- step4: 去除干扰因素(多个样本整合)
- step5: 判断重要的基因
- step6: 多种降维算法
- step7: 可视化降维结果
- step8: 多种聚类算法
- step9: 聚类后找每个细胞亚群的标志基因
- step10: 继续分类
其中一个步骤是判断重要的基因,我分享过:比较5种scRNA鉴定HVGs方法 里面就提到了 。这不是深圳华大基因第一次丢单细胞转录组数据在预印本了
BGI-Shenzhen团队之前也有一个牵头的乳腺癌的10X单细胞转录组测序项目,题目是:Comprehensive analysis of immune evasion in breast cancer by single-cell RNA-seq
关注的是乳腺癌病人的肿瘤微环境的细胞类型,尤其是免疫细胞如何作用于癌症细胞。链接是:. doi: http://dx.doi.org/10.1101/368605 bioRxiv preprint first posted online Jul. 13, 2018; 研究纳入了 15个病人,总共五万多单细胞,是商业的10x技术。- luminal A (P01, 5 P02, P03, P04, P05, P06, P07A, P07B and P08)
- luminal B (P09 and P10)
- HER2+ (P11 and P12)
- TNBC (P13, P14 and P15).
单细胞建库是:Chromium Single-Cell Instrument (10X Genomics) to generate single-cell gel bead-in-emulsions (GEMs). 有趣的是测序阶段使用的是 BGIS EQ-500 。 同时做了少量的wgs和wes数据,使用的也是 BGIseq Human Exome V4 Kit (BGI) 自己的wes捕获芯片。补充知识
为什么我会搜索到这两个预印本的单细胞转录组研究呢,其实是因为我恰好看到了MIRALCS系统,在这个系统上可以完成细胞的分离及扩增,从而减少了操作误差,实验的可靠性更高和平行性更好
此方法学的文章:
Wu et al. Full-length single-cell RNA-seq applied to a viral human cancer: applications to HPV expression and splicing analysis in HeLa S3 cells. GigaScience (2015) 4:51.
当然了,大家熟知的4种单细胞分离的技术,其实是:- 激光显微捕获切割技术(LCM)
- 口吸管分离技术
- 流式细胞分离技术(FACS)
- 免疫磁珠分离技术(MACS)