学TCGA数据库并不是为了发“套路”文章,那些基本上是本科生毕业设计的水准,但是阅读这样的文章的确可以很大程度上帮助大家认识TCGA数据库,主要是了解它可以做什么,比如我昨天在生信技能树介绍的:[需要5个步骤来说明你想研究的基因的重要性](https://mp.weixin.qq.com/s/L7wE70rmKlEKfi6ZGV7mew) ,就是纯粹使用TCGA数据库说明自己研究课题的目标基因很重要,可以节约大量的课题经费,避免重复设计那些明明是可以通过公共数据库获得结论的实验。
TCGA数据挖掘真的是绵绵不绝,这里就不再赘述了,从基因集到ceRNA,到可变剪切,肿瘤免疫, 再到现在的m6A和自噬基因, 马上缺氧应该是也要出来了,每次一个策略就是33篇数据挖掘文章。真的是很不走心,比如最近有学徒一直咨询我关于m6A,我发现仅仅是ccRCC的就有4篇,有一个很简陋的文章,完全是6个网页工具图表拼凑的,再加上一点点多组学。
文章:Identification of METTL14 in Kidney Renal Clear Cell Carcinoma Using Bioinformatics Analysis
链接:https://doi.org/10.1155/2019/5648783 (2019)
发表历程:Received 18 Aug 2019,Accepted 04 Nov 2019,Published 30 Dec 2019
- GTExPortal and TCGAportal
- OncoLnc and starBase
- circBank
- HPA
首先是TCGAportal看13个m6A基因的表达量热图
因为不同基因表达量不一样,我们其实并不想知道基因互相之间的高低,我们想看到的是基因在normal和tumor组的高低,但是下面的热图很明显是不直观的,是热图的大忌!
如下:
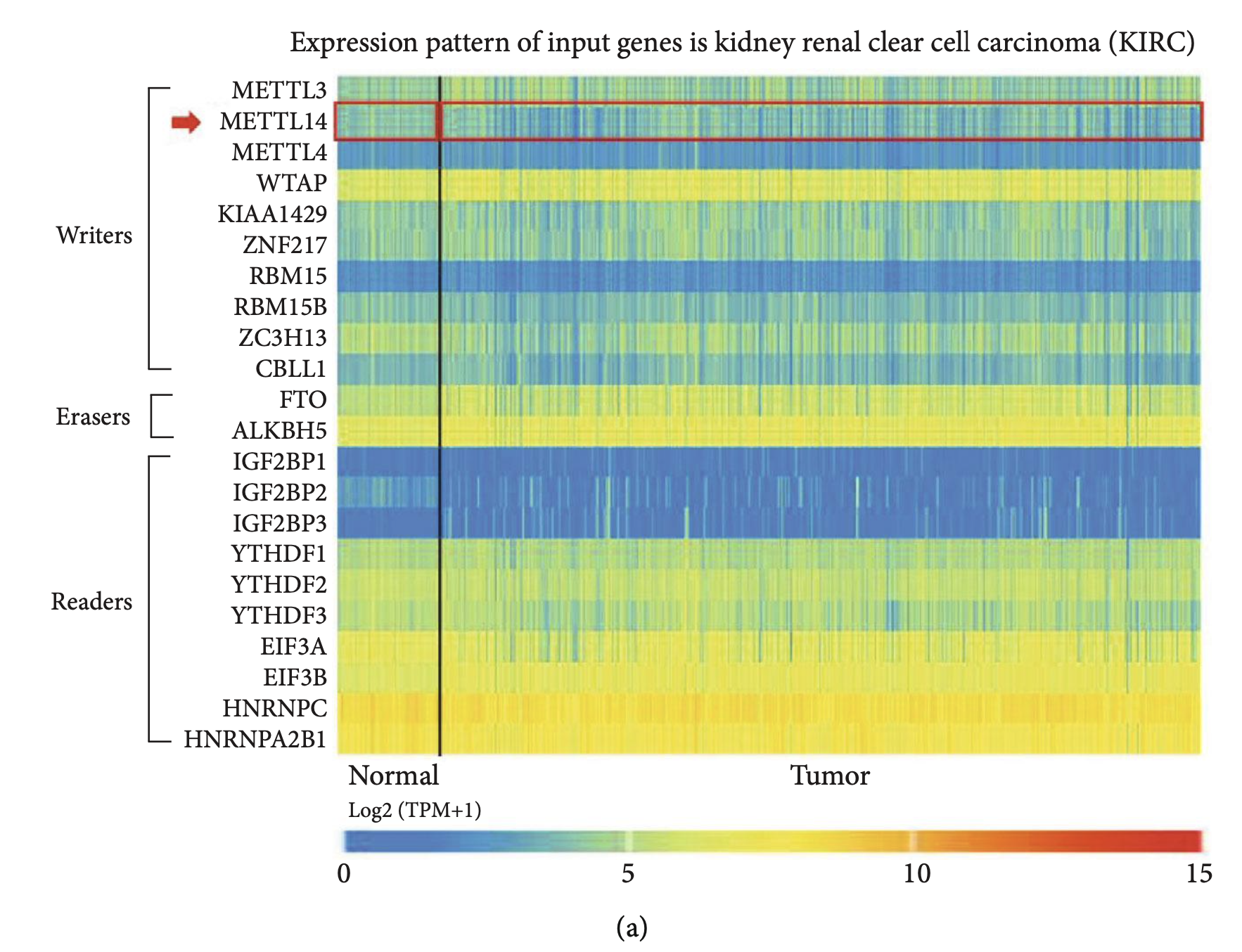
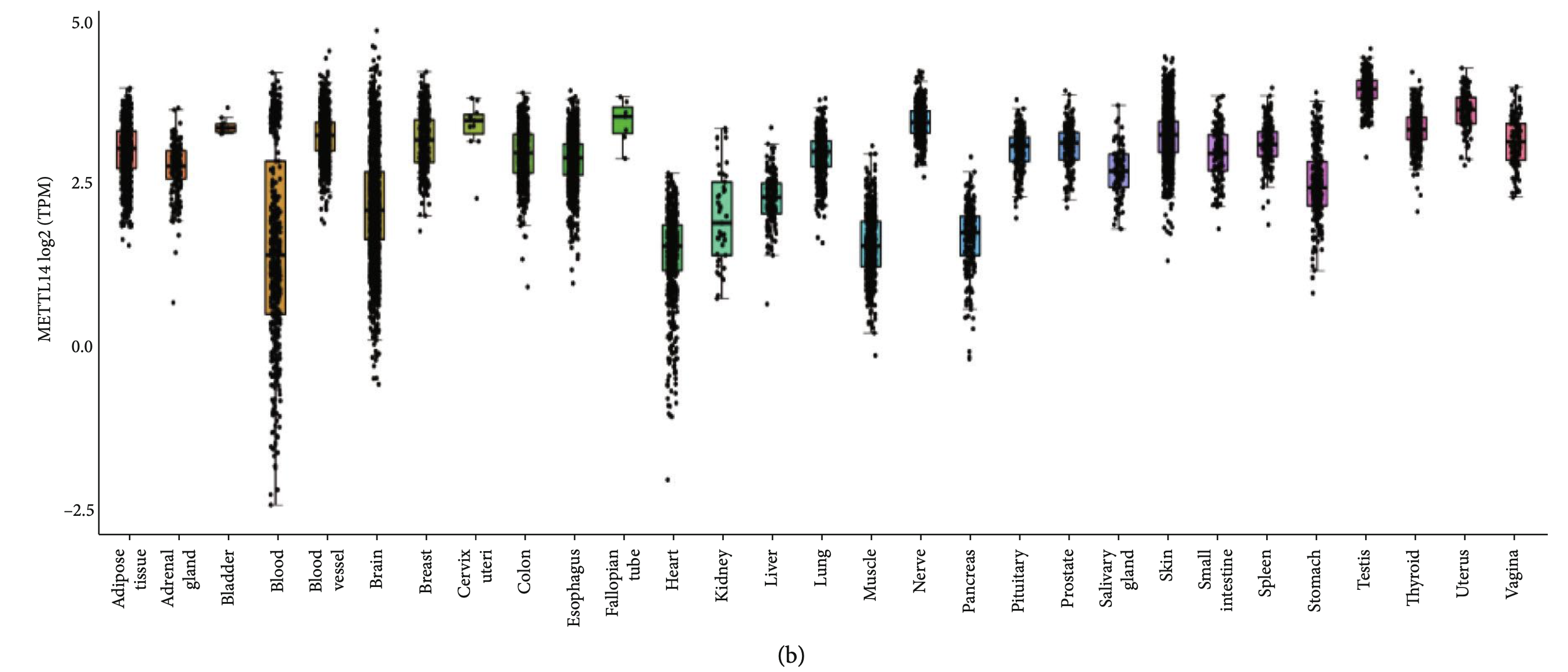
作为生信技能树粉丝的你们,经过了我这么多教程的熏陶,应该是知道上面的热图如何改进吧,以及如何去获取13个m6A基因在ccRCC的表达量矩阵!然后是GTExPortal的一个基因的多器官组织表达量分布图
我布置的学徒作业:在CCLE数据库里面根据指定基因在指定细胞系里面提取表达矩阵 , 提到过类似的,需要下载CCLE数据库的RNA-seq表达矩阵,然后在R里面根据指定基因在指定细胞系里面提取表达矩阵即可。作业发在生信菜鸟团,见:学徒带你一步步从CCLE数据库里面根据指定基因在指定细胞系里面提取表达矩阵进行热图可视化。
GTEx,The Genotype-Tissue Expression (GTEx) project,首次被提出来是2013年,上百位科学家联名在Nature Genetics杂志发表的文章首次介绍了“基因型-组织表达工程”,并成立了“基因型-组织表达研究联盟”(Genotype-Tissue Expression Consortium,GTEx)以下简称“GTEx”)。本质上也是需要下载指定基因表达量而已
如下:
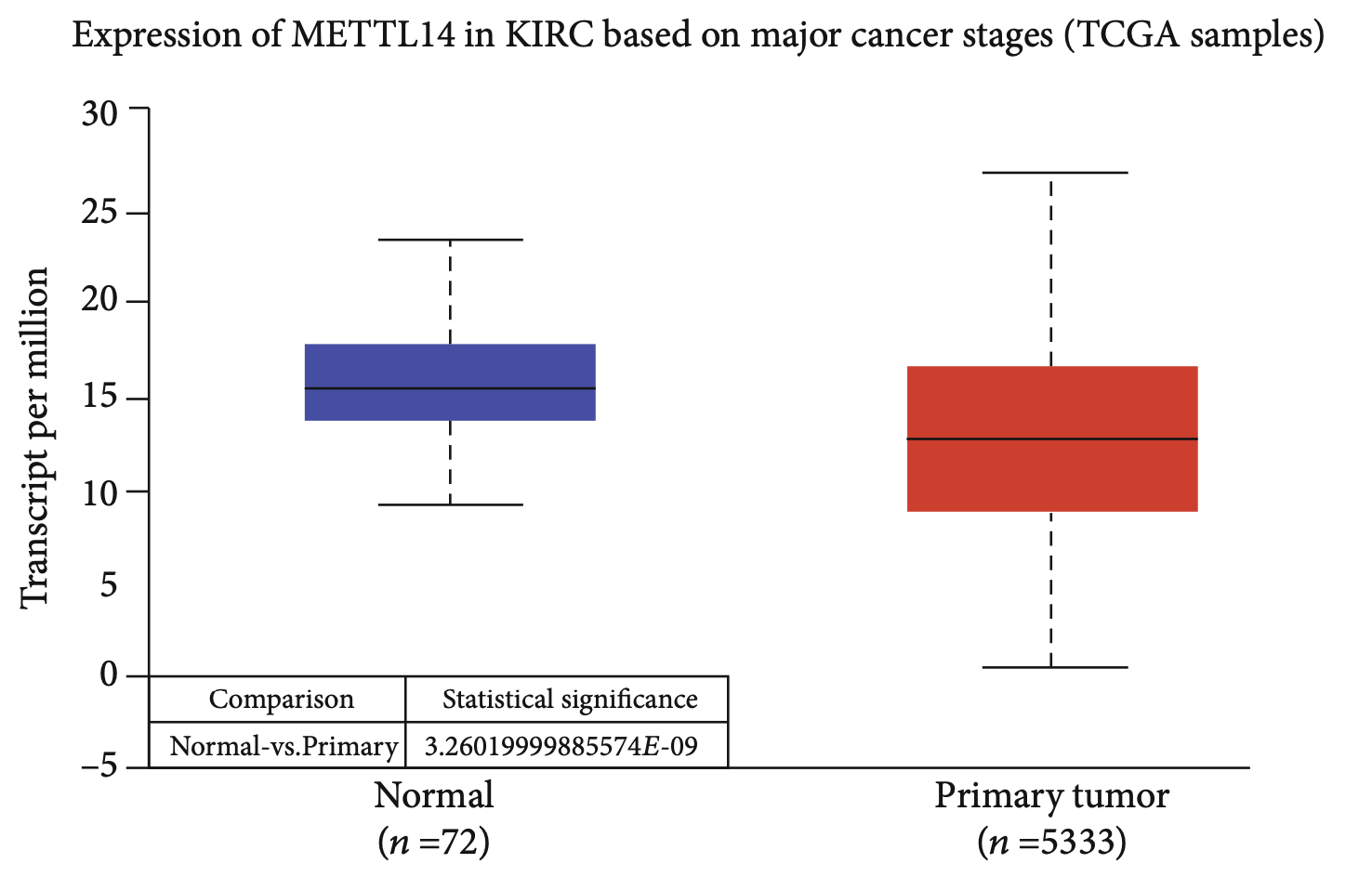
接着是一个基因的肿瘤表达差异图
如下:
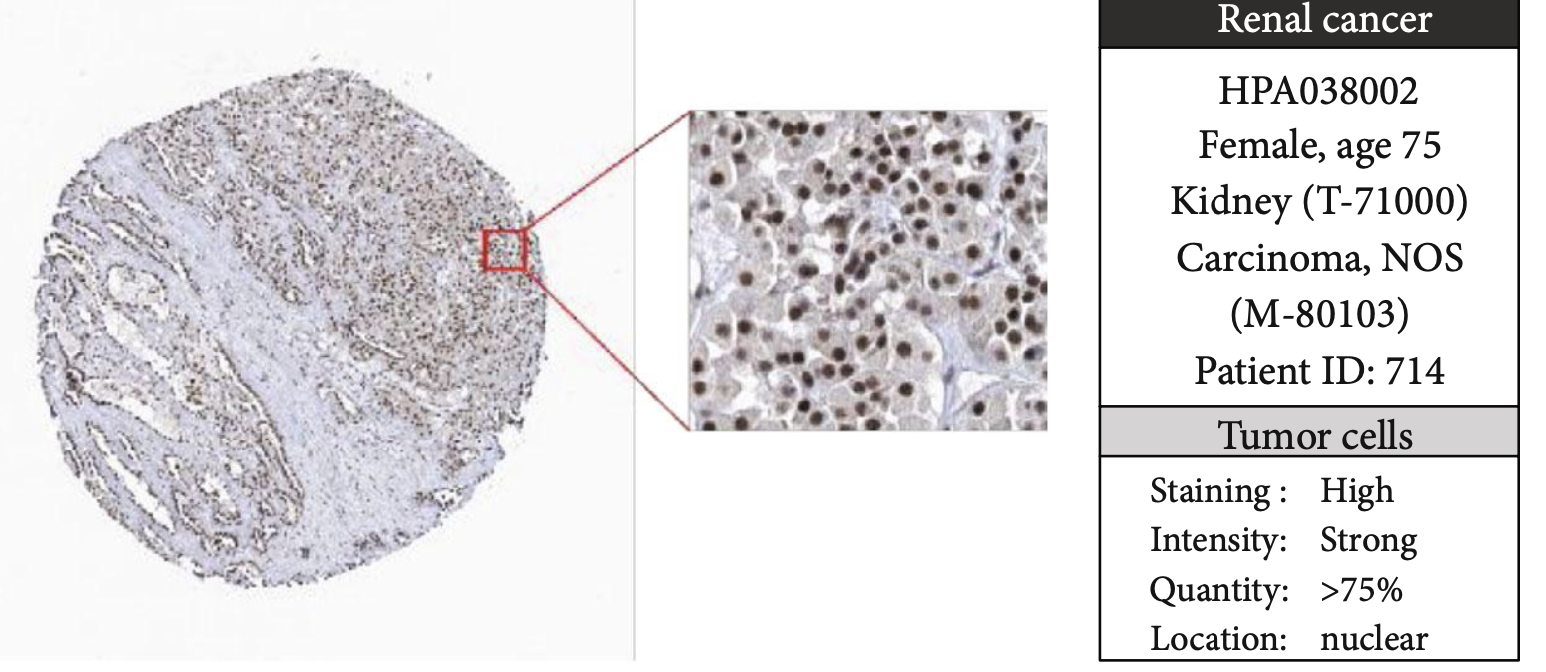
接着是HPA数据库的指定基因的IHC图
前面我也在生信技能树分享过:学会看IHC病理切片从HPA开始
如下:
然后是oncoLNC数据库的生存分析
从图的样式来说,应该是km-plot风格,但是其实我是不相信km-plot网页工具结果的。我们已经多次介绍过生存分析:
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
作者出图如下:
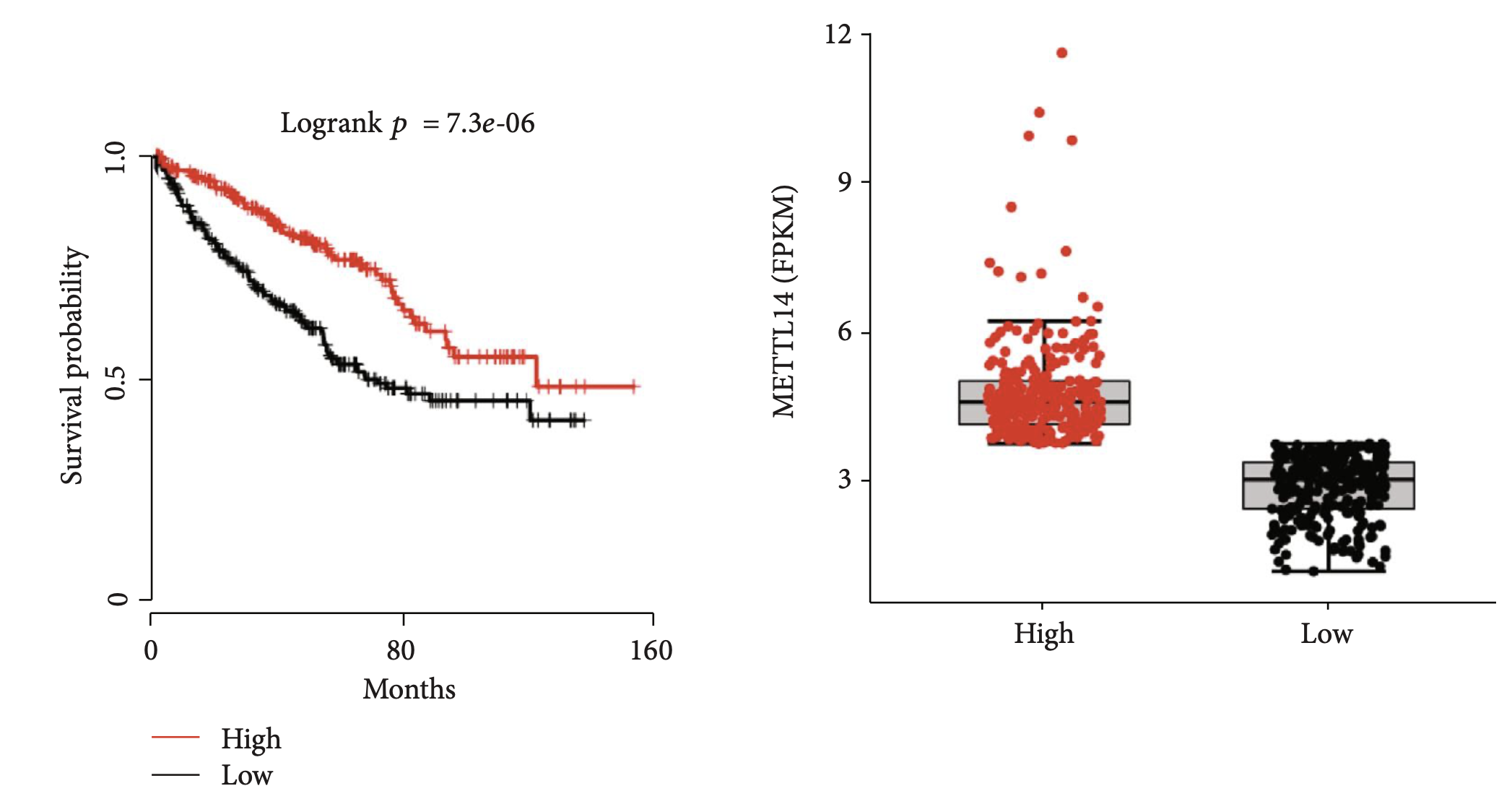
还有mRNA表达水平和CNV的箱线图
这里就是作者觉得网页工具一直出表达量的图有点不好意思,就加入了CNV信息,算是搭上了多组学的便车。
如下:
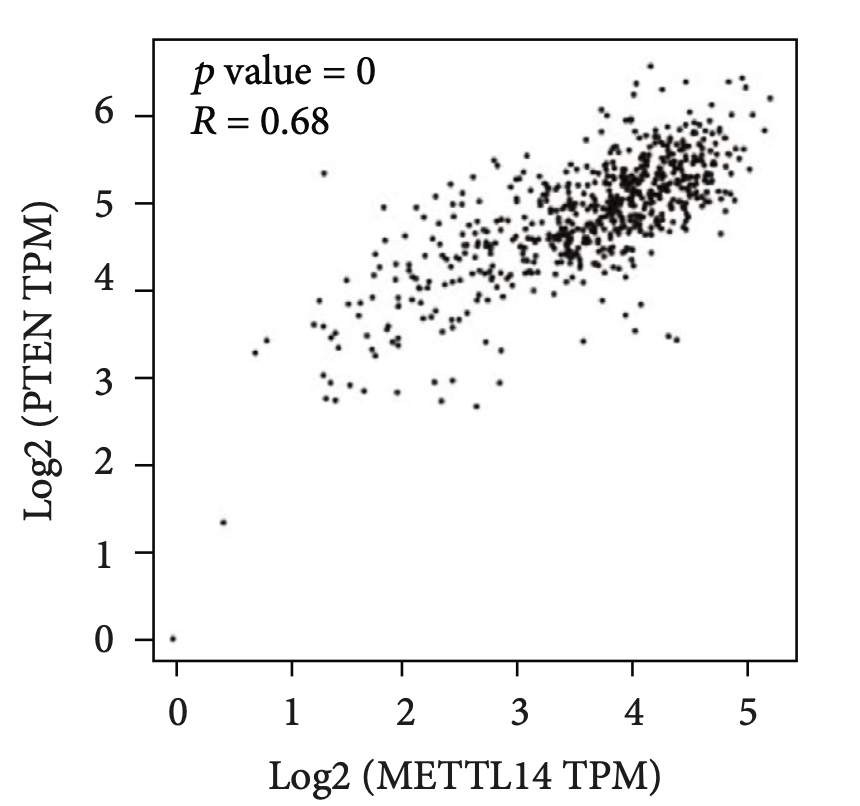
以及两个基因的mRNA表达量相关性散点图
同样的也是UCSC的xena浏览器,或者GEPIA2可以做到,感兴趣的可以去学习:GEPIA2详解(中国智造-肿瘤数据库),当然了,也可以自行编程探索。需求最大的是tcga数据库的生存分析和表达量差异,看看这两个视频:
- https://www.bilibili.com/video/av25643438?p=9
- https://www.bilibili.com/video/av49363776?p=6
如下:
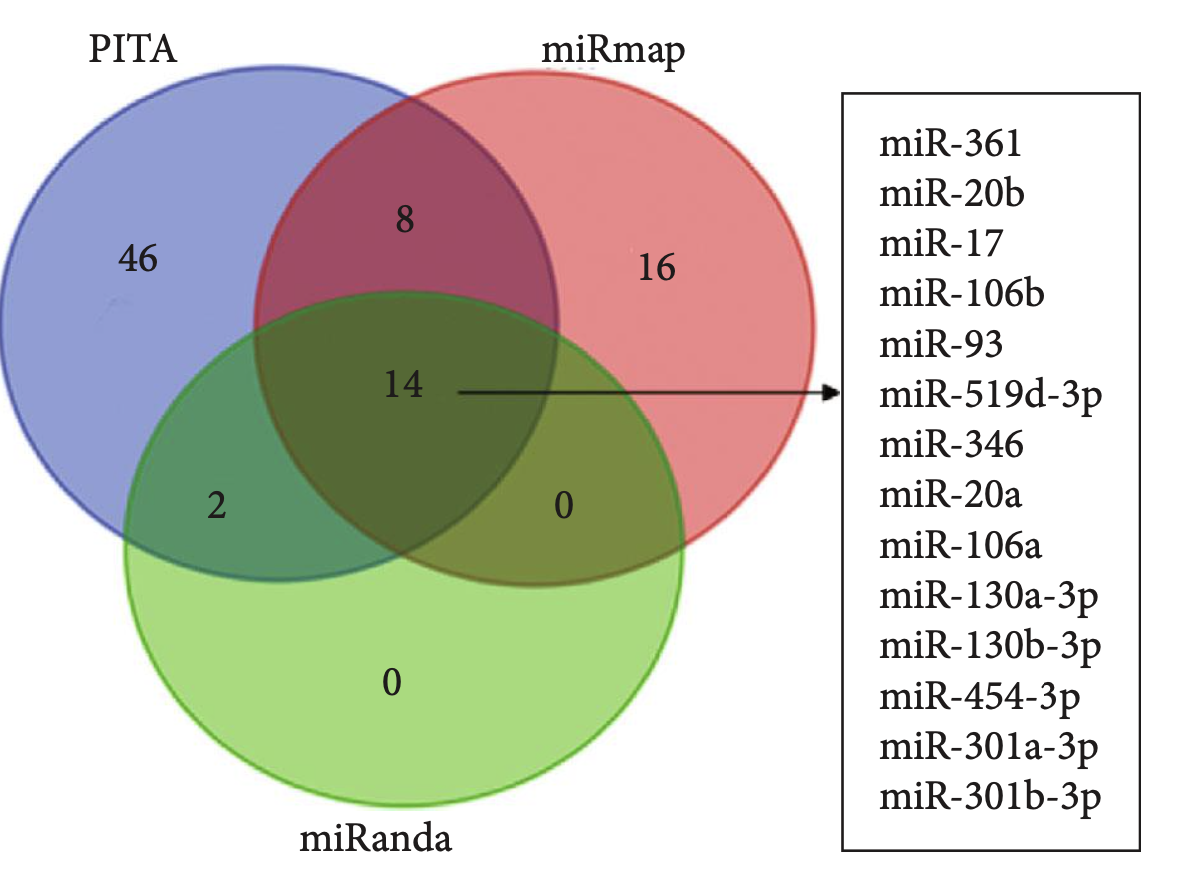
还有多个miRNA数据库的交集
自己看文章里面,数据库忒多了。感觉绘制下面的miRNA也是凑图吧!
如下:
其实还不够猛,仅仅是网页数据库比较多,多组学也就是mRNA,CNV,miRNA,还有很多都没有利用起来呢!这样的数据挖掘文章里面多组学通常是噱头
TCGA数据库是目前最综合最全面的癌症病人相关组学数据库,包括:
- DNA Sequencing
- miRNA Sequencing
- Protein Expression array
- mRNA Sequencing
- Total RNA Sequencing
- Array-based Expression
- DNA Methylation
- Copy Number array
知名的肿瘤研究机构都有着自己的TCGA数据库探索工具,比如: - Broad Institute FireBrowse portal, The Broad Institute
- cBioPortal for Cancer Genomics, Memorial Sloan-Kettering Cancer Center
所以我挑选了部分,写了6个数据下载系列教程: - TCGA的28篇教程- 使用R语言的cgdsr包获取TCGA数据(cBioPortal)
- TCGA的28篇教程- 使用R语言的RTCGA包获取TCGA数据 (离线打包版本)
- TCGA的28篇教程-使用R语言的RTCGAToolbox包获取TCGA数据(FireBrowse portal)
- TCGA的28篇教程- 批量下载TCGA所有数据 ( UCSC的 XENA)
- TCGA的28篇教程-数据下载就到此为止吧
- TCGA的28篇教程-整理GDC下载的xml格式的临床资料
最后还是推荐ucsc的xena数据源下载即可。文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!