组会上同学分享了一篇实验生物学文章,有趣的是里面用到了RNA-seq,而且数据量不行,不过分析的点非常少,由此可以说明RNA-seq技术的常规化了。
背景介绍
多发性骨髓瘤(Multiple Myeloma,MM)
骨髓瘤又称浆细胞瘤,有多种类型,其中多发性骨髓瘤(Multiple Myeloma,MM)最为常见。
多发性骨髓瘤(MM)是B细胞恶性肿瘤,它导致破骨细胞活化和成骨细胞减少,引起骨质破坏和疼痛性骨折。
MDSC(髓源性抑制细胞)是一群髓系来源具有抑制功能的天然免疫细胞,在肿瘤进展中发挥负向免疫调控作用。
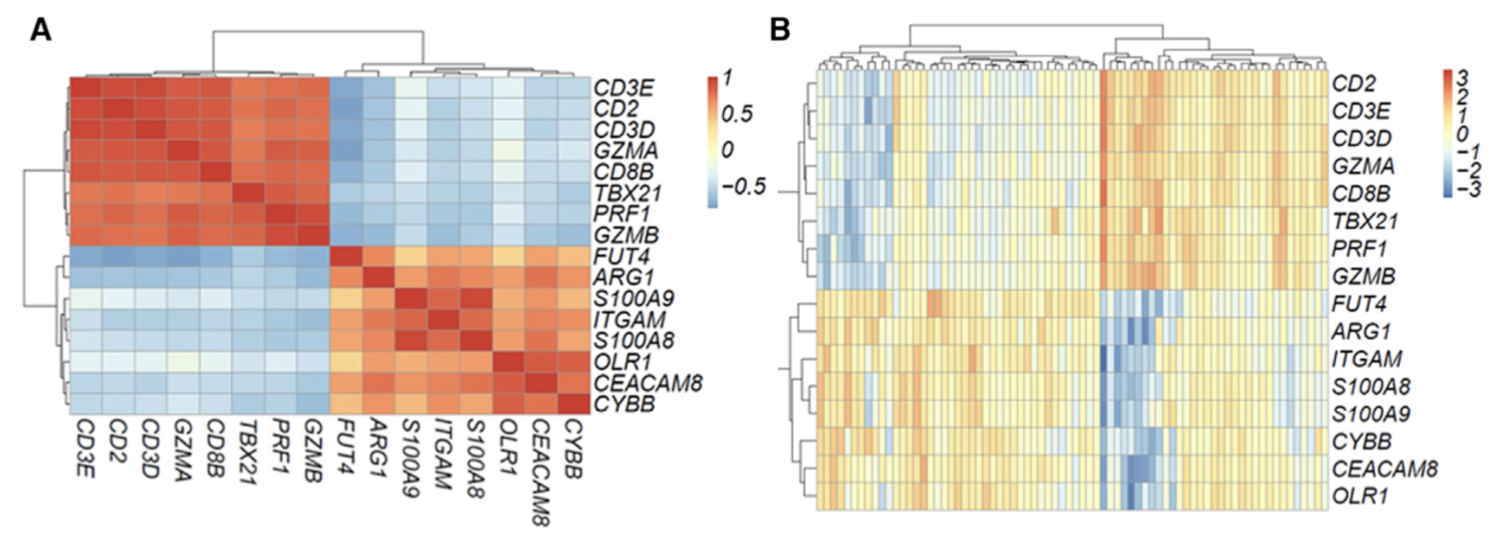
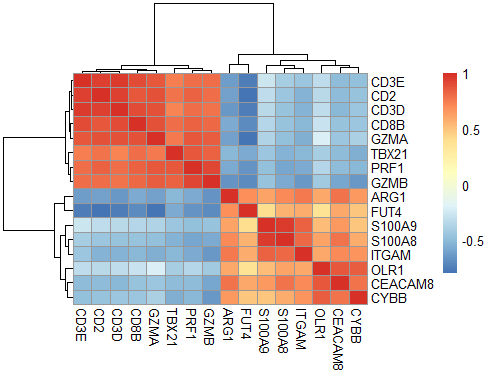
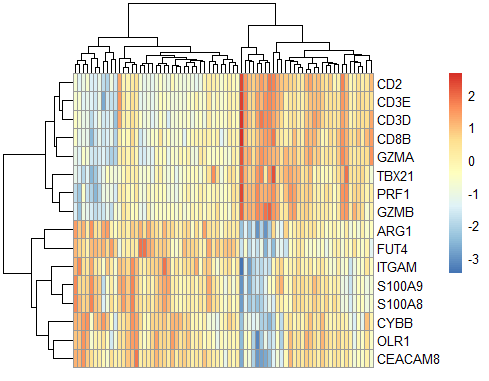
PMN-MDSC signature genes (ITGAM, ARG1, CYBB, OLR1, FUT4, CEACAM8, S100A8,S100A9)
cytotoxic lymphocyte signature genes (CD2,CD3E, CD3D, TBX21, CD8B, PRF1, GZMA,GZMB)
2014的200人队列
Multiple Myeloma (Broad, Cancer Cell 2014) Lohr et al. Cancer Cell 2014
https://www.cbioportal.org/study?id=mm_broad
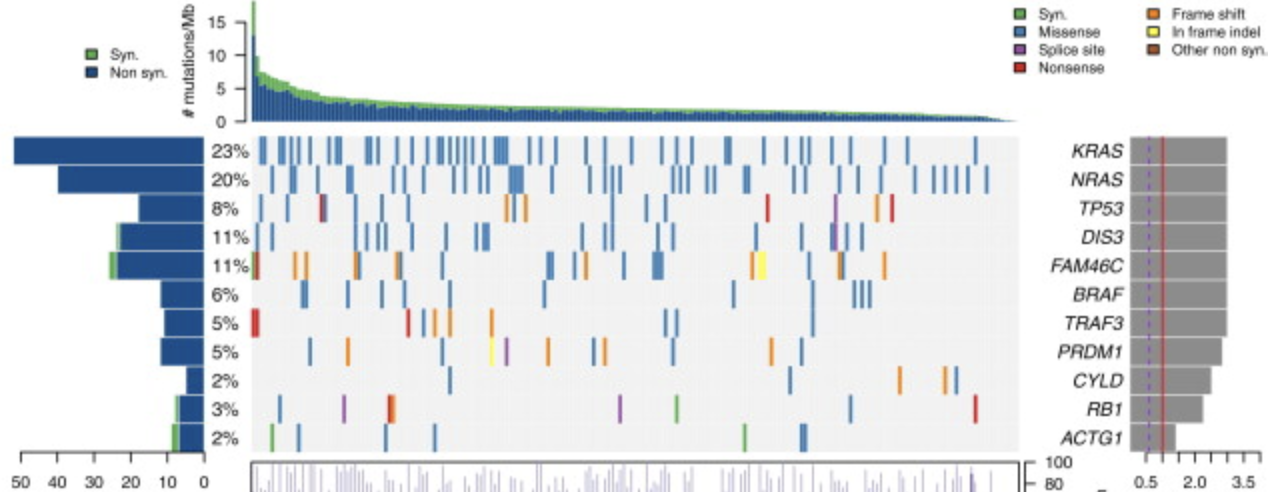
As part of the MMGI study, the Broad Institute has completed the genome sequencing, using WGS and WES, for 213 patients. A frequently mutated list of 9 genes including NRAS, KRAS, TP53, PNRC1, MAGED1, FAM46C, DIS3, CCND1 and ALOX12B were identified initially.
仅仅是DNA层面的研究,包括WGS和WES,得到了KRAS、NRAS、TP53这些热点突变基因列表。
关于MM的转录组视频研究不多。实验设计
首先73个病人的RNA-seq测序,数据开放下载:
重点关注MDSC and cytotoxic lymphocyte基因集的表达量相关性图和表达量热图:
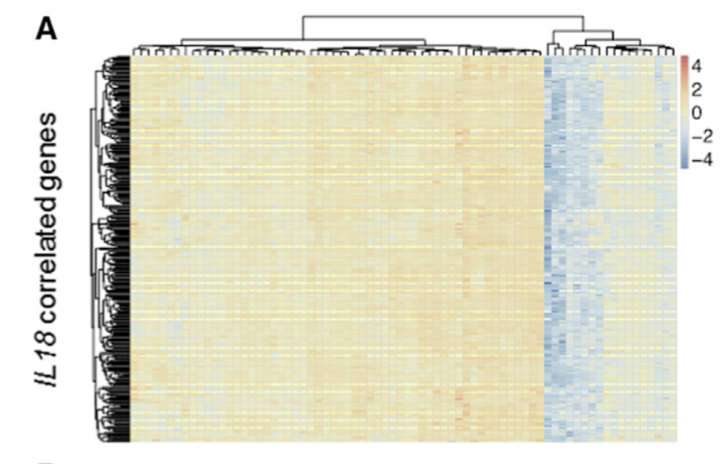
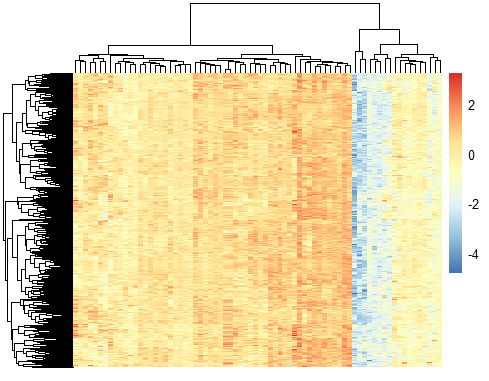
展示跟 IL18基因表达量相关性大于0.7的基因的表达量热图:
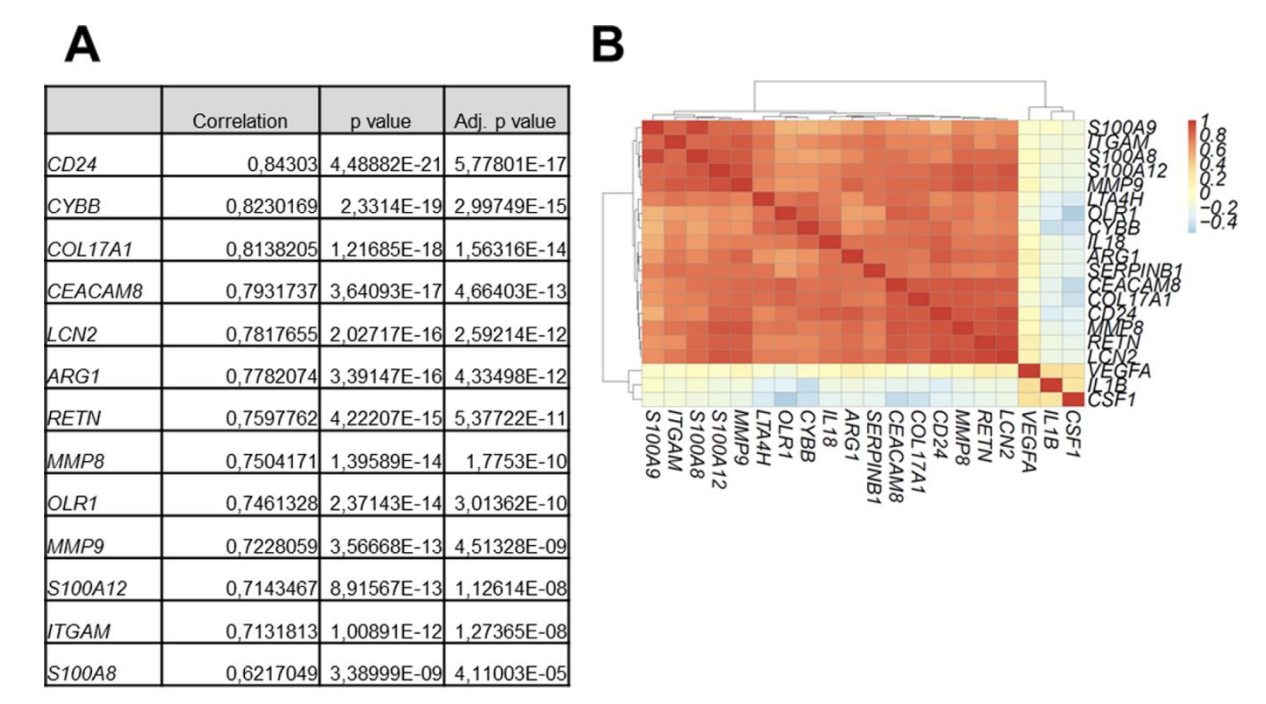
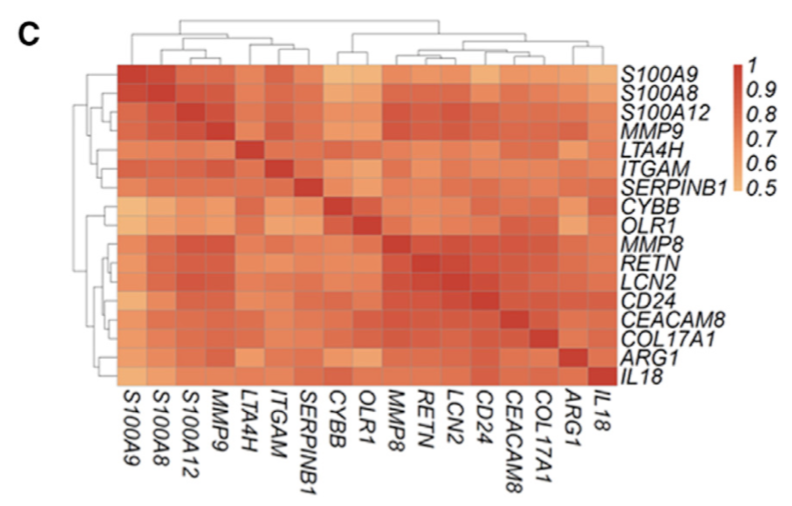
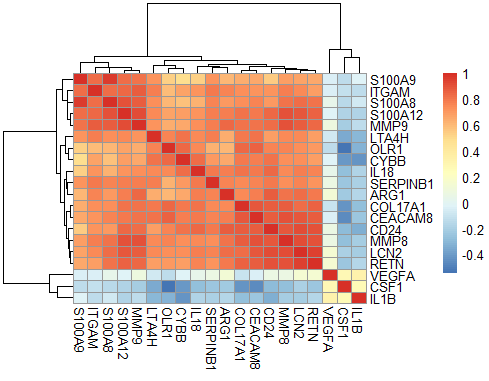
展示指定基因跟 IL18基因表达量相关性,以及相关性热图,基因集如下:
再关注 PMN-MDSC signature genes
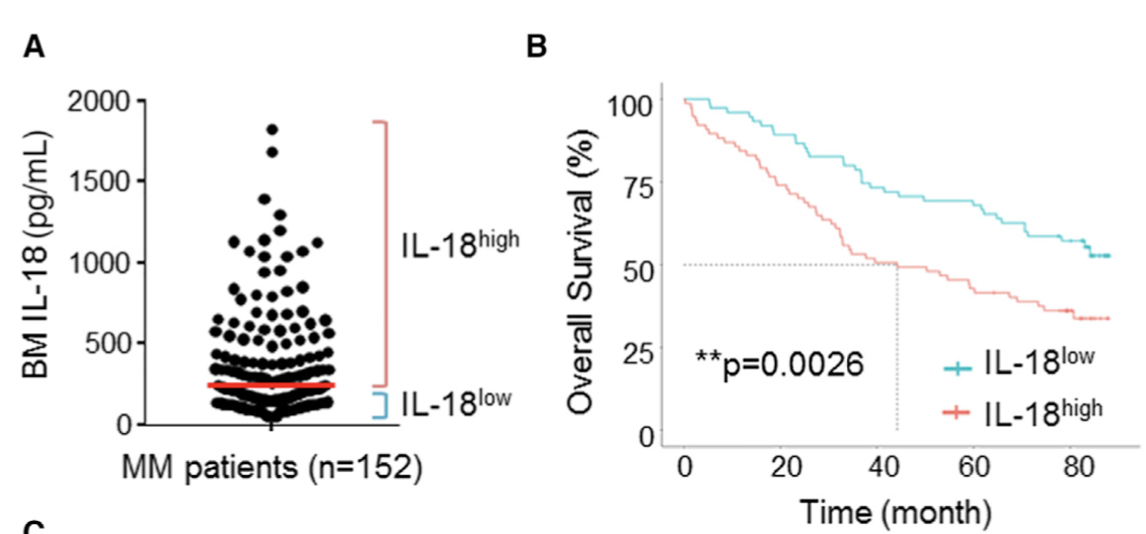
扩大病人队列,收集完整的临床随访信息,就可以做生存分析及其它。
数据分析复现
其实还是一堆热图,有点儿简单,就几十行代码,由资质平平的学徒写出。
a=read.table('GSE104171_NormalizedMatrix.txt.gz', sep = ' ',header = T,row.names = NULL) cor_gene<-c('CD2','CD3E','CD3D','TBX21','CD8B','PRF1','GZMA','GZMB','ITGAM','ARG1','CYBB','OLR1','FUT4','CEACAM8','S100A8','S100A9') experSet<-a[a$row.names%in%cor_gene,] rownames(experSet)<-experSet$row.names exprSet=experSet[,-1] pheatmap::pheatmap(cor(t(exprSet))) matrix_chosen<-t(scale(t(exprSet))) tmp<-colnames(matrix_chosen) tmp<-rep('',ncol(matrix_chosen)) pheatmap::pheatmap(matrix_chosen,labels_col=tmp,legend_breaks=seq(-3,3,1)) ######correlation cor_g=cor(t(a[,-1])) which(a[,1]=='IL18') cor_IL18=data.frame(gene=a[,1], cor=cor_g[,7258]) cor_gene7<-cor_IL18$cor>0.7 a_cor<-a[cor_gene7,-1] chosen_cor<-t(scale(t(a_cor))) tmp1<-colnames(chosen_cor) tmp1<-rep('',ncol(chosen_cor)) pheatmap::pheatmap(chosen_cor,show_rownames = F,labels_col=tmp1,legend_breaks=seq(-4,4,2)) #####特定基因相关性及热图 target<-c('CSF1','VEGFA','IL1B','IL18','ITGAM','ARG1','S100A9','S100A8','LTA4H','S100A12','CEACAM8','MMP9','SERPINB1','CYBB','OLR1', 'RETN', 'LCN2','CD24','MMP8','COL17A1') tar_cor<-cor_g[a[,1]%in%target,a[,1]%in%target] rownames(tar_cor)<-a[,1][a[,1]%in%target] colnames(tar_cor)<-a[,1][a[,1]%in%target] pheatmap::pheatmap(tar_cor)图形结果展示,大家随便看看吧:
TCGA数据库应用
在这儿就不连篇累牍地讲TCGA了,毕竟,我悄咪咪地上新了TCGA的视频,大家可以移步到B站https://www.bilibili.com/video/av49363776 或 YouTube https://www.bilibili.com/video/av49363776 去学习。
关于倒计时删除视频这种事情,我可是说到做到的。相关研究
Dana-Farber研究所的科学家们对75名多发性骨髓瘤患者的110份血液样本进行基因组测序,并利用检测的结果预测cfDNA外显子测序的准确性。此外,作者还收集了健康人群血液中的cfDNA,将其与骨髓瘤患者的cfDNA进行比对。结果显示,cfDNA外显子测序能够充分地鉴定所有遗传突变的情况,而且这些突变与通过对骨髓细胞进行基因组测序得到的结果十分吻合。
2017年12月 5 日,北京大学生科院李程研究组在《自然-通讯》发表最新研究成果,题为“3D genome of multiple myeloma reveals spatial genome disorganization associated with copy number variations”。李程研究组使用代表两种亚型的骨髓瘤细胞(近二倍体U266和近三倍体RPMI-8226)和正常B细胞(GM12878),对拷贝数变异(CNV)和三维基因组的关系展开研究。
《Inhibition of microrna-138 enhances bone formation in multiple myeloma bonemarrow niche》是发表在Leukemia (2018) (IF=11.702) 上的一篇文章,在本文研究中,作者探讨了miR-138在骨髓瘤骨病中的作用,并评估了anti-miR-138寡核苷酸在骨髓瘤骨病中的可能作。研究发现,与健康对照相比,在多发性骨髓瘤病人的间充质干细胞(MSC)和骨髓瘤细胞中,miR-138的表达显著增加。