前面我们组建了:免费视频课程《RNA-seq数据分析》交流群 ,很多人马上学习了全套实战视频,并且实践了一个转录组项目流程,新手问的最多的问题是:为什么我给的RNA-seq表达矩阵代码需要的是counts格式输入,而且使用3个不同的R包做差异分析,这3个R包该分别如何设置阈值来进行统计学显著的差异表达基因筛选,以及多个R包数量有时候差异很大,该如何取舍。
我的回答,统一是:不要看数量,要看质量!!!
早在教程:RNA芯片和测序技术的比较(学徒作业),我其实就提出来了,比较同样实验设计的两个表达量探索研究,一个研究使用的是芯片,一个是测序,看看两者的差异基因情况的overlap情况,这样的策略其实是太粗糙了。正确的做法应该是看两次差异分析的基因的logFC的散点图,如下:
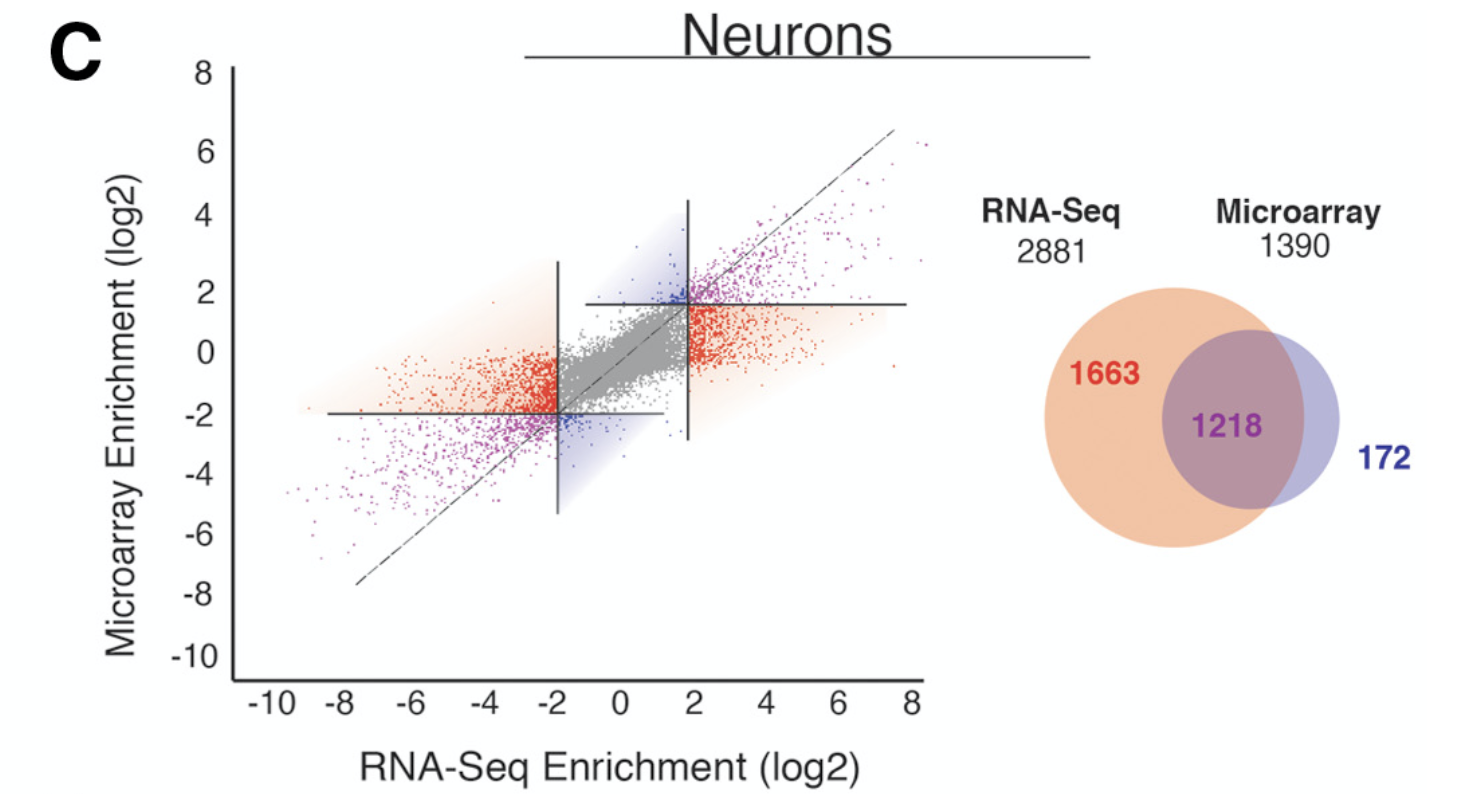
而且你可以进行更细致的探索,我们这里以文章:《RNA sequencing atopic dermatitis transcriptome profiling provides insights into novel disease mechanisms with potential therapeutic implications》为例子:
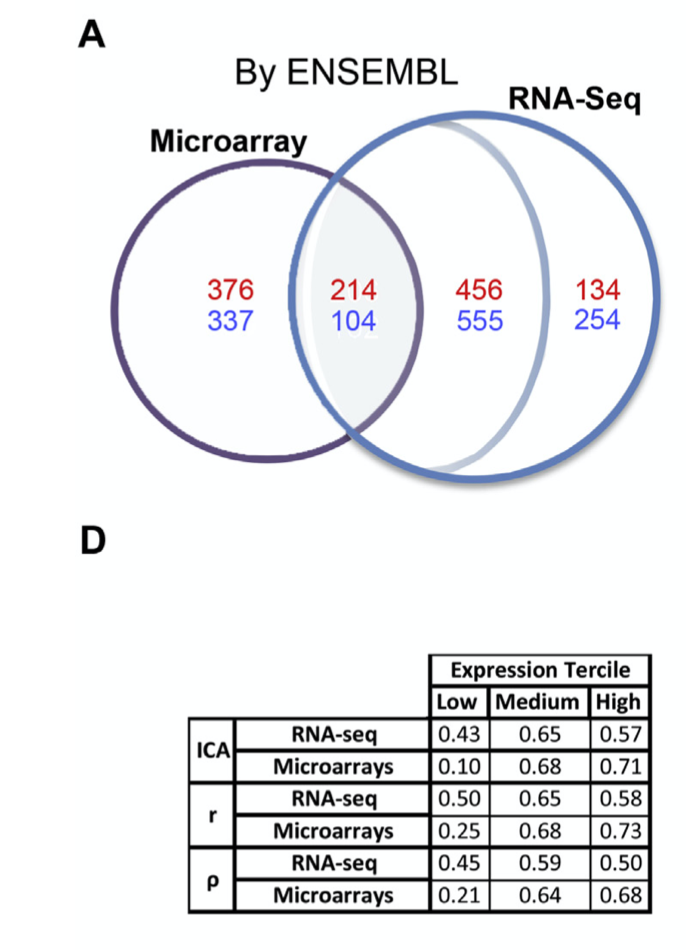
比如把基因按照表达量划分高中低三组后,再去看表达量相关性:
再比如选取那些两次差异不统一的基因进行后续功能富集,看看那些基因是否有很多生物学意义。
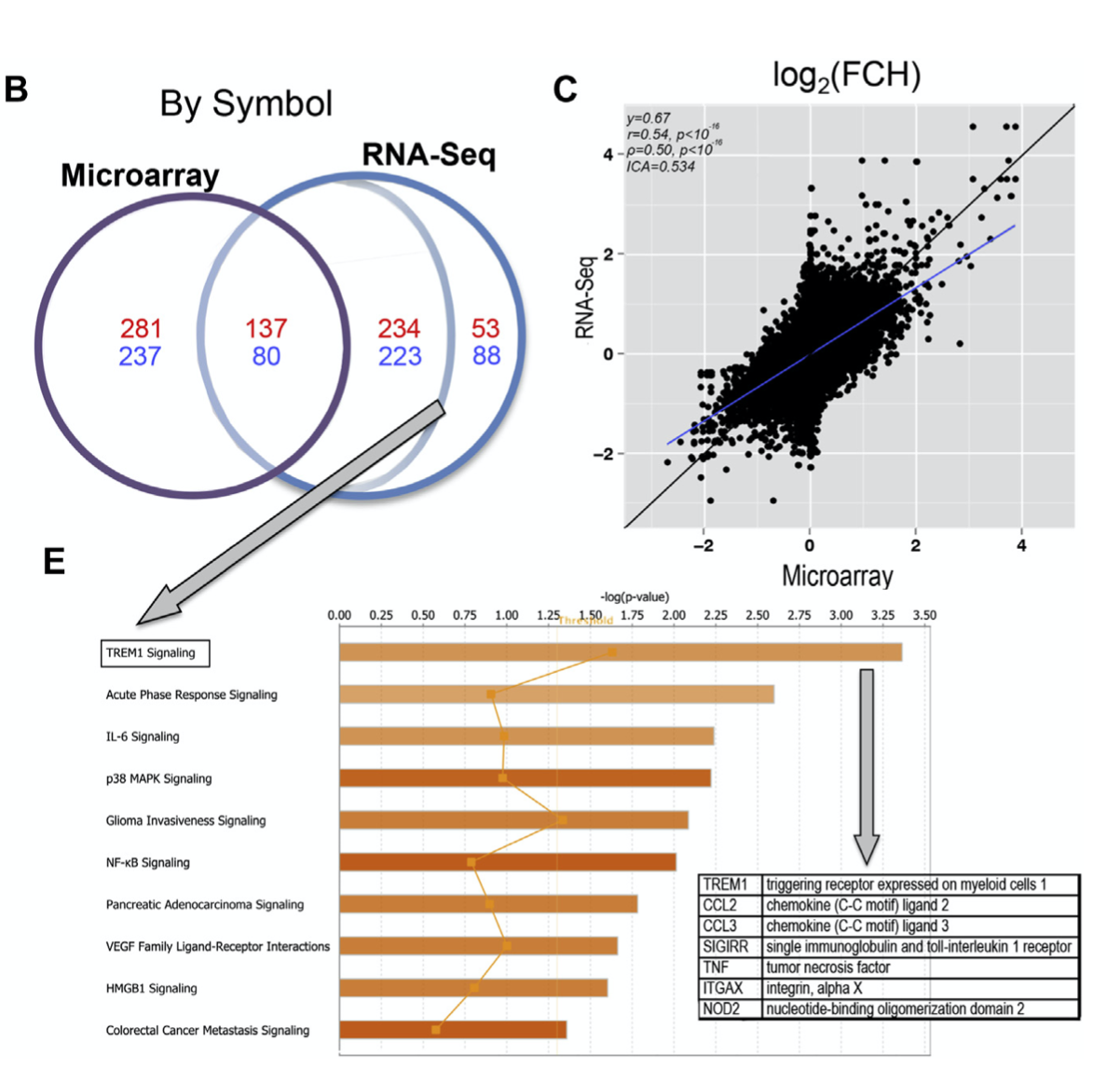
这样的探索才是合格的,首先要搞清楚流程,然后搞清楚流程里面的哪些细节是可以调整的, 而且理解调整前后的结果的变化的差异程度能够被接受与否。
以及如何论证不同流程,不同软件,不同参数,不同阈值的结果的差异背后的生物学意义。多做一些实战项目是有助于你理解差异分析的作用和本质,比如我带学徒就会安排他们一些图表复现:
- 1. 公共数据辅助乳腺癌的免疫治疗机制研究
- 2. 有生物学意义的复杂热图
- 3. 干扰MYC‑WWP1通路重新激活PTEN的抑癌活性——3步搞定GSEA分 析
- 4. 按基因在染色体上的顺序画差异甲基化热图
- 5. 热图、⻙恩图、GO富集分析图(有了转录组数据不知道该怎么写⽂ 章,看我就对了!)
- 6. 纯R代码实现ssGSEA算法评估肿瘤免疫浸润程度
- 7. 肿瘤异质性+免疫浸润细胞数据挖掘(可能是Y简单的3分⽂章了)
- 8. ArrayExpress数据库的基因芯⽚原始数据处理,3D主成分图及聚类热 图
- 9. 学徒数据挖掘第⼆期汇总之多分组基因注释代码⼤放送
- 10. TCGA数据辅助甲基化区域的功能研究
- 11. 你确定你的差异基因找对了吗?
- 12. 看nature⽂章是如何设计和使⽤普通转录组数据
- 13. 不⼀定正确的多分组差异分析结果热图展现
- 14. 如果传统bulk转录组数据队列⾜够⼤也可以使⽤单细胞流程
- 15. 最简单的芯⽚挖掘也会出错(菜⻦团周⼀数据挖掘专栏第?期)
- 16. 乳腺癌的IHC分类和PAM50分型的差异情况
- 17. 你要挖的公共数据集作者上传了错误的表达矩阵肿么办(如何让⾼⼿ ⼼⽢情愿的帮你呢?)
赠送全套生物信息学资料
如果你也有一些生物信息学相关经验或者想法需要分享,生信技能树提供平台哈,你可以发笔记也可以开直播,或者录制视频教程!只需要完成任意学徒作业,并且提交笔记给生信技能树jimmy,发邮件到 jmzeng1314@163.com ,合格者可以拿到全套生物信息学资料,只要你想要学的,只要我有,统统给你!
文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班:
如果你没有服务器的话,做NGS数据分析实战可能会有点勉强,建议考虑:每天不足一块钱,定制生信云送给你