看到一个研究,根据KIAA1429的表达量把TCGA数据库的乳腺癌患者分组后做生存分析,发现有统计学显著,描述如下:
Breast cancer patients were divided into low-KIAA1429 expression (n = 528) group and high-KIAA1429 expression (n=528) group. The high KIAA1429level correlated with poor survival rate. P = 0.003
可视化如下:
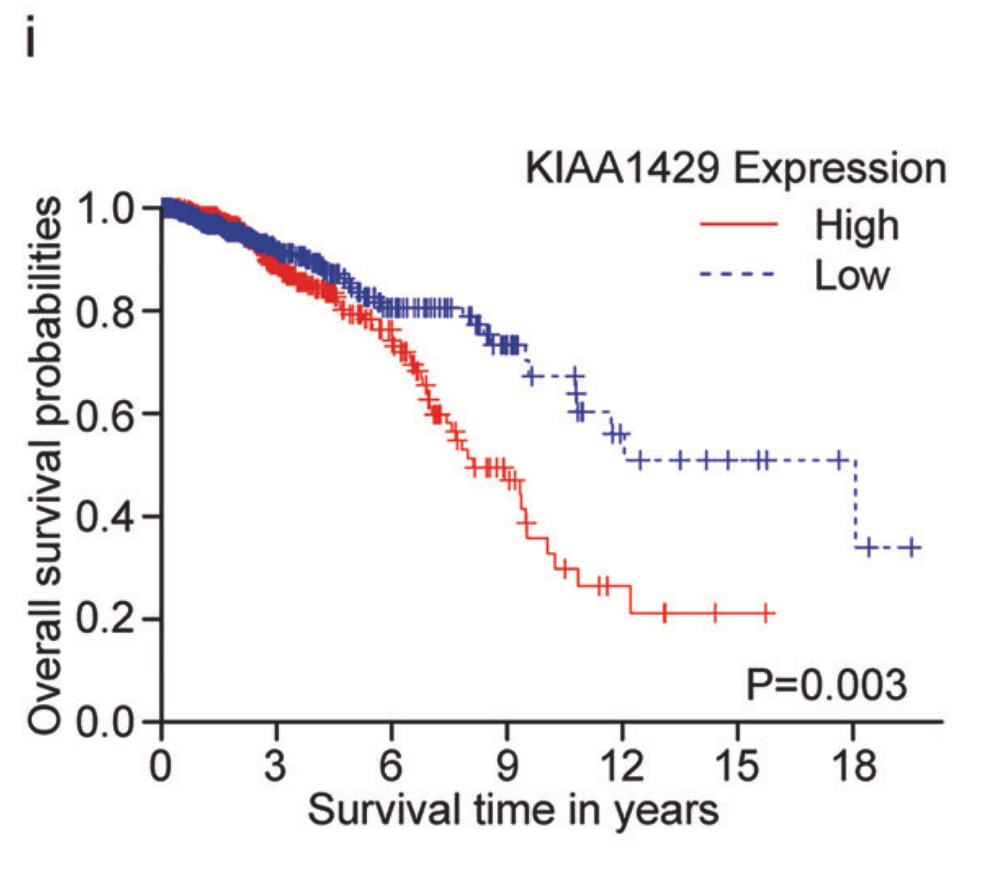
但是我注意到,它里面的时间范围有点长,接近20年,但是我们目前谈论肿瘤病人的生存情况,通常是3年或者5年生存率。如果把这个图时间范围限定一下,很容易看到,在第3年或者第5年,*这个统计学显著性可能是不存在的。
我们该如何计算3年或者5年生存率
统计学检验等数据分析是一回事,如何解释这个结果又是一回事。仅仅是看统计学p值,肯定不行,太多人问:生存分析和Cox回归中,生存曲线最后交叉,如何衡量治疗效果?
比如我就看到有人提问:在生存分析中,如果两组的生存曲线有交叉,书上说Log-rank检验不再适合,同时SPSS软件中的Breslow和Tarone-ware检验也不适合。搜到文献一篇,《生存曲线交叉时统计推断方法的比较和选择》,通过理论分析得出结论,建议用Two-stage方法。但并未详细说明如何进行Two-stage检验。
一个很重要的问题就是如何把影响生存的混杂因素区分出来?
有一个文章就是考虑TCGA数据库的乳腺癌患者的生存分析的10年和20年时间点,
生存分析是一本书书籍的知识点
如果你仅仅是要模仿该文献的生存分析,其实我在生信技能树多次分享过生存分析的细节;
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值
很容易通过R代码,或者网页工具做出来同样的图表,但是生存分析其实是有一本书的知识点,如果想吃透还是蛮难的。

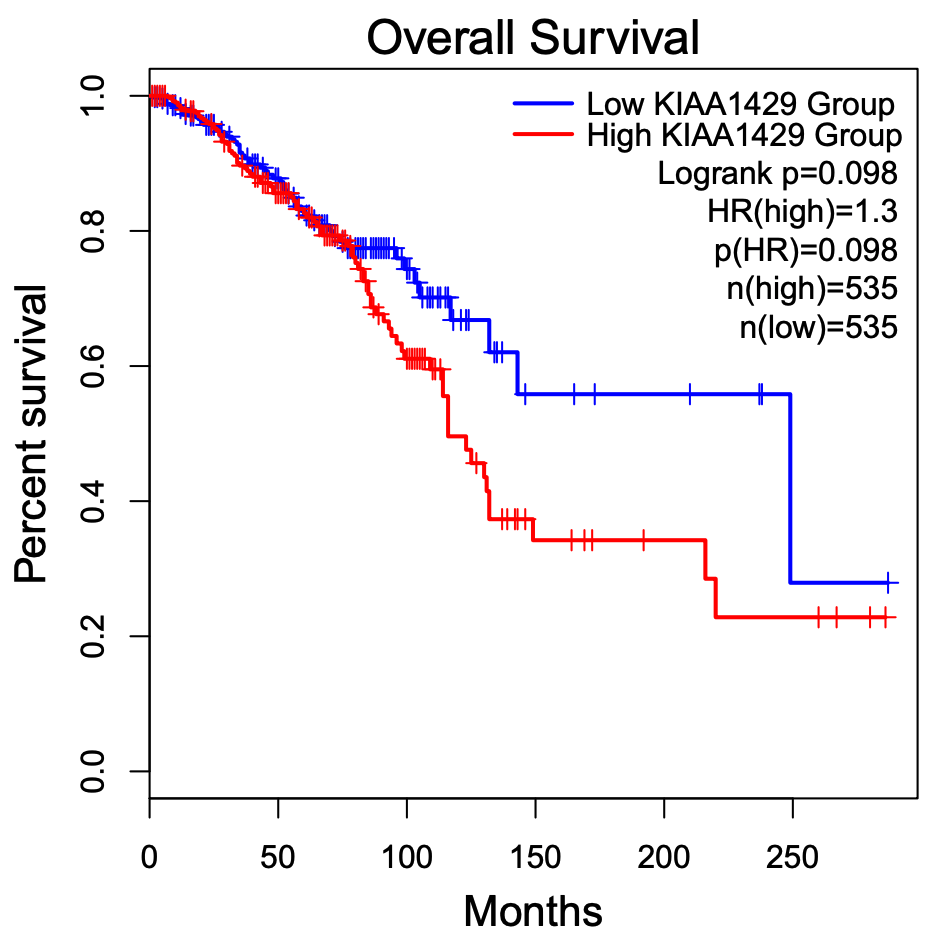
比如gepia2工具
这个工具备受推崇,宣传量也是很足了,简单的输入基因名字,选择癌症,即可出图:
- 网址:http://gepia2.cancer-pku.cn/#survival
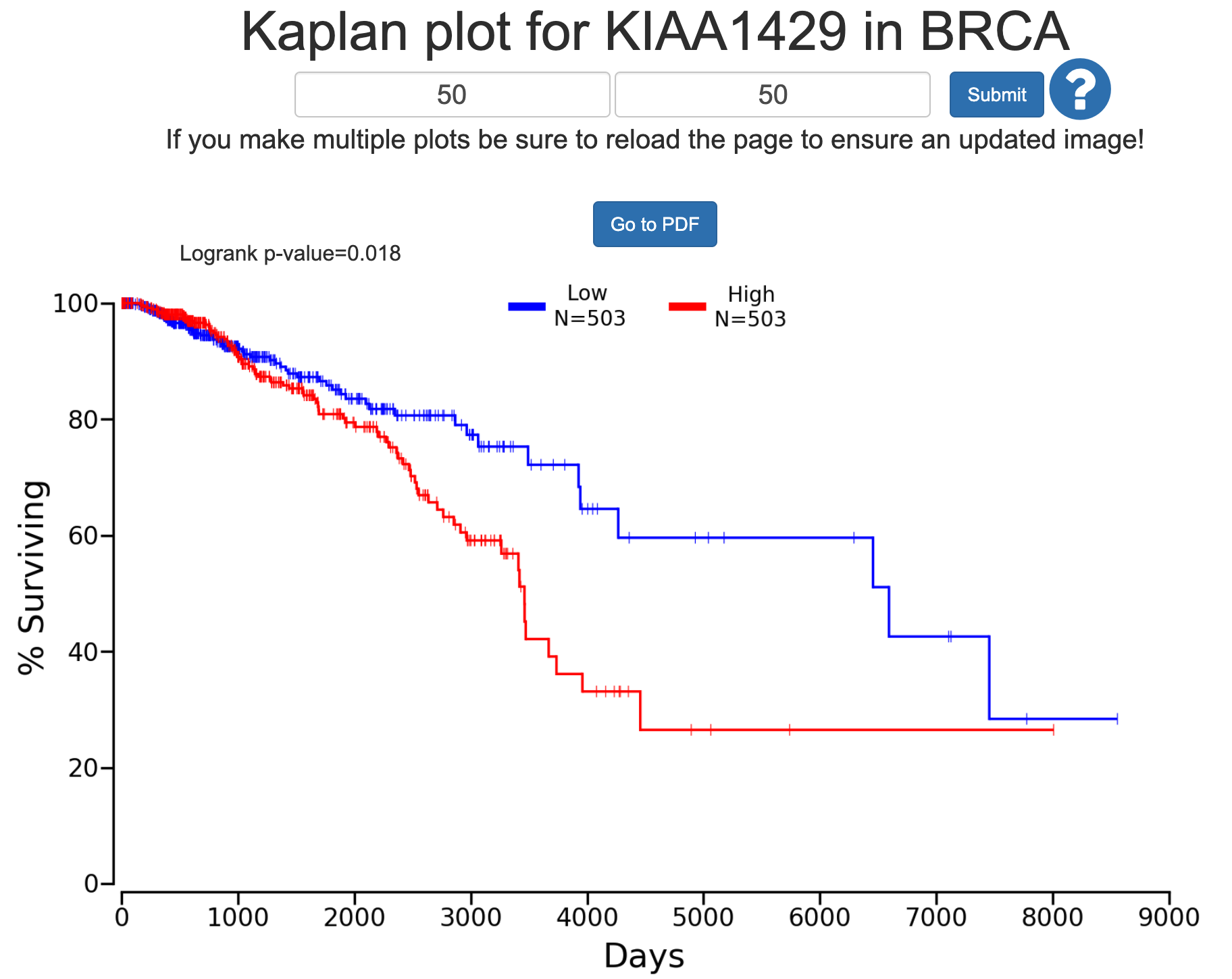
也可以是oncolnc
这个是老牌网页工具了,最开始大家都是推荐它,不知道这些年随着TCGA数据库的临床数据的更新,该工具有没有更新。
- 网址:http://www.oncolnc.org/kaplan/?lower=50&upper=50&cancer=BRCA&gene_id=25962&raw=KIAA1429&species=mRNA
类似的工具实在是太多了,我就不一一截图展示了: - http://tumorsurvival.org/TCGA/Breast_TCGA_BRCA/index.html
甚至都并不一定要TCGA数据库
比如,同样的基因表达量把病人分成两组生存分析,有文章就是自己拿到病人队列,做简单的qPCR实验拿到自己的数据:
- a, KIAA1429 expression in 70 pairs of HCC tissues and adjacent normal tissues from WCH dataset by using qPCR.
- b-c ,Kaplan-Meier analyses of the correlations between KIAA1429 expression and overall survival or disease-free survival of 70 HCC patients.
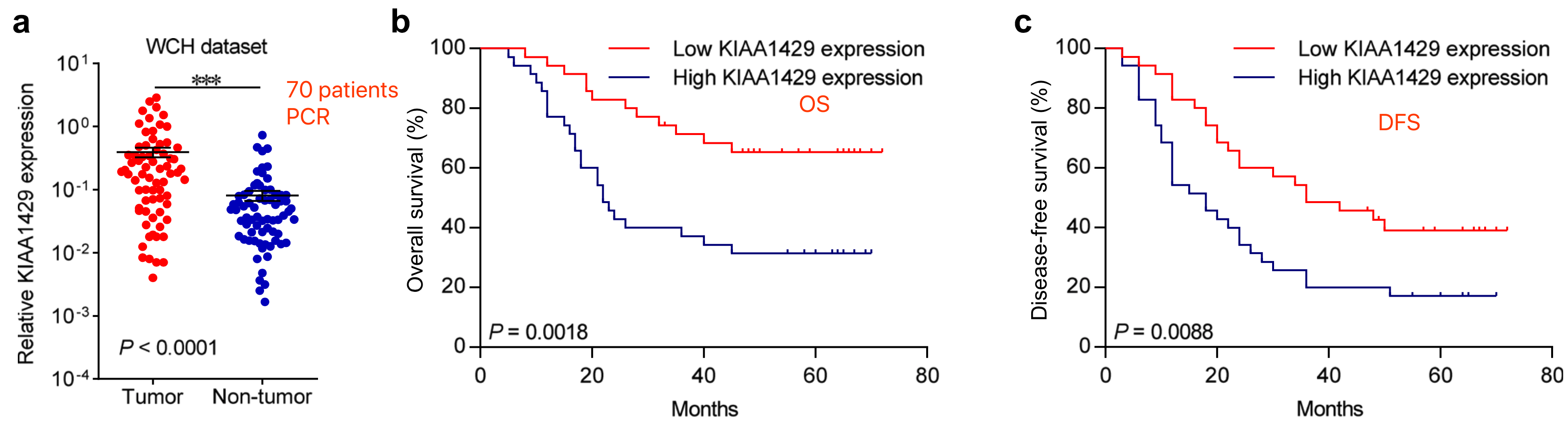
可以看到,其实还有不同时间点的差异,比如OS, PFI, DFI, and DSS,建议大家仔细研读文章《An Integrated TCGA Pan-Cancer Clinical Data Resource to Drive High-Quality Survival Outcome Analytics》,下载其附件:TCGA-CDR (Table S1, tab TCGA-CDR)生存分析公开课
我们前面其实公布了“gsea和wgcna和id转换”钉钉群,但是那个群马上就满了1000人,我们也没办法办法扩大规模,所以只能是新建一个群哈。如果你已经在“gsea和wgcna和id转换”钉钉群,就不用重复加群了哈,我们会把直播课程同步转播在“gsea和wgcna和id转换”群的,多个群联播的!
请大家直接下载钉钉软件,搜索“ID转换和生存分析”群的钉钉群号: 35371384 ,对这几个细节知识点感兴趣的可以加入,我们这个月25号(这周六晚上)八点准时授课。
另外,很多人不熟悉钉钉群加入方式,自行搜索先,不要再留言找不到群,是你自己软件操作有问题!文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班: - 数据挖掘学习班第5期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第7期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?