前些天我和我的学徒们在生信技能树分享了教程:人人都可以学会生存分析(学徒数据挖掘),提到根据公共数据库(比如TCGA)的信息来检查具体的某个或者某几个基因的生存意义已经是非常简单了,只需要很简单的数据库网页工具认知,或者一点点代码能力。
但是今天逛朋友圈发现了一个超级神器,德克萨斯大学MD安德森癌症中心梁晗教授课题组开发了一个在线网页工具:DrBioRight,” a natural language-oriented, artificial intelligence (AI)-driven omics data analysis platform (https://drbioright.org). 该文章于2020年9月24见刊在Cancer Cell杂志,文章题目是《Next-Generation Analytics for Omics Data》。居然是聊天的方式做生存分析。
DrBioRight的3大功能区
我们直接进入DrBioRight的主页: (https://drbioright.org). 如下所示:
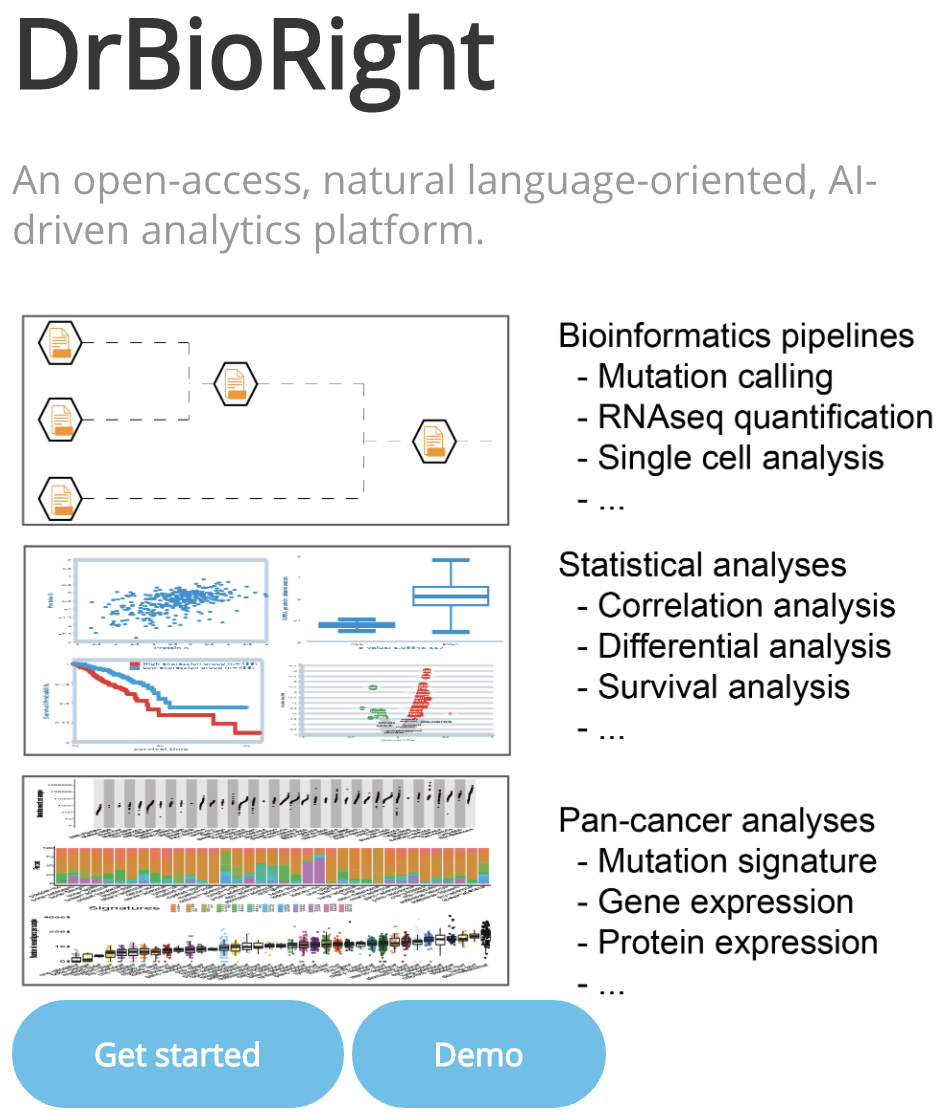
从该工具主页可以看到,它里面集成了常见的生物信息学数据处理流程,我感觉在不久的将来肯定会囊括我在b站的74小时生信工程师教学视频合辑:
- 免费视频课程《RNA-seq数据分析》
- 免费视频课程《WES数据分析》
- 免费视频课程《ChIP-seq数据分析》
- 免费视频课程《ATAC-seq数据分析》
- 免费视频课程《TCGA数据库分析实战》
- 免费视频课程《甲基化芯片数据分析》
- 免费视频课程《影像组学教学》
- 免费视频课程《LncRNA-seq数据》
- 免费视频课程《GEO数据挖掘》
然后,它基本上把基于R语言的统计可视化全部搞定了,相关性分析,生存分析,差异分析都不在话下。正常情况下,从来没有编程经验的小伙伴要把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
起码需要一个月!!!详见:《生信分析人员如何系统入门R(2019更新版)》
最后,它集成了大量的数据库资源,包括TCGA, GTEX, CCLE等等,基本上大家无需下载它们的源数据文件,就可以在它的网页上面进行各式各样的探索。TCGA数据库是目前最综合最全面的癌症病人相关组学数据库,包括:
- DNA Sequencing
- miRNA Sequencing
- Protein Expression array
- mRNA Sequencing
- Total RNA Sequencing
- Array-based Expression
- DNA Methylation
- Copy Number array
知名的肿瘤研究机构都有着自己的TCGA数据库探索工具,比如:
- Broad Institute FireBrowse portal, The Broad Institute
- cBioPortal for Cancer Genomics, Memorial Sloan-Kettering Cancer Center
但是你现在有了DrBioRight,一站式搞定全部资源。
使用DrBioRight做生存分析
DrBioRight目前还不是万能的,它的核心卖点就是聊天对话式窗口,其文章举的例子就是:“perform survival analysis in breast cancer on TP53 gene expression” ,我也简单测试了一下, 结果如下:
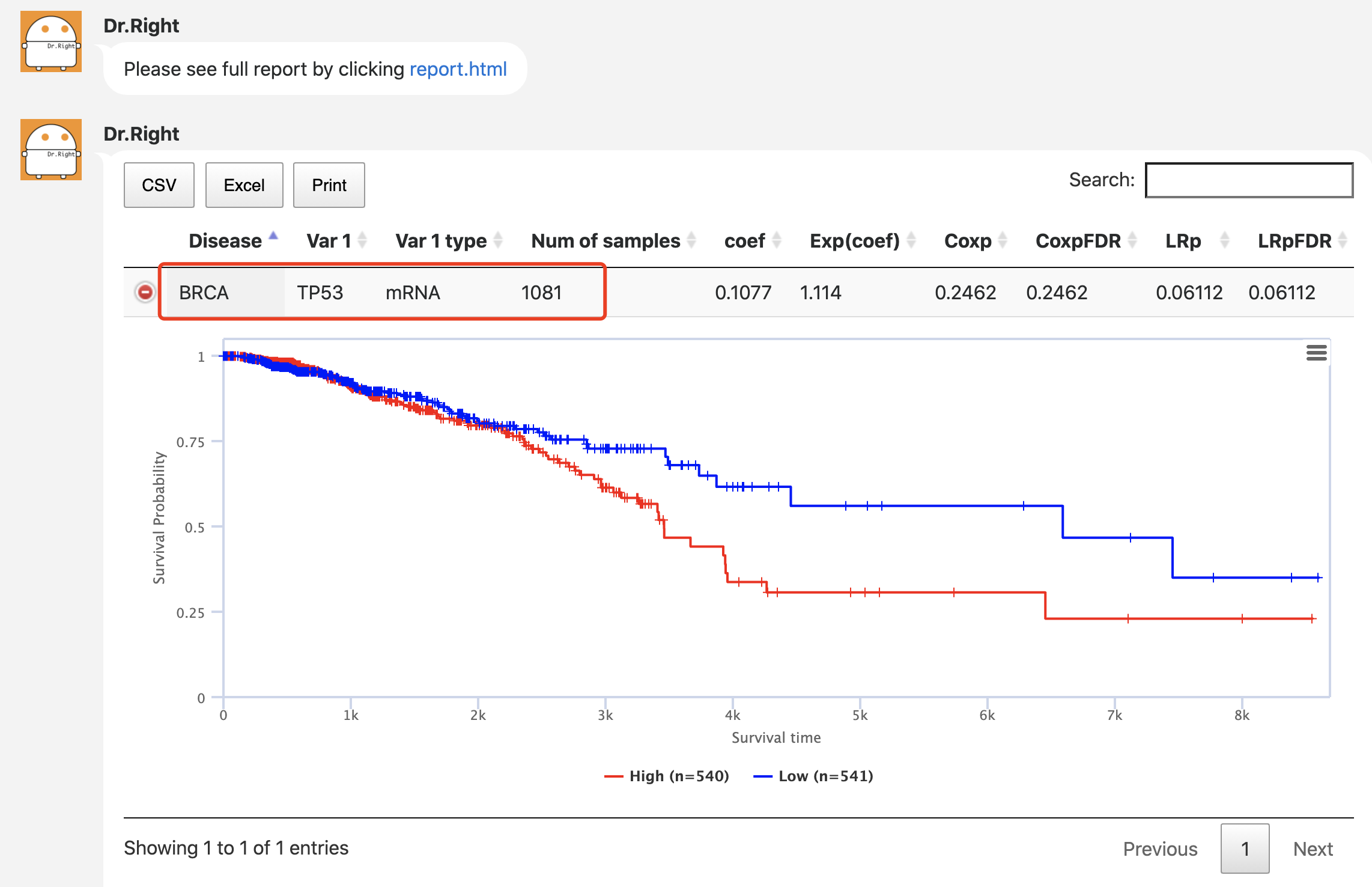
最让我喜欢的功能是,它还附带了一个临时报告:https://drbioright.org/reports/5f7d85a4f8a91ff1c46388e8/report.html 打开后惊喜满满~~
不过生存分析何止是这样的简单查询呢,如果你确实想把生存分析学会而且学好,我在生信技能树多次分享过生存分析的细节也值得你认真读完;
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值
“ID转换和生存分析”群的钉钉群号: 35371384,如果你下载钉钉软件,搜索进群这个能力都没有,我还是建议你放弃学生信哈!
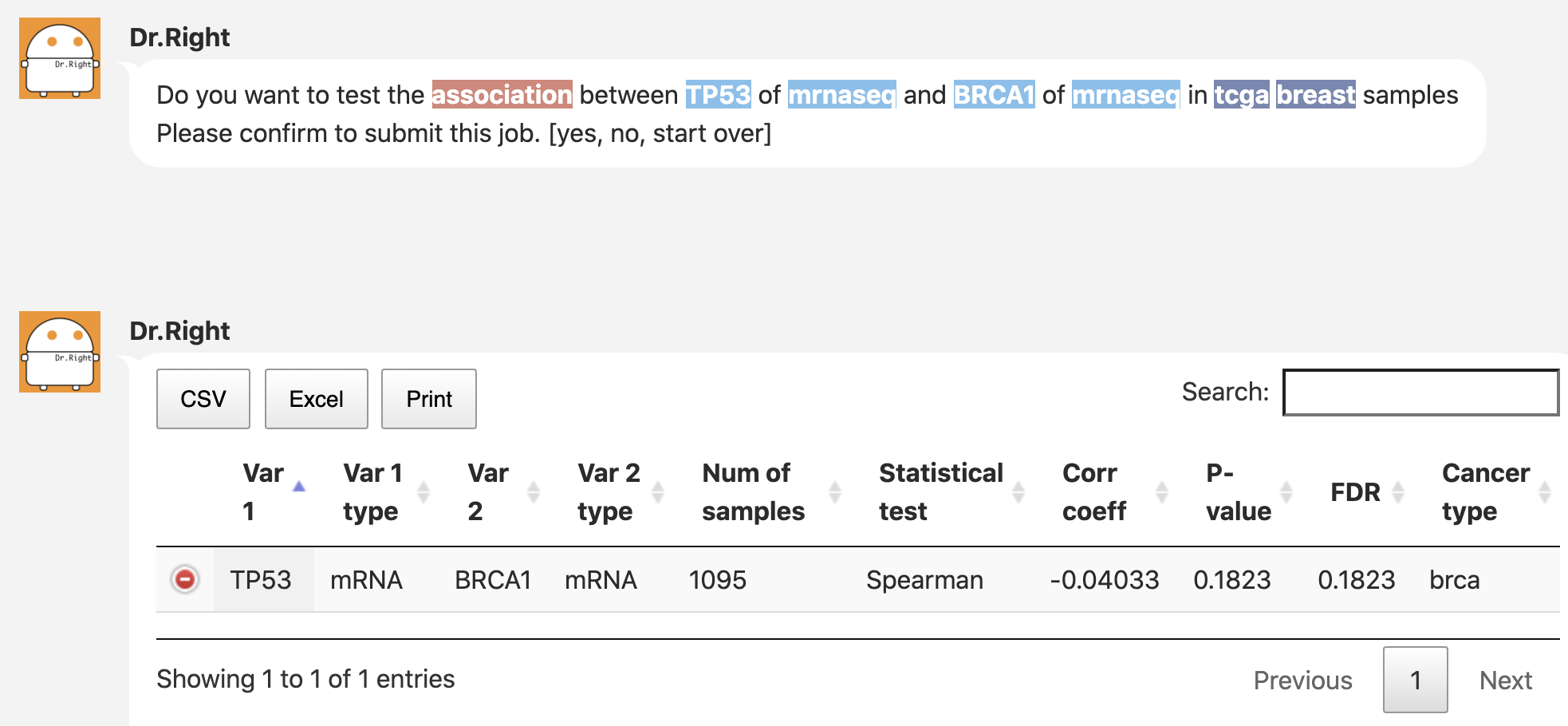
然后我不停的调试这个对话窗口,其实很容易理解它的工作原理,它就是检测我输入语句的关键词而已:
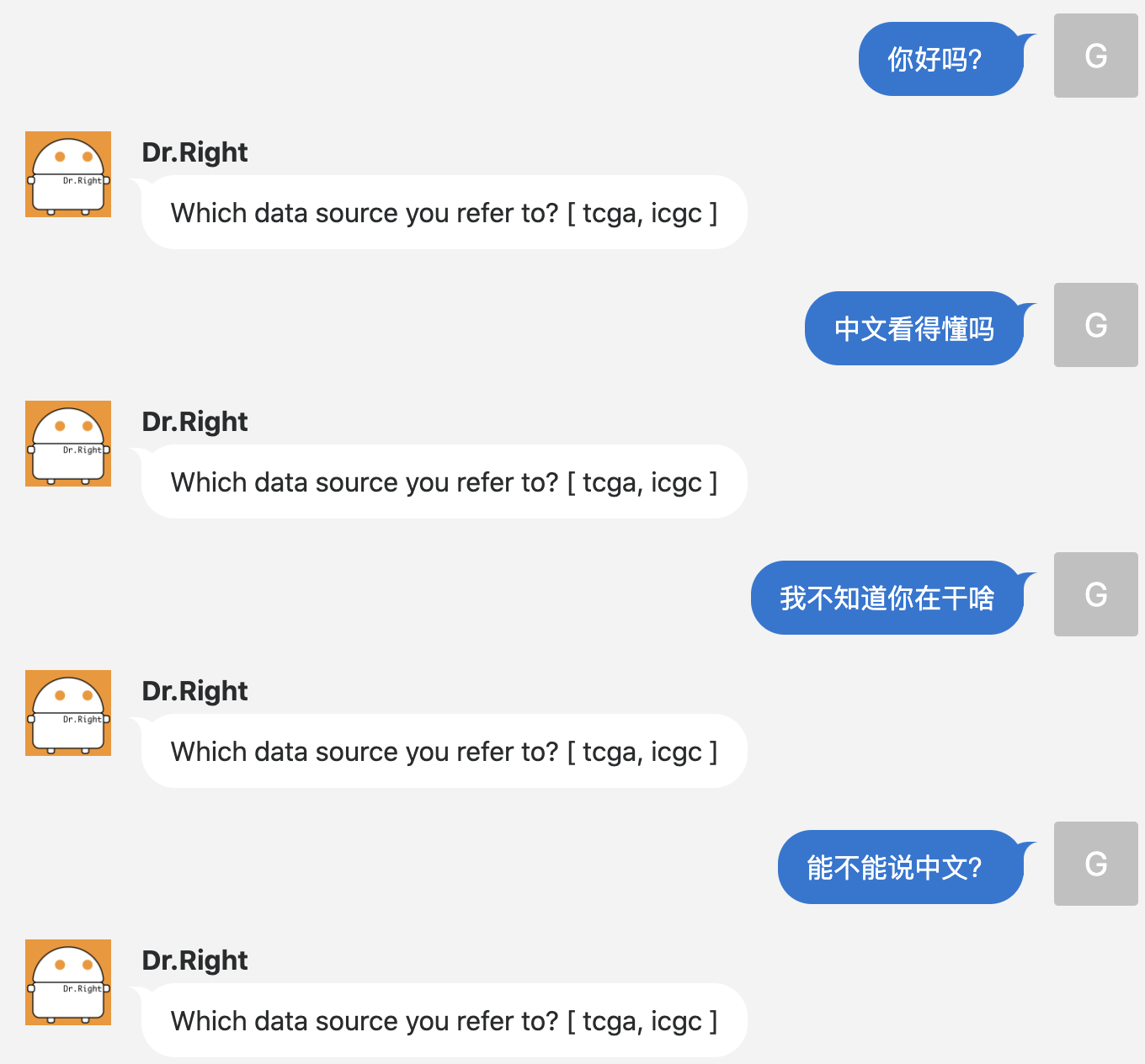
也顺便测试了一下,发现它不能识别中文:
我喜欢把TCGA数据库的应用划分为8个领域:
- 1、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)的预后(生存曲线)
- 2、探索各类肿瘤与对照的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(箱线图)
- 3、探索各类肿瘤与对照的全局(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(差异分析流程)
- 4、探索各类肿瘤中两个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平相关性(散点图)
- 5、探索各类肿瘤中多个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平总结(热图)
- 6、探索各类肿瘤中单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)与所有其它分子相关性并且排序
- 7、探索各类肿瘤中单个基因突变或者单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的预后(生存曲线)
- 8、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)分组后的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)特性的分布
大家有空可以测试一下DrBioRight工具 (https://drbioright.org)是否可以全部完成这些需求了?
DrBioRight未来可期
如果仅仅是一个基于自然语言处理的网页工具,再加上一点人工智能的噱头,就太小儿科了。DrBioRight的开发团队目前已经完成了转录组数据处理,以及一些单细胞数据分析图表绘制的功能。
For example, a user can start an analysis by simply asking, “Could you do an RNA-seq analysis?” and then provide the source or location of the raw data (e.g., an SRA ID). Through a dialog with DrBioRight, the user can finish the entire analysis step by step, including quality control, read mapping, gene expression quantification, differential expression analysis, and gene set enrichment analysis.
但是数据分析从来不是瓶颈
这10年,NGS基本上变革了传统的分子生物学研究,大家遇到的问题既不是测序费用高昂,也不是测序数据无法分析,反而是数据分析结果无法结合到自己的课题。
直白一点说,给你生存分析结果图表,你看不懂!
前面的转录组数据全部给你处理了,quality control, read mapping, gene expression quantification, differential expression analysis, and gene set enrichment analysis 一站式流程全部搞定,图表给你,你能看懂吗?
认真学习其中的细节还是很有必要的,如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班马上10月12日就开课):
- 数据挖掘学习班第7期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第9期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?
号外:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!