生信技能树联盟创始人jimmy手把手带你完成一个GEO数据库(GSE76275)挖掘实例,从必备R包安装,表达矩阵下载,PCA/boxplot/heatmp的数据检查,探针ID到基因ID的转换,根据生物学分组进行差异分析并且绘制火山图、热图,还有简单的超几何分布检验的KEGG等数据库注释结果。也有一点表达矩阵进行GSEA/GSVA分析,以及WGCNA分析。最后还根据TNBC(三阴性乳腺癌)的生物学特征提取指定基因的表达量,使用genefu这个R包进行PAM50分类。
视频学习方式
直接B站咯:https://m.bilibili.com/video/BV1dy4y1C7jz
下载TCGA数据库的BRCA的芯片表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。
- 代码在:https://github.com/jmzeng1314/TCGA_BRCA
下载TCGA数据库的BRCA的RNA-seq表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。 - 代码在:https://github.com/jmzeng1314/TCGA_BRCA
下载GTEx数据库的RNA-seq表达矩阵,并且提取其中属于breast的样本,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。 - 代码在: https://github.com/jmzeng1314/gtex_BRCA
下载METEBRIC数据库的RNA-seq表达矩阵,同样进行PAM50分类,结合临床信息,并且对应比较GSE76275数据集。 - 代码在:https://github.com/jmzeng1314/METABRIC
首先需要安装必备R包
需要自行下载学习R语言及熟练使用Rstudio编辑器,根据课程配套代码安装R包。对初学者来说,尤其是中国大陆的学生,安装R包,最重要的是切换镜像,代码如下:
options(BioC_mirror="https://mirrors.tuna.tsinghua.edu.cn/bioconductor/") options("repos" = c(CRAN="http://mirrors.cloud.tencent.com/CRAN/")) options(download.file.method = 'libcurl') options(url.method='libcurl') BiocManager::install("miRNAtap",ask = F,update = F) BiocManager::install("topGO",ask = F,update = F) BiocManager::install("miRNAtap.db",ask = F,update = F)另外:r全套资料都是在B站,需要提前学完哦!
第一步:下载表达矩阵
首先需要了解GEO数据库,建议通读: 解读GEO数据存放规律及下载,一文就够
然后根据课件代码使用GEOquery包进行下载,详细教程见:从GEO数据库下载得到表达矩阵 一文就够
示例数据集:GEO数据库(GSE76275) 对应的3篇SCI文章见课程附件。(3篇文章都需要解读)第三步:检查表达矩阵
首先根据全局基因表达绘制PCA图如下:
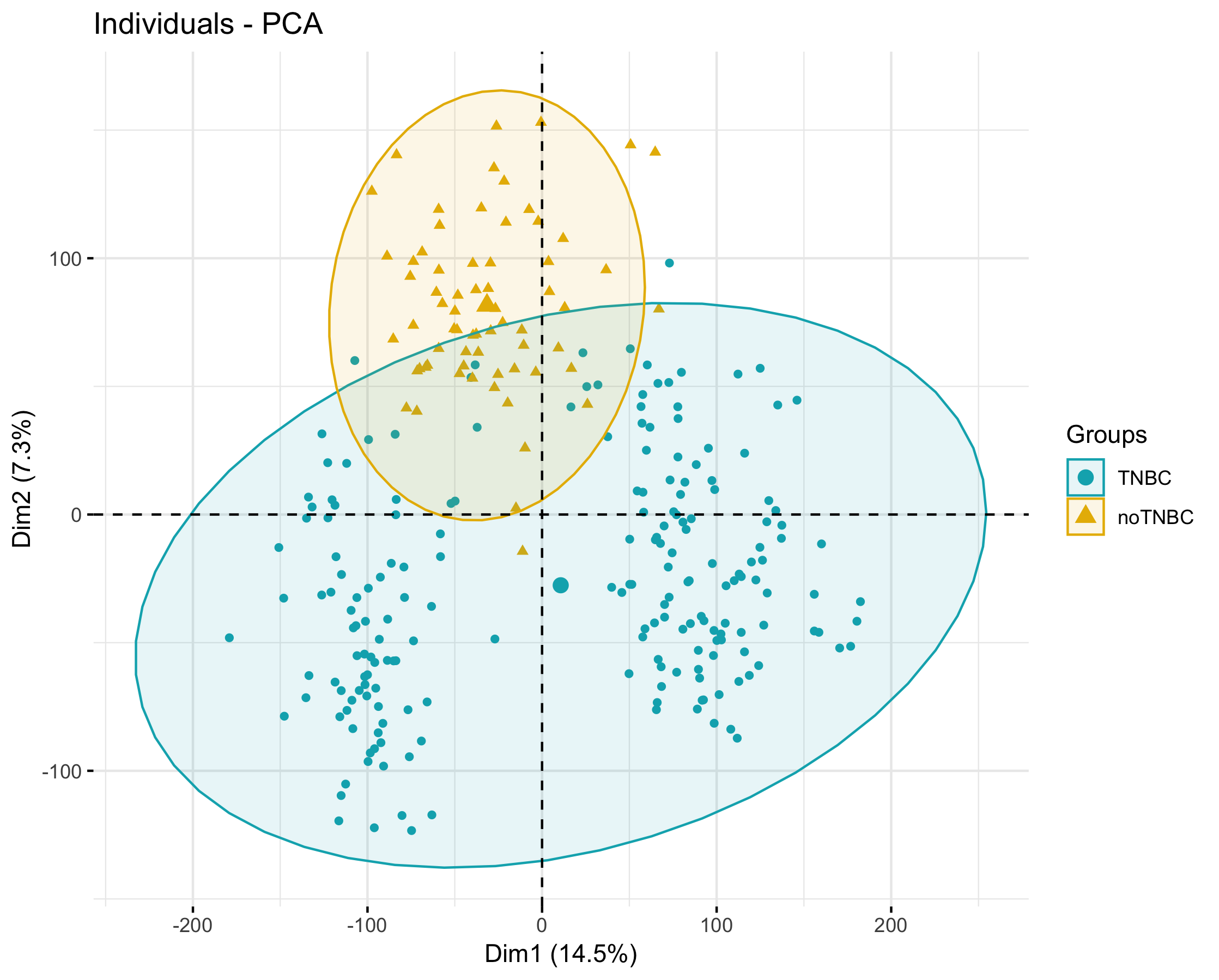
代码见课程附件,具体参数视频里有介绍,结果很容易理解,可以看到那些不属于TNBC的样本跟TNBC组有着很清晰的界限,而且可以看到TNBC组本身也有着界限分明的两列,反映了TNBC的异质性:
还有top 1000的sd的基因提取出来绘制热图:
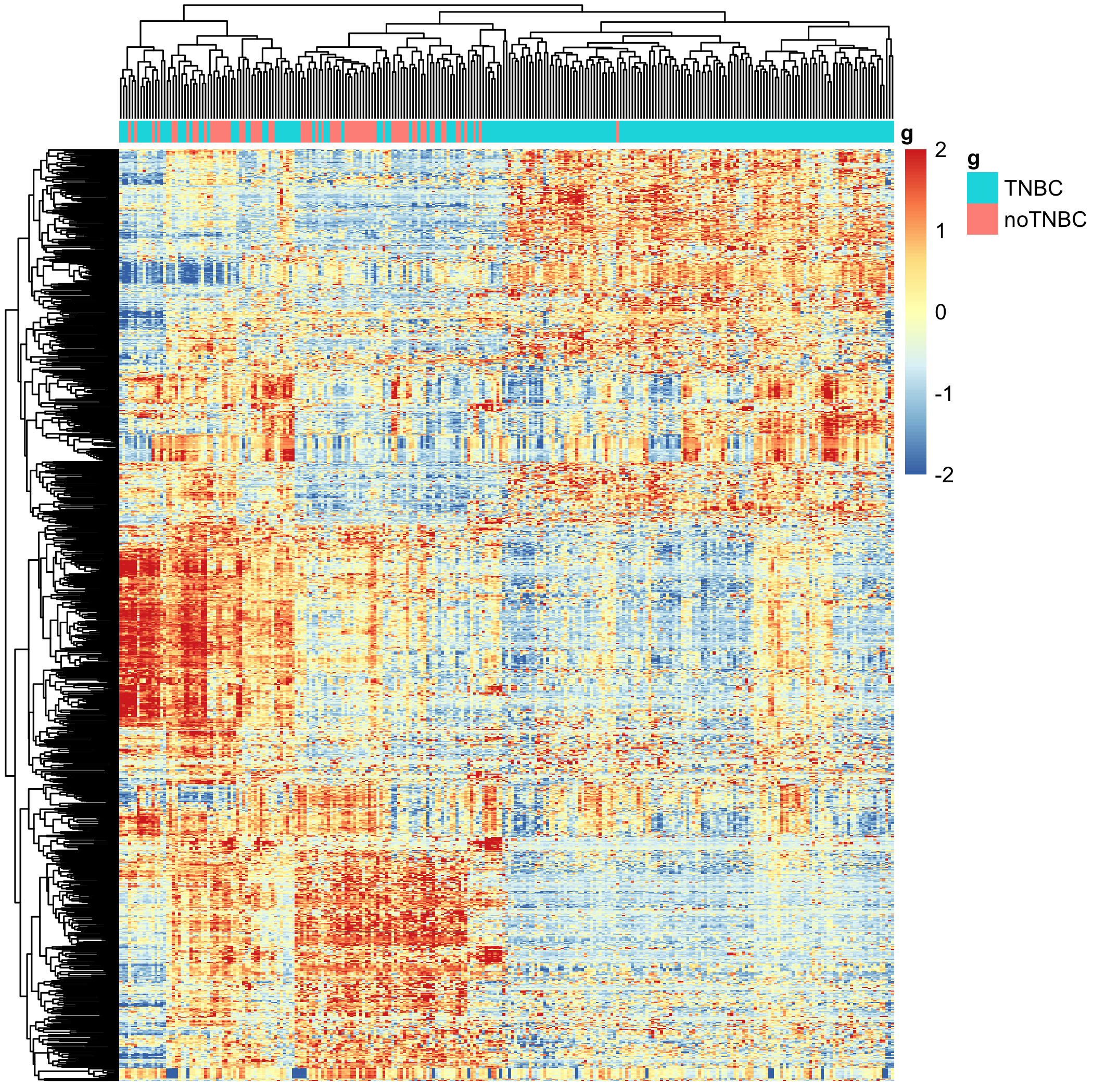
这个结果跟PCA分析结果相呼应!更多图表都在课程的配套代码里面;第4步:差异分析
这里走标准的limma流程,详细推文介绍:根据分组信息做差异分析- 这个一文不够的
第5步:GO/KEGG数据库注释
得到的差异分析结果,也可以走标准的火山图,热图,GO/KEGG数据库注释,见推文:差异分析得到的结果注释一文就够
第6步:GSEA/GSVA分析
代码在GitHub哈:https://github.com/jmzeng1314/GSE76275-TNBC
TCGA数据库挖掘
代码在:https://github.com/jmzeng1314/TCGA_BRCA
GTEx数据库挖掘
代码在:https://github.com/jmzeng1314/gtex_BRCA
METABRIC数据库挖掘
代码在:https://github.com/jmzeng1314/METABRIC
增加WGCNA流程代码
对表达矩阵挑选top5000的MAD基因,以及top10000后,分别独立走WGCNA流程看结果。
背景知识参考:https://github.com/jmzeng1314/my_WGCNA文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班: - 数据挖掘学习班第7期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第9期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?
号外:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!另外,:绝大部分生信技能树粉丝都没有机会加我微信,已经多次满了5000好友,所以我开通了一个微信好友,前100名添加我,仅需150元即可,3折优惠期机会不容错过哈。我的微信小号二维码在:0元,10小时教学视频直播《跟着百度李彦宏学习肿瘤基因组测序数据分析》