在朋友圈看到了有人吐槽她下载的表达矩阵里面出现日期基因,挺好玩的,就把gse号码要过来了,是 GSE122083,其日期基因如下:
我看了看 GSE122083 数据集背后的文献,居然是单细胞哦!
rm(list=ls())
options(stringsAsFactors = F)
library(Seurat)
a=read.table('GSM3454528_naive_cells.txt.gz',header = T)
a[1:4,1:4]
raw.data=a[,-1]
rownames(raw.data)=a[,1]
得到如下的报错:
> dim(a)
[1] 18413 3516
Error in `.rowNamesDF<-`(x, value = value) :
duplicate 'row.names' are not allowed
In addition: Warning message:
non-unique values when setting 'row.names': ‘2-Mar’, ‘CCDC7’, ‘CYB561D2’, ‘LINC01422’, ‘LINC01481’, ‘MATR3’, ‘RGS5’, ‘TMEM256-PLSCR3’
我实在是很难理解, 3500多个细胞已经是 3500多列的矩阵,作者怎么就敢使用Excel打开,不怕电脑奔溃吗?
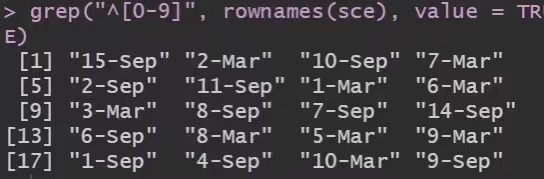
也就是说,不到两万个基因,居然是也有基因名字重复了。而且2-Mar这个日期基因赫然在列。
这么多日期基因可怎么办哦
> grep("^[0-9]",a[,1],value = T)
[1] "15-Sep" "2-Mar" "10-Sep" "7-Mar" "2-Sep"
[6] "11-Sep" "1-Mar" "6-Mar" "3-Mar" "8-Sep"
[11] "7-Sep" "14-Sep" "6-Sep" "8-Mar" "5-Mar"
[16] "9-Mar" "1-Sep" "4-Sep" "10-Mar" "9-Sep"
[21] "2-Mar"
自己构建日期基因对应表格
首先去下载 gtf文件:
这个 gencode.v36.annotation.gtf.gz 文件也就是不到50M,所以很快就下载完毕,然后使用下面的代码格式化:
zcat gencode.v36.annotation.gtf.gz | grep -v '_PAR_Y' |\
perl -alne '{next unless $F[1] eq "HAVANA";next unless $F[2] eq "gene";/gene_id \"(.*?)\.\d+\"; gene_type \"(.*?)\"; gene_name \"(.*?)\"/;print "$3\t$2\t$1\t$F[0]\t$F[3]\t$F[4]"}' \
> HAVANA_v36_human_gene_info
zcat gencode.v36.annotation.gtf.gz | grep -v '_PAR_Y' |\
perl -alne '{next unless $F[1] ne "HAVANA";next unless $F[2] eq "gene";/gene_id \"(.*?)\.\d+\"; gene_type \"(.*?)\"; gene_name \"(.*?)\"/;print "$3\t$2\t$1\t$F[0]\t$F[3]\t$F[4]"}' \
> ENSEMBL_v36_human_gene_info
得到了 基因信息文件,使用Excel打开,自己主动把基因转换好!
当然了,其实绝大部分情况下没有必要如此大费周章啦,删除即可,绝大部分基因都不一定是那么的重要!
你看文献本身就删除了很多基因,而且仅仅是选取了top5000的 高变基因 :

学徒作业
完成这两个10x样品的基础分析,各自独立的聚类分群和注释:
参考前面的例子:人人都能学会的单细胞聚类分群注释 ,同时结合文献的信息:
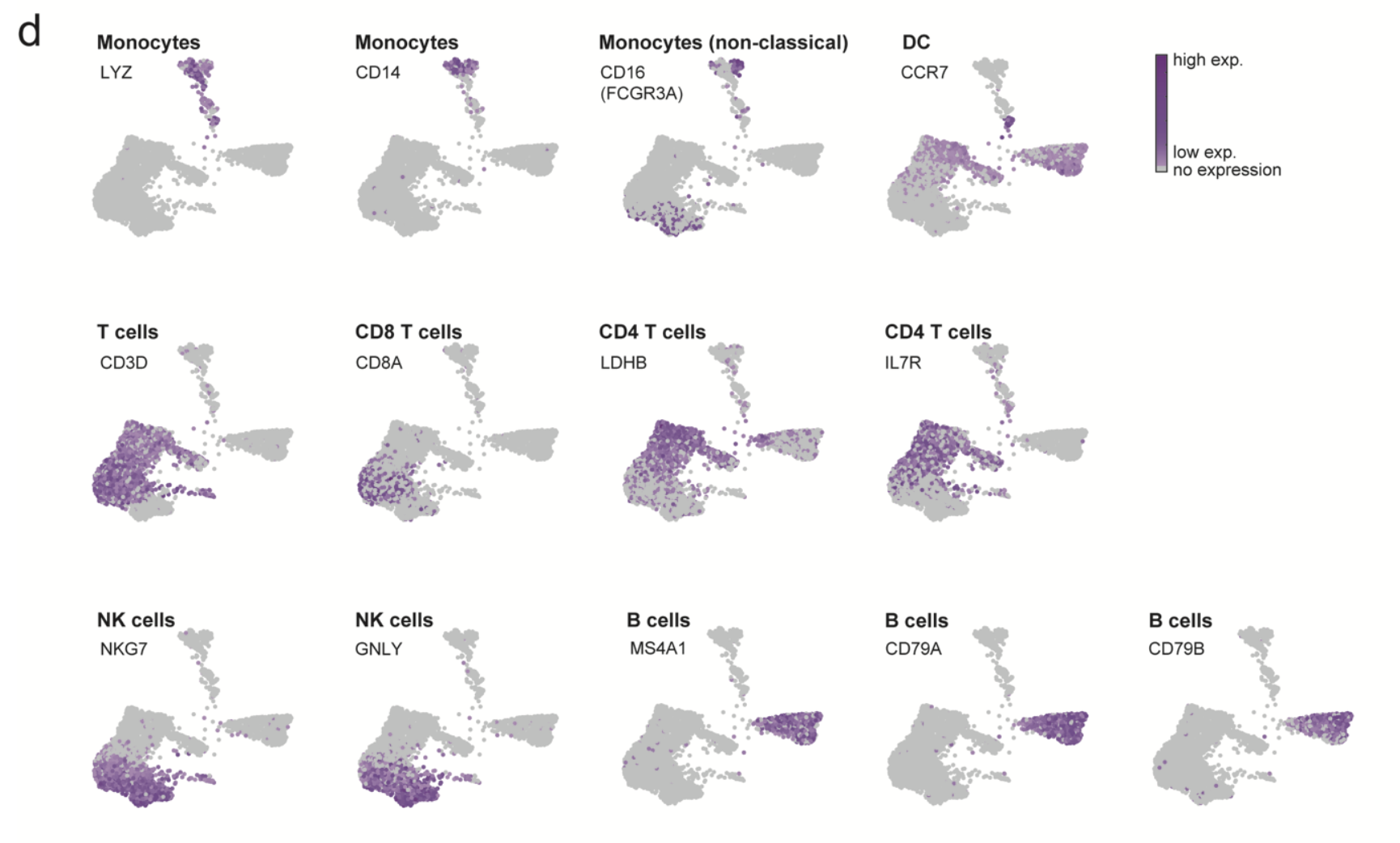
- NK (NKG7 and GNLY),
- NKT (CD3D and NKG7),
- CD8 T cells (CD3D and CD8A),
- CD4 T cells (CD3D,LDHB and IL7R),
- B cells (MS4A1, CD79A and CD79B),
- monocytes (LYZ and CD14 and/or CD16),
- DC (LYZ and CCR7)
这些不同细胞亚群的标记基因分别可视化如下: