于2021年3月发表在CELL杂志的文章, 标题是:《In vivo CD8+ T cell CRISPR screening reveals control by Fli1 in infection and cancer》,链接是:https://doi.org/10.1016/j.cell.2021.02.019
首先是RNA-seq数据
链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE149838
GSM4514055 RNA-seq_Fli1KO_rep1
GSM4514056 RNA-seq_Fli1KO_rep2
GSM4514057 RNA-seq_Fli1KO_rep3
GSM4514058 RNA-seq_Fli1KO_rep4
GSM4514059 RNA-seq_Fli1KO_rep5
GSM4514060 RNA-seq_WT_rep1
GSM4514061 RNA-seq_WT_rep2
GSM4514062 RNA-seq_WT_rep3
非常简单而且常规的差异分析,图表如下所示:
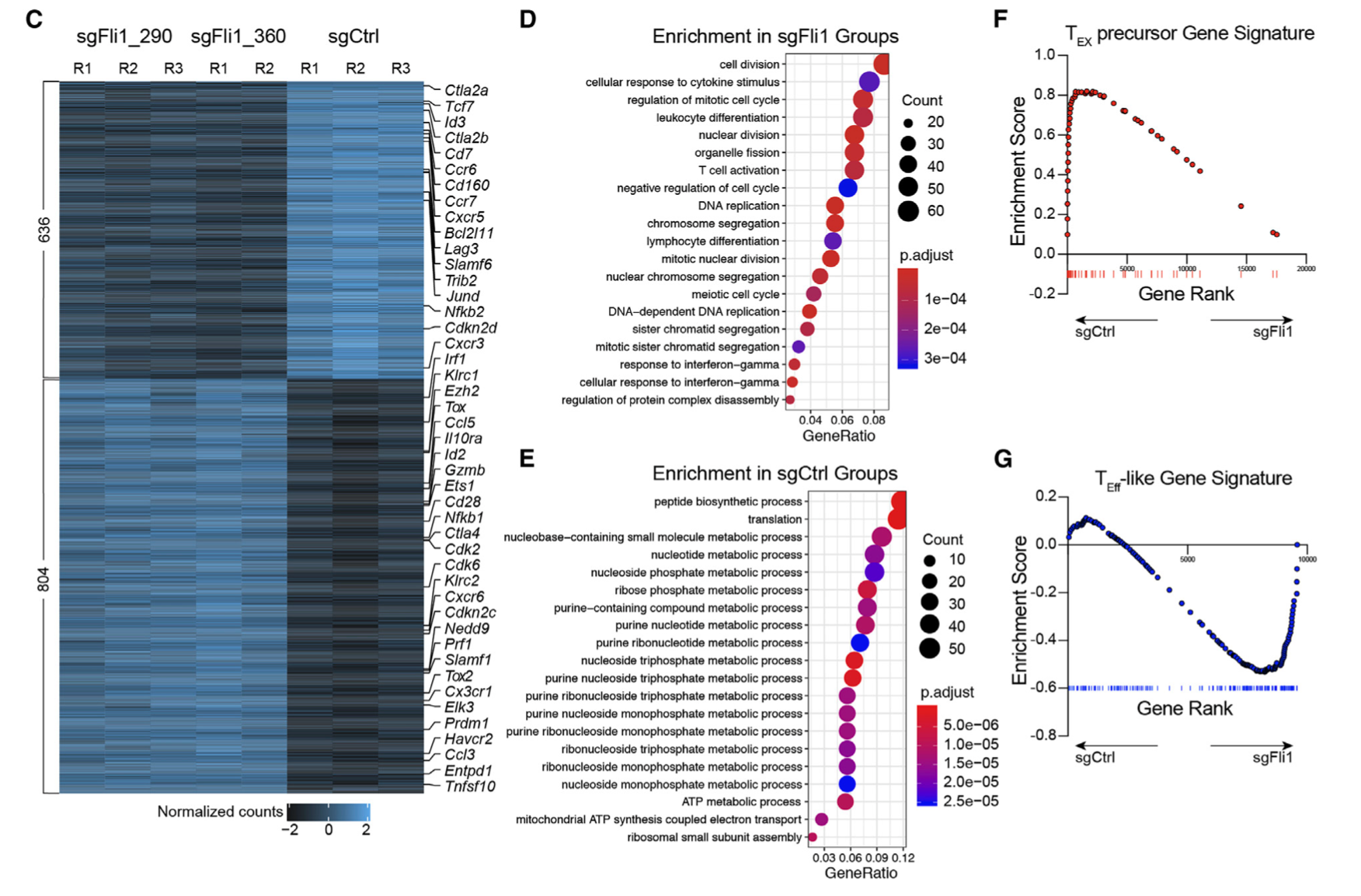
常规的差异分析呢,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
这个GSE149838是提供表达量矩阵文件的,大家可以自行下载这个 GSE149838_Zeyu_Fli1koRNAseq_rawcounts.csv.gz 文件,然后走一下我们的转录组流程,看看能不能拿到同样的生物学意义的图表!
然后是ATAC-seq数据
链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE149836
GSM4514043 ATAC-seq_Fli1KO_rep1
GSM4514044 ATAC-seq_Fli1KO_rep2
GSM4514045 ATAC-seq_Fli1KO_rep3
GSM4514046 ATAC-seq_Fli1KO_rep4
GSM4514047 ATAC-seq_WT_rep1
GSM4514048 ATAC-seq_WT_rep2
首先呢也是简单的差异分析,重点是 differentially accessible (DA) peaks 而不是前面的RNA-seq数据的差异表达基因:
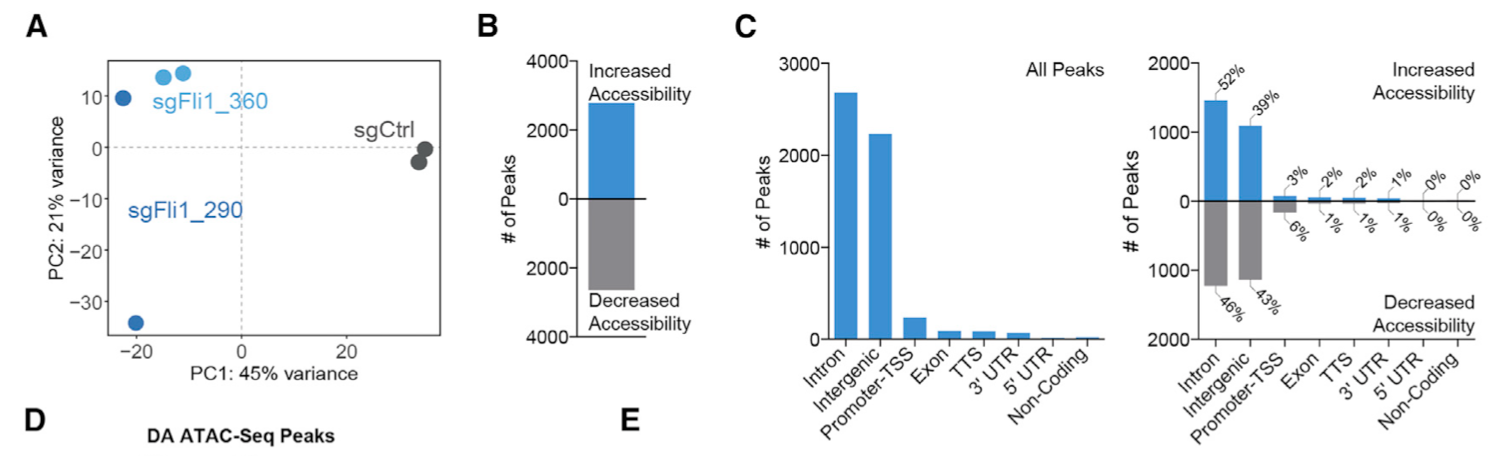
然后同样的有热图:
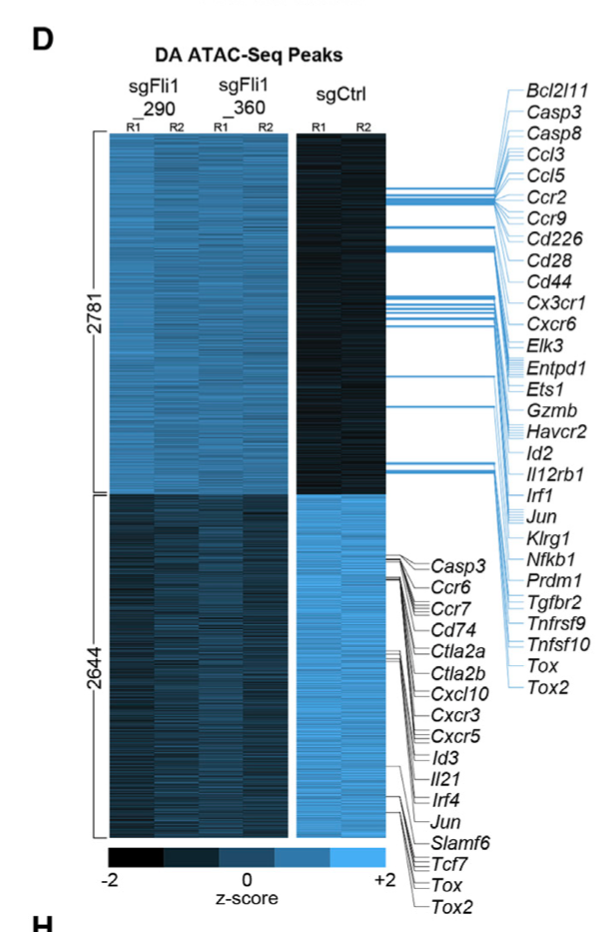
(D) Heatmap shows differentially accessible peaks between sgCtrl group and 2 sgFli1 groups (adjusted p value <0.05, log10 fold change >0.6). Selected genes assigned to the peaks are indicated.
因为这个数据集提供了peaks的bed文件,所以直接读入R里面进行数据分析即可哈!
GSM4514043_290_1_S4_peaks.bed.gz 529.0 Kb
GSM4514044_290_2_S5_peaks.bed.gz 460.6 Kb
GSM4514045_360_1_S6_peaks.bed.gz 535.0 Kb
GSM4514046_360_2_S7_peaks.bed.gz 495.8 Kb
GSM4514047_R_1_S2_peaks.bed.gz 456.6 Kb
GSM4514048_R_2_S3_peaks.bed.gz 442.7 Kb
文章里面提供了详细的软件和R包,比较容易follow的,如果确实有困难也可以考虑看我的视频:
- 视频免费在B站,https://www.bilibili.com/video/BV1C7411C7ez 大家学习的时候记得发弹幕交流哈
- 同步查看视频配套代码 :https://www.jianshu.com/p/5bce43a537fd
- ATAC-SEQ实战演练的素材 链接:https://share.weiyun.com/5rYmPT1 密码:dr3ub6 包括一些公司PPT,综述以及文献。测试数据下载方式也是在里面了。
- ATAC-SEQ 实战演练的思维导图:文档链接:https://mubu.com/doc/2DG1mC2kdg 密码:rf2n
- 学徒学习笔记:https://mp.weixin.qq.com/s/7wNRrpkqcuQmJ7ASlpytqw
最后是RNA-seq和ATAC-seq数据整合分析
首先ATAC-seq数据差异分析拿到的 differentially accessible (DA) peaks 可以去对应到基因组的基因,然后RNA-seq数据通常就有差异表达基因,两个基因集就可以取交集,做韦恩图:
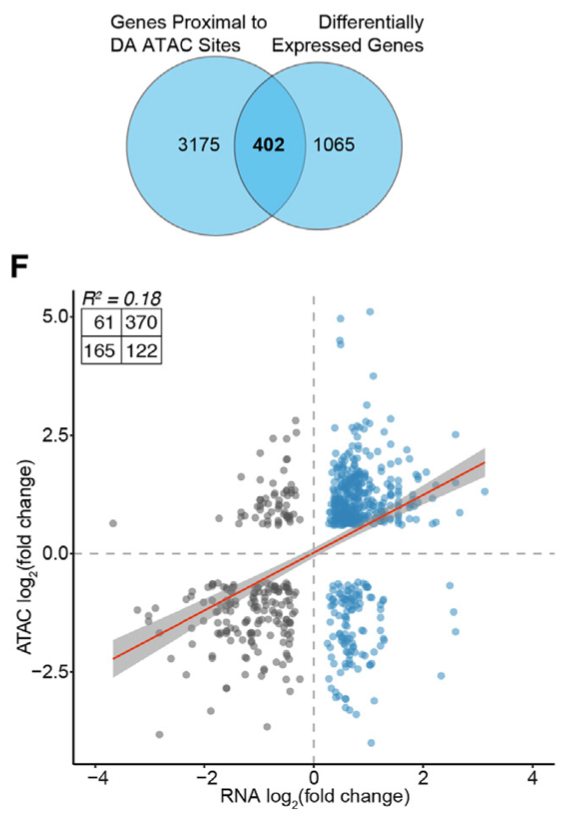
可以看到,这个图里面并没有秀全部的基因,仅仅是差异的那些,RNA-seq和ATAC-seq数据各自的差异都有自己的流程和阈值,两个联合起来就是散点图啦!
这样的RNA-seq和ATAC-seq数据整合分析最近五年(2017-2021)很流行,比如,标题是:《Early chromatin shaping predetermines multipotent vagal neural crest into neural, neuronal and mesenchymal lineages》,链接是:https://www.nature.com/articles/s41556-019-0428-9.pdf,也有:
再比如2017年发表的来自美国Jackson基因组医学实验室Jacques Banchereau团队的文章《The chromatin accessibility signature of human immune aging stems from CD8+ T cells》,描述了衰老过程中染色质动态变化的特征,发现了关键的基因及其调控特征。研究团队招募了51例健康年轻组(年龄22-40岁)与26名年龄大于65岁的健康老年组,利用ATAC-seq和RNA-seq发现了两组间显著差异的基因。文章是J Exp Med. 2017 Oct,doi: 10.1084/jem.20170416 ,数据在:https://ega-archive.org/datasets/EGAD00001003602
类似的性别+衰老,还有2020的NC文章《Sexual-dimorphism in human immune system aging》,也是 RNA-seq和ATAC-seq数据整合!
RNA-seq和ATAC-seq数据就足够了吗
RNA-seq和ATAC-seq数据固然是从两个层面来说明了问题,但是研究者这个时候还做了 Cut and Run chipseq
链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE149837
GSM4514049 CnR_Fli1_rep1
GSM4514050 CnR_Fli1_rep2
GSM4514051 CnR_Fli1_rep3
GSM4514052 CnR_Igg_rep1
GSM4514053 CnR_Igg_rep2
GSM4514054 CnR_Igg_rep3
更多数据层面信息
那么咱们《生信技能树》的B站免费NGS数据处理视频课程值得你拥有,目前,已经组建了微信交流群的有下面这些: