任意基因在任意癌症里面与任意基因的表达量相关性是否受到任意基因的突变与否的状态的影响而改变!
表达量相关性通常蕴涵着两个基因背后的调控关系,而这些信息都是有大量的公共数据库资源可以供探索,比如发表于April 2021, 的文章;《Mutant p53 suppresses innate immune signaling to promote tumorigenesis》, 链接是:https://doi.org/10.1016/j.ccell.2021.01.003
主要的结论是:IFNB1 mRNA was more correlated (Pearson correlation) with wild-type p53 than with mtp53
如下图所示,在TP53野生型组的TNBC病人里面,TP53基因表达量跟IFNB1相关性还不错,但是呢,如果TNBC病人同时也有TP53突变这个组里面的TP53基因表达量跟IFNB1相关性就消失了!

图例是:. (I) Pearson Correlation coefficient of Different chemokines and cytokines expression (Red marked IFNB1) with WTp53 (n=39) and mutant p53 (n=74) as determined from TCGA dataset.
首先下载TCGA数据库的BRCA数据集
然后根据表型提取TNBC子集,然后根据突变信息把病人区分成TP53突变与否,然后下载表达量矩阵!
- TP53 wild type subgroups and TP53 missense mutation subgroups
计算相关性
在 WTp53 (n=39) and mutant p53 (n=74) 两个表达量矩阵内部,各自计算全部的基因与TP53表达量的相关性,得到如下表格:
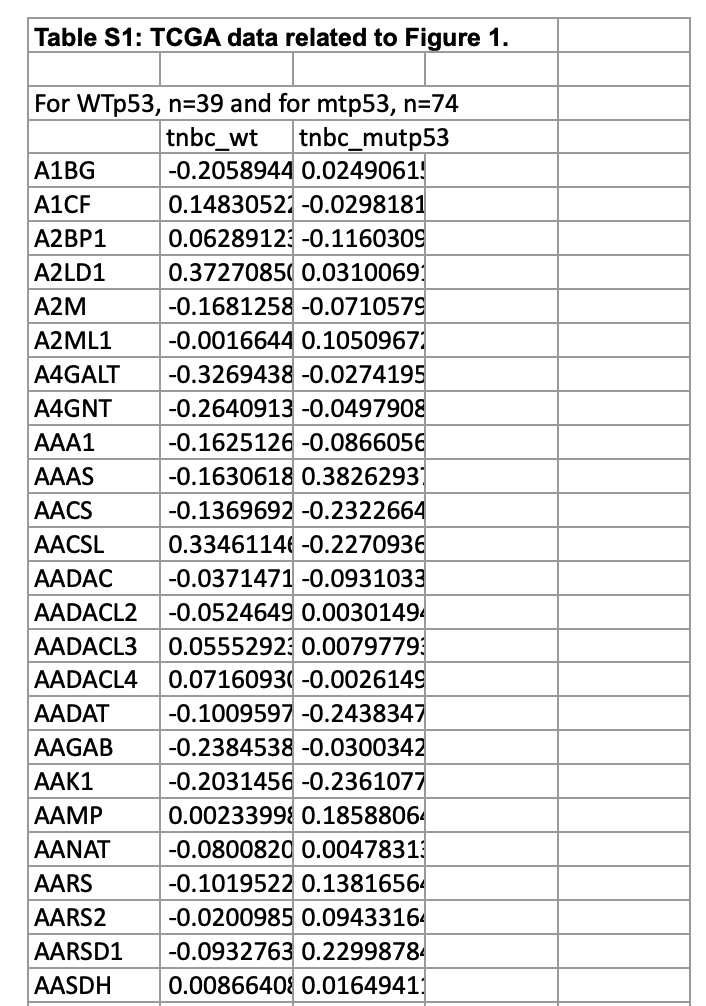
挑选感兴趣的基因
靠生物学背景啦,比如作者就挑选的是白细胞介素和趋化因子相关的基因,列表如下:

但是还是有一个问题,即使是这样的生物学背景挑选,基因数量仍然是不少,为什么作者就定位到了 IFNB1 基因呢?作为学徒作业
完成前面的散点图:图例是. (I) Pearson Correlation coefficient of Different chemokines and cytokines expression (Red marked IFNB1) with WTp53 (n=39) and mutant p53 (n=74) as determined from TCGA dataset.