提起生物信息学灌水大家首先想到的肯定是我们中国特色的临床医师畸形科研现状,不过最近我看到了一个有意思的文章:《Integrated bioinformatic analysis identifies UBE2Q1 as a potential prognostic marker for high grade serous ovarian cancer》,如下所示:
看名字就觉得应该不是中国人,
数据分析描述:
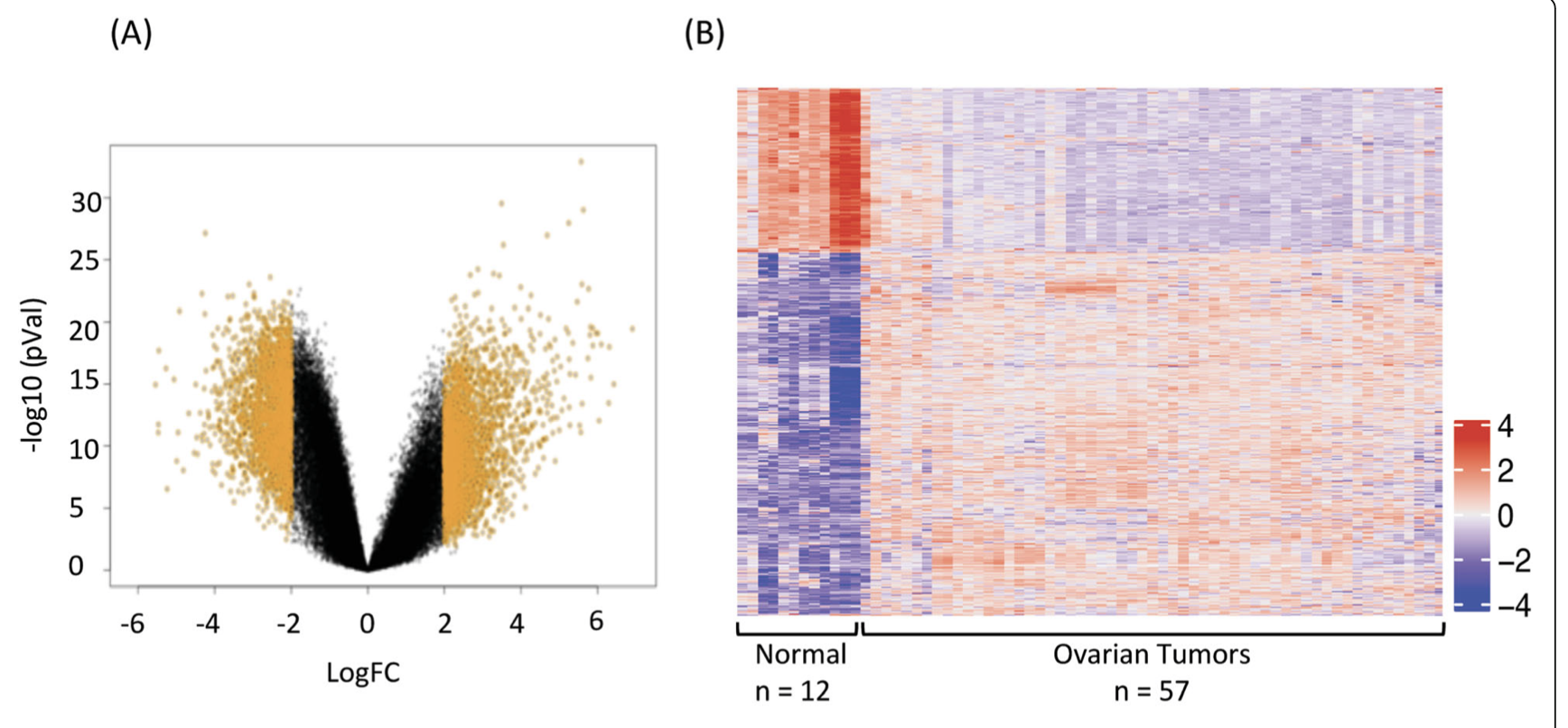
- GSE66957这个表达量芯片的 ovarian cancer patient tumor samples (n = 57) 和 normal ovarian surface epithelial tissue samples (n = 12) 的差异分析
- log2FC大于2,而且p值小于 0.01 ,得到了 810 genes were downregulated and 1797 genes were upregulated.
- Clust generated 5 clus- ters of genes, which in total included 427 genes.
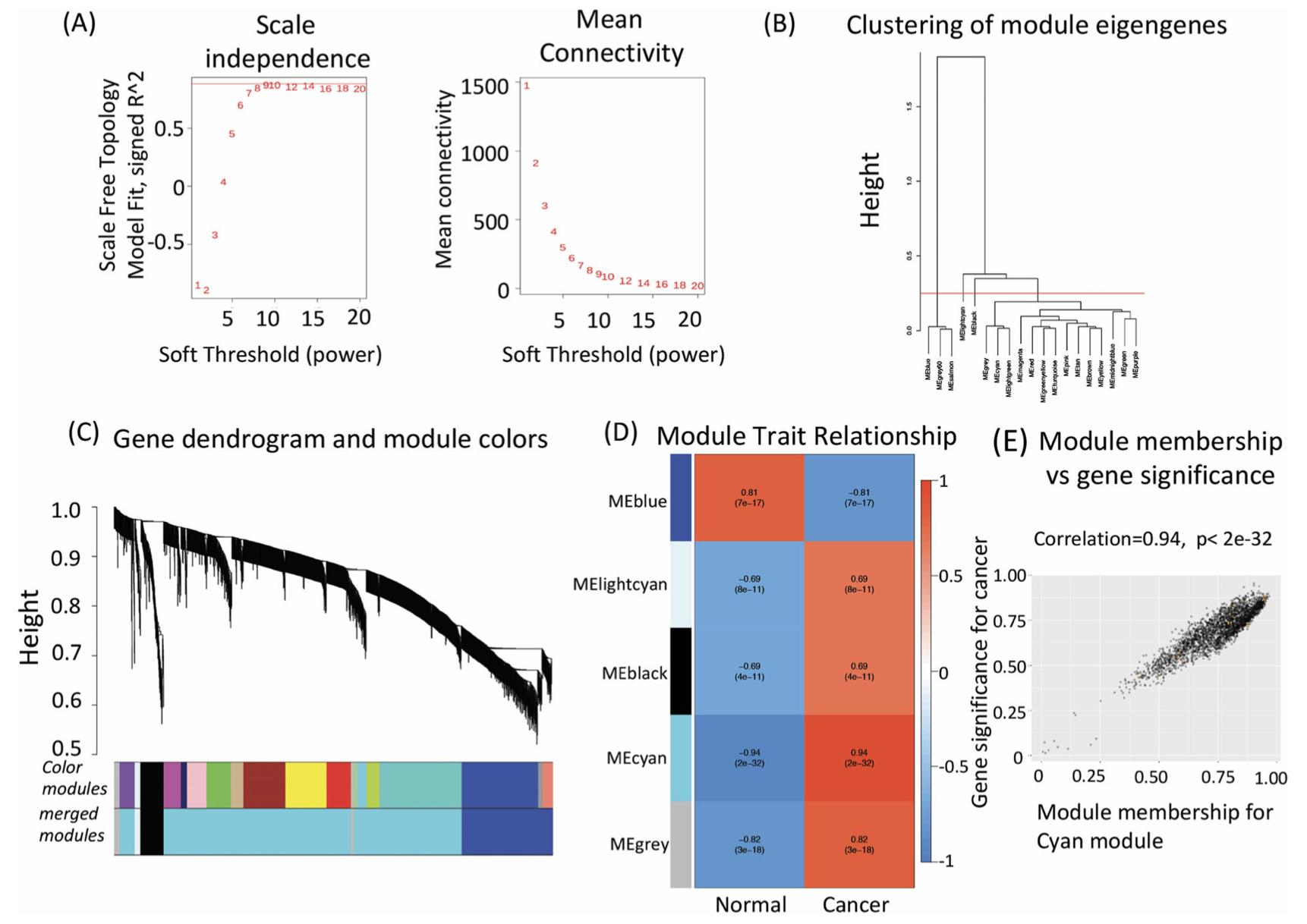
- 然后是WGCNA分析得到了 5 gene modules with co-expressing differen- tially expressed genes
- 两个分析找交集,Gene overlapping of cluster4 and cyan module identified only two genes “UBE2Q1” and “B4GALT3”
- 不同基因集进行 GO categories (BP, CC, MF, KEGG) 的富集分析注释。
差异分析就很简单了,比较容易复现,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
研究者给出来的也就是超级简陋的图表,如下所示:
WGCNA也是超级容易啦,生信技能树多次写教程分享WGCNA的实战细节,见:- 一文学会WGCNA分析
- 一文看懂WGCNA 分析(2019更新版) (点击阅读原文即可拿到测序数据)
- 通过WGCNA作者的测试数据来学习
- 重复一篇WGCNA分析的文章(代码版)
- 重复一篇WGCNA分析的文章(解读版)(逆向收费读文献2019-19)
- 关键问题答疑:WGCNA的输入矩阵到底是什么格式
- WGCNA-流程及原理细节直播互动授课(今晚八点)
其他:- 从GEO数据库下载得到表达矩阵 一文就够
- 3种缺失值情况需要区别对待
相信如果把上述教程全部看完的你,肯定是可以完成下面的图表:
不过,WGCNA的官方文档并不支持这样的首先做差异基因,然后做WGCNA的操作流程。其次,如果仅仅是两个分组,这样的wgcna分析其实就是对基因进行聚类分析而已,说明这个文章的研究着的统计学功底太弱了。
可以看到这个研究找hub 基因的思路还是蛮少见的,两个分析找交集,Gene overlapping of cluster4 and cyan module identified only two genes “UBE2Q1” and “B4GALT3”。 也许确实每个文章都有自己的道理吧。
不过呢,这里并不是说鼓励大家做生物信息学的灌水啦,只不过呢,连印度阿三都开始作妖了,你却看不懂这些图表,不懂利用已有的生物大数据,是不是有点。。。。。。文末友情推荐
与十万人一起学生信,你值得拥有下面的学习班:
- 数据挖掘(GEO,TCGA,单细胞)2021第4期
- 生信爆款入门-2021第4期