最近刷了刷植物领域单细胞文献,有一个蛮早期的拟南芥根部单细胞研究:《High-Throughput Single-Cell Transcriptome Profiling of Plant Cell Types》对拟时序分析描述的很清楚,适合做科普!
首先当然是对原始单细胞转录组表达量矩阵的降维聚类分群啦,这个在前面的例子:人人都能学会的单细胞聚类分群注释 ,我们演示了第一层次的分群。本文是植物学,但是原理是类似的,只不过不同细胞亚群的标记基因不一样。
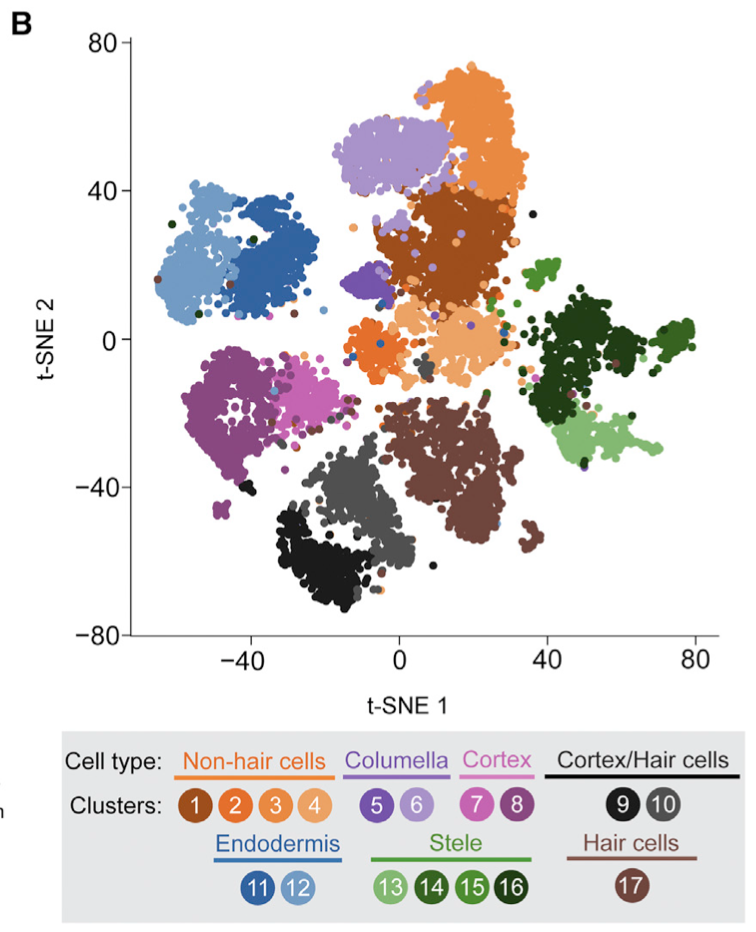
然后你需要有背景知识才能做 拟时序分析 :文章是这样描述: focusing on endodermal cells, which have a known trajectory from undifferentiated, to state I (defined by the formation the Casparian strip), to state II (defined by the formation of a secondary cell wall made of suberin) (Andersen et al., 2015)
参考文献也列的明明白白,可以看到 发育顺序如下所示,从undifferentiated 到state I 然后到state II,这个背景知识决定了你要不要做拟时序分析,已经针对哪部分细胞亚群进行拟时序分析哦!
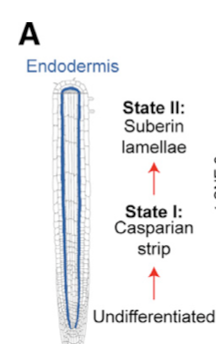
然后你需要提取指定单细胞亚群去运行拟时序分析,最经典的软件工具当然是monocle啦 ,代码超级简单,我们在《单细胞天地》公众号也多次分享过,而且三年前的我们一个单细胞公开课就是重点讲解这个代码: 「生信技能树」使用Smart-seq2单细胞转录组数据探索小鼠性腺发育,链接是:https://www.bilibili.com/video/BV1454y1q77Z
运行结果如下所示:
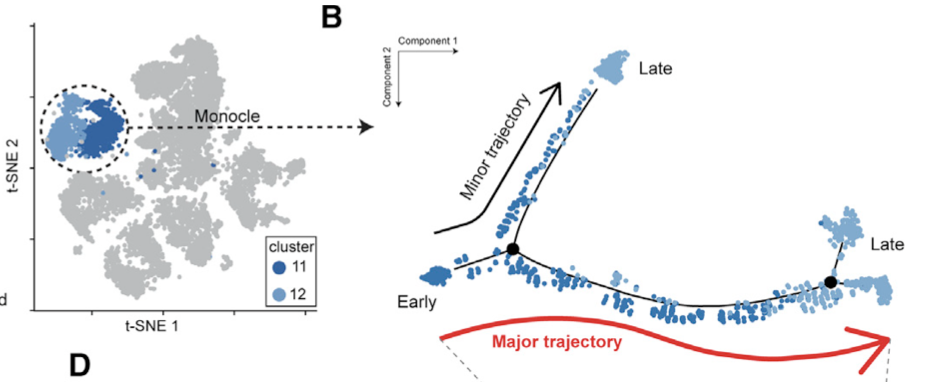
可以看到,有两个发育分支,接下来就可以展现一下重要的基因,在拟时序的两个分支上面:

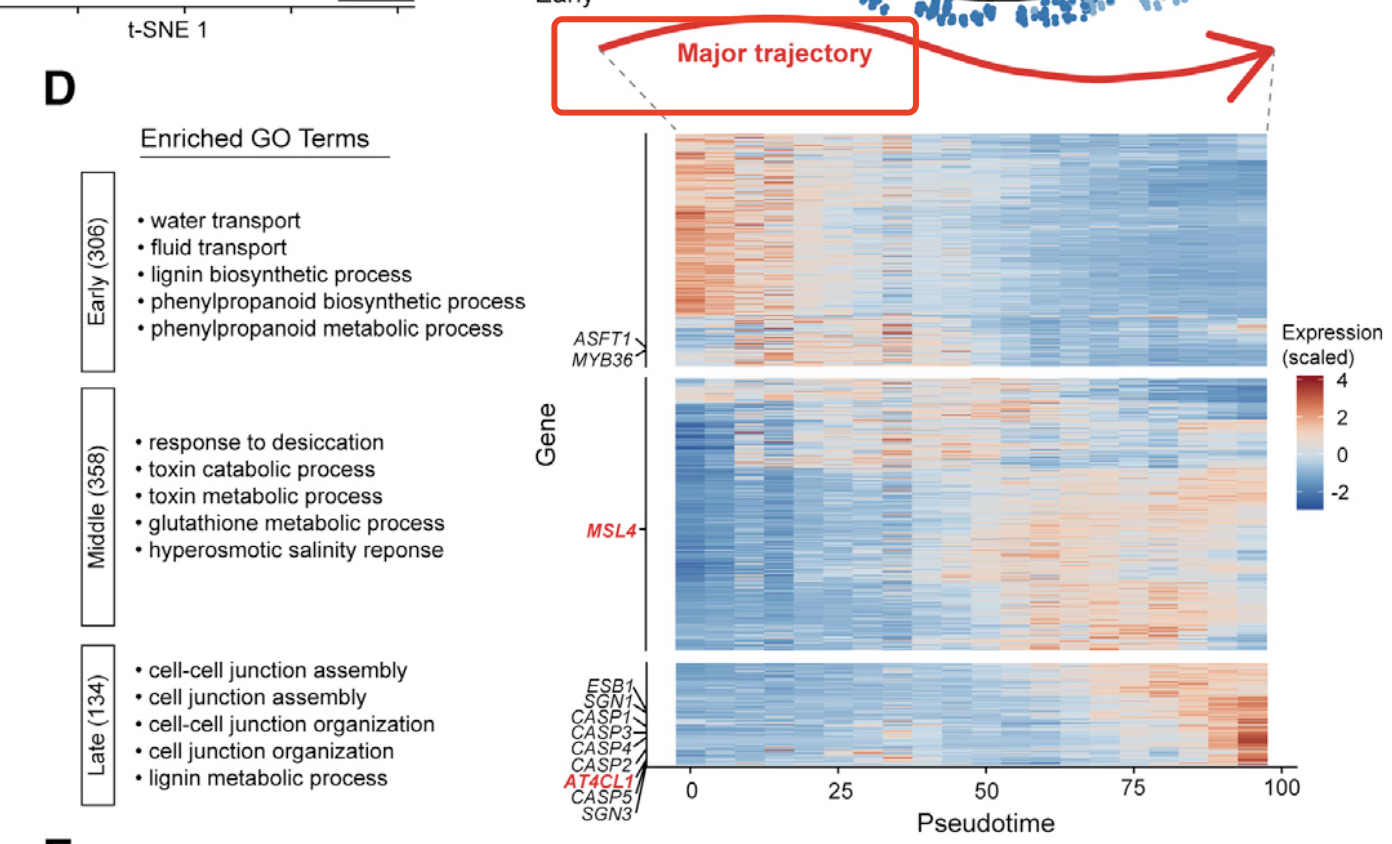
然后是拟时序的的各自特征基因的生物学功能数据库注释:
这一套图表的代码也是在「生信技能树」使用Smart-seq2单细胞转录组数据探索小鼠性腺发育,链接是:https://www.bilibili.com/video/BV1454y1q77Z ,学完了就能做出来哦!
总结一下这10个步骤:
- 降维聚类分群
- 提取指定单细胞亚群
- 运行monocle
- 判断拟时序分析推断好的发育轨迹
- 随着轨迹变化的基因获取
- 获取的基因的生物学功能数据库注释
- 还有四个步骤在这个文章里面并没有体现,这里略
拟时序分析是否可以对全部单细胞亚群呢?
在发育生物学有这样的操作,比如:
- 2019年的Nature文章”The single-cell transcriptional landscape of mammalian organogenesis”的使用Monocle鉴别出了数百种细胞类型和56条细胞发育轨迹,并定义了数千个相应的标记基因。
- 2019年的Science 文章 “A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution”,确定了502种不同的末端和前末端细胞类型,这些细胞类型分别对应1068个细胞谱系分支。
如果你对单细胞数据分析还没有基础认知,可以看基础10讲: - 01. 上游分析流程
- 02.课题多少个样品,测序数据量如何
- 03. 过滤不合格细胞和基因(数据质控很重要)
- 04. 过滤线粒体核糖体基因
- 05. 去除细胞效应和基因效应
- 06.单细胞转录组数据的降维聚类分群
- 07.单细胞转录组数据处理之细胞亚群注释
- 08.把拿到的亚群进行更细致的分群
- 09.单细胞转录组数据处理之细胞亚群比例比较