很多《生信技能树》的粉丝虽然一直在关注我们,但是他们总觉得我们这样的数据处理很遥远,感觉自己可能一辈子都不会接触ngs组学,纯粹的动物实验分子实验操作。
实际上你的关注本身就说明了问题,只不过呢你欠缺那临门一脚,人生很长,你的科研生涯可能还有35年之久,你现在学会数据处理,这个技能的掌握其实是最大化受益!现在,哪怕是全新细胞系模型的提出也需要ngs数据支持啦,比如文章:《Establishment and Characterization of a Brca1−/−, p53−/− Mouse Mammary Tumor Cell Line》
主要是基于一个已有的基因工程小鼠模型,Genetically engineered mouse models of cancer (GEMMC) ,就是 K14cre; Brca1F/F; p53F/F mice ,从里面经过各种复杂的实验技术养成细胞系。但是仍然是大篇幅描述了他们是如何利用好ngs数据来完善他们的生物学故事。主要是一个WGS测序数据,其数据分析描述如下:

数据也是公开可以获取的, 可以参考:使用ebi数据库直接下载fastq测序数据 , 需要自行配置好,然后去EBI里面搜索到的 fq.txt 路径文件:
- 项目地址是: https://www.ebi.ac.uk/ena/browser/view/PRJEB36418
脚本如下:# conda activate download # 自己搭建好 download 这个 conda 的小环境哦。 cat fq.txt |while read id do ascp -QT -l 300m -P33001 \ -i ~/miniconda3/envs/download/etc/asperaweb_id_dsa.openssh \ era-fasp@$id . done # nohup bash step1-aspera.sh 1>step1-aspera.log 2>&1 &这个脚本会根据你在EBI里面搜索到的 fq.txt 路径文件,来批量下载fastq测序数据文件。虽然仅仅是一个样品,但是我这里仍然是使用了 批量下载哦!
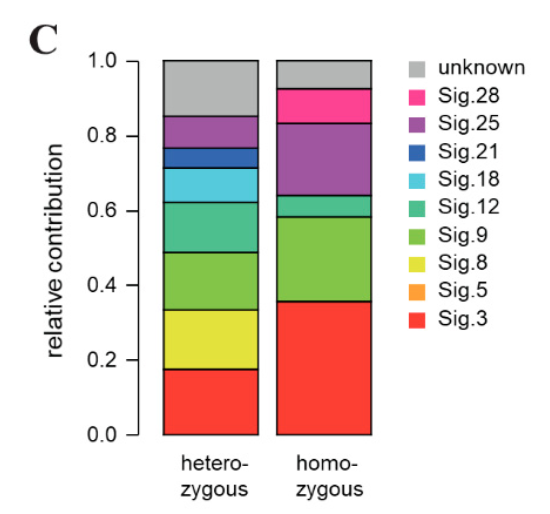
主要的数据分析结果图表,如下所示 :
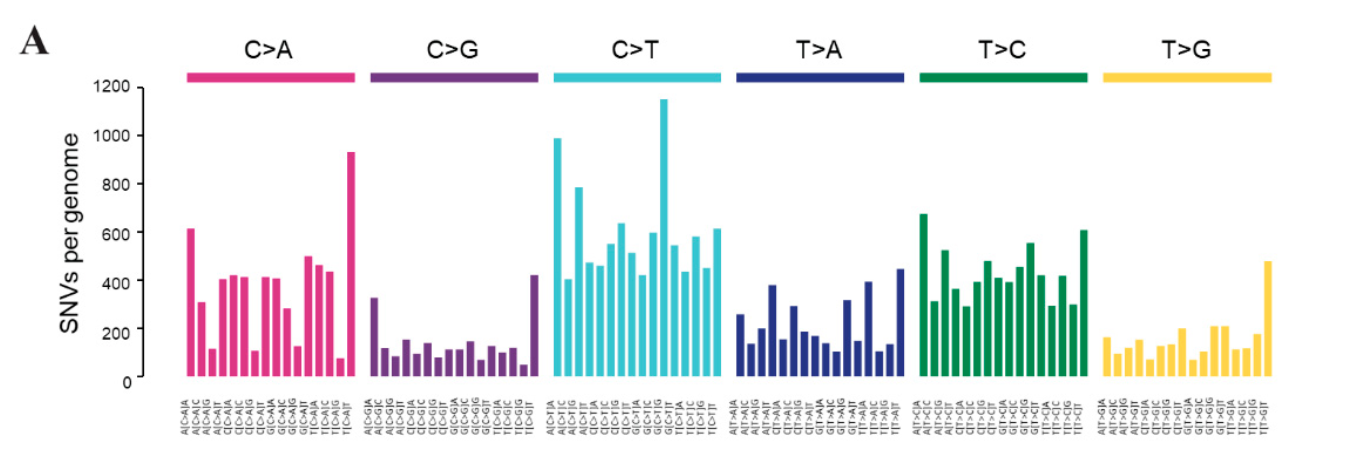
Sanger研究所科学家【1】提出来了肿瘤somatic突变的signature概念 ,把96突变频谱的非负矩阵分解后的30个特征,在cosmic数据库可以查询到的30个特征。不同的特征有不同的生物学含义【2】,比如文章【3】 就是使用了 这些signature区分生存! - 【1】https://software.broadinstitute.org/cancer/cga/msp
- 【2】https://en.wikipedia.org/wiki/Mutational_signatures
- 【3】https://www.nature.com/articles/s41586-019-1056-z
拷贝数变异作者仅仅是展现了自己关注的基因组区域;

我在教程:比较不同的肿瘤somatic突变的signature 也分享了如何比较不同方法拿到的signature,这样它们的生物学意义就可以联系起来了。主要是R包deconstructSigs可以把自己的96突变频谱对应到cosmic数据库的30个突变特征。如下所示:
学徒作业
下载这个PRJEB36418里面的测序数据,就两个文件而已:
fasp.sra.ebi.ac.uk:/vol1/fastq/ERR383/001/ERR3839731/ERR3839731_1.fastq.gz fasp.sra.ebi.ac.uk:/vol1/fastq/ERR383/001/ERR3839731/ERR3839731_2.fastq.gz然后比对到小鼠参考基因组,并且使用GATK找到变异位点后,复现这个文章里面的3个图表!