昨天我们介绍了2023年5月某院士团队发表在顶刊的文章:《Epithelial cells activate fibroblasts to promote esophageal cancer development》。详见:肿瘤相关成纤维单细胞亚群的iCAF和myCAF迷思,从大伙的留言可以看出来可能是误解了我的本意。我并没有批评把肿瘤相关成纤维(Cancer-Associated Fibroblasts,CAFs)区分成为了 iCAF和myCAF的命名方式。
众所周知,单细胞亚群的命名策略有三种:
- 首先是经典的生物学名字(淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞))
- 然后是,各个亚群的top特异性高表达量基因,比如肿瘤相关成纤维单细胞亚群的iCAF和myCAF迷思里面的3个normal fibroblasts (NFs) 亚群就是
- 最后是,各个亚群的特异性的生物学功能通路转录因子等等
如果是经典的生物学名字那么不同的单细胞转录组研究很难会出现冲突,因为差不多是形成了共识。但是这个共识在第三层次的分群就会出现冲突,比如T细胞可以按照功能进行划分,naive, memory ,effector,cytotoxic,Exhaustion: - naive (LEF1, SELL, TCF7),
- effector (IFNG),
- cytotoxicity (GZMB, PRF1),
- early and general exhaustion (PDCD1, CTLA4, ENTPD1 ) .
- antigen presentation (CD74, HLA-DRB1/5, HLA-DQA2)
但是《Tumor and immune reprogramming during immunotherapy in advanced renal cell carcinoma》,这个文献里面有一个非常诡异的降维聚类分群和生物学命名:
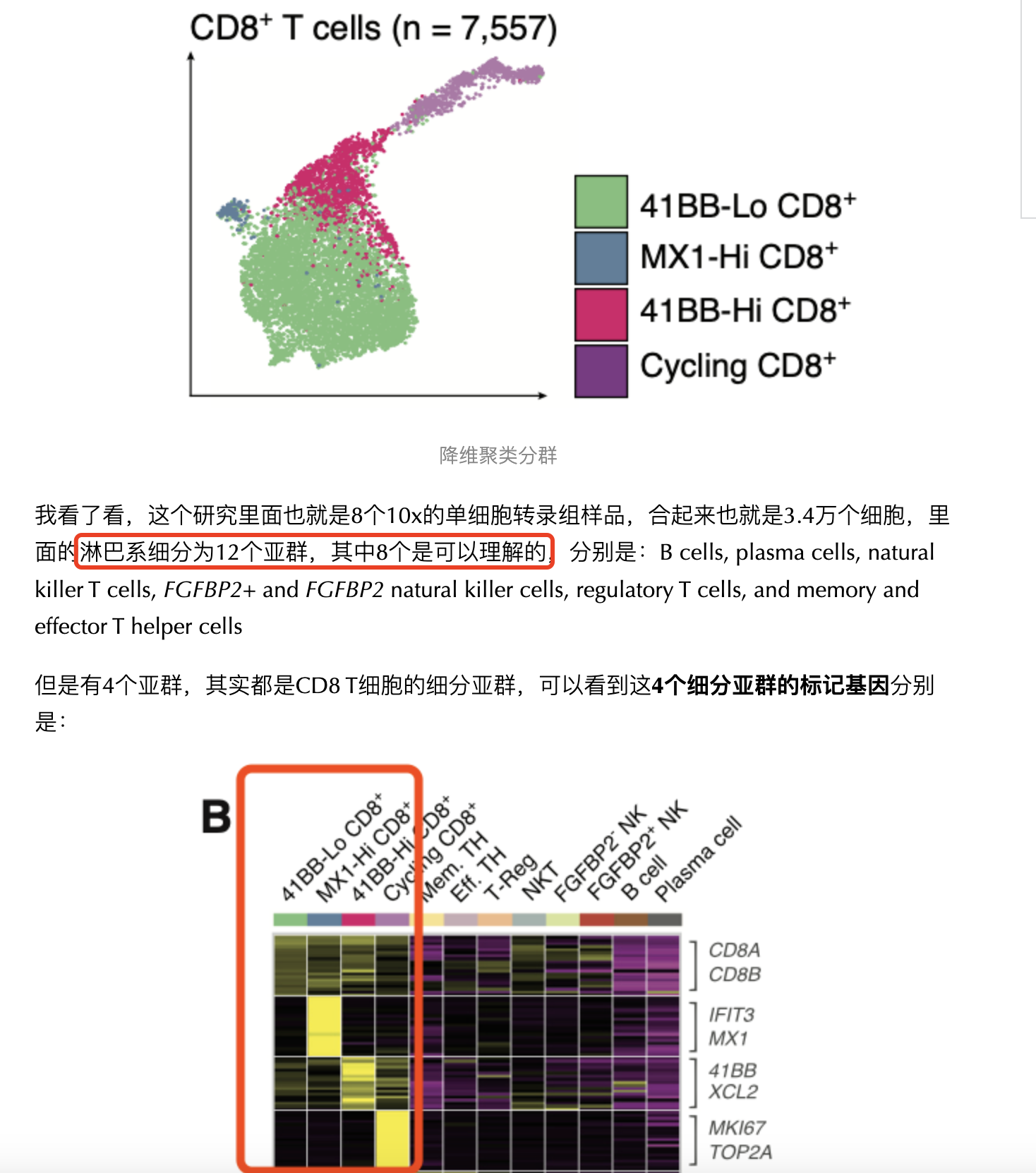
这个41BB的真正的基因名字是TNFRSF9,而且它的蛋白质名字是CD137,虽然说它在免疫学领域非常出名。但是单细胞转录组技术肯定是不仅仅是仅限于免疫学研究,那么这样的命名方式和研究成果就很难在科研界共享。使用top的特异性基因或者通路来命名天然就有主观性
虽然说我前面的推文:肿瘤相关成纤维单细胞亚群的iCAF和myCAF迷思,看起来是批评了 iCAF和myCAF的命名方式,但是实际上它反而是一种进步,如果一直是使用top的特异性基因或者通路来命名天然就有主观性,不同的研究更难直观的比较,比如2022年12月:《Interferon-dependent SLC14A1+ cancer-associated fibroblasts promote cancer stemness via WNT5A in bladder cancer》文章里面的成纤维细胞亚群如下所示:

就很难跟前面的 Myo-fibroblast (mCAF) or inflammatory fibroblast (iCAF) phenotypes 对应起来,文章的第一层次降维聚类分群如下所示:

8个病人的肿瘤和正常组织配对取样, 就是16个10x的单细胞转录组样品,本来应该是10万左右的细胞数量,被作者“严格”过滤剩下了了不到一半,然后第一层次降维聚类分群后提取里面的1000多个成纤维继续细分成为了4个亚群。
需要跟前面的 Myo-fibroblast (mCAF) or inflammatory fibroblast (iCAF) phenotypes 对应起来,所以其中C1和C2可以看做是iCAF,然后C4是mCAF,但是C3被命名为了 interferon- regulated CAF (irCAF) ,因为如下所示:

什么是真正的mCAF
我前面的推文:肿瘤相关成纤维单细胞亚群的iCAF和myCAF迷思,其实点出来了2023年5月某院士团队发表在顶刊的文章:《Epithelial cells activate fibroblasts to promote esophageal cancer development》,iCAF可能是周细胞,而myCAF就是平滑肌细胞,那么文章的价值就大打折扣了。
但是这个2022年12月:《Interferon-dependent SLC14A1+ cancer-associated fibroblasts promote cancer stemness via WNT5A in bladder cancer》文章里面的成纤维细胞亚群里面的Myo-fibroblast (mCAF)就大概率是真实的,它大量的引用了前人的文章和数据:

而且成纤维的绝大部分标记基因都是在4个亚群都高表达量的,COL1A1, S100A4, DCN, and mesenchymal marker VIM, were expressed in all 4 subclusters at high levels