看到了《生信菜鸟团》公众号组建了一个孟德尔随机化的交流群,大家热火朝天的推荐各种学习资源和公众号合集。
其中有一个资源是最新的(2023年10月)NC文章《Genome-wide association analysis of plasma lipidome identifies 495 genetic associations》里面的数据在GWAS catalog ,里面的索引号是 GCST90277238-GCST90277416,但是这个公众号的小伙伴却不知道该如何批量下载, 或者说发现规律去写代码,而且手动整理好全部的链接后下载然后把它当做是宝贝来宣传。。。。

首先需要熟悉GWAS catalog
其实没什么好说的,就是一个网页,直接查询即可。文章数据在GWAS catalog ,里面的索引号是 GCST90277238-GCST90277416 ,所以我们查询 GCST90277238试试看:
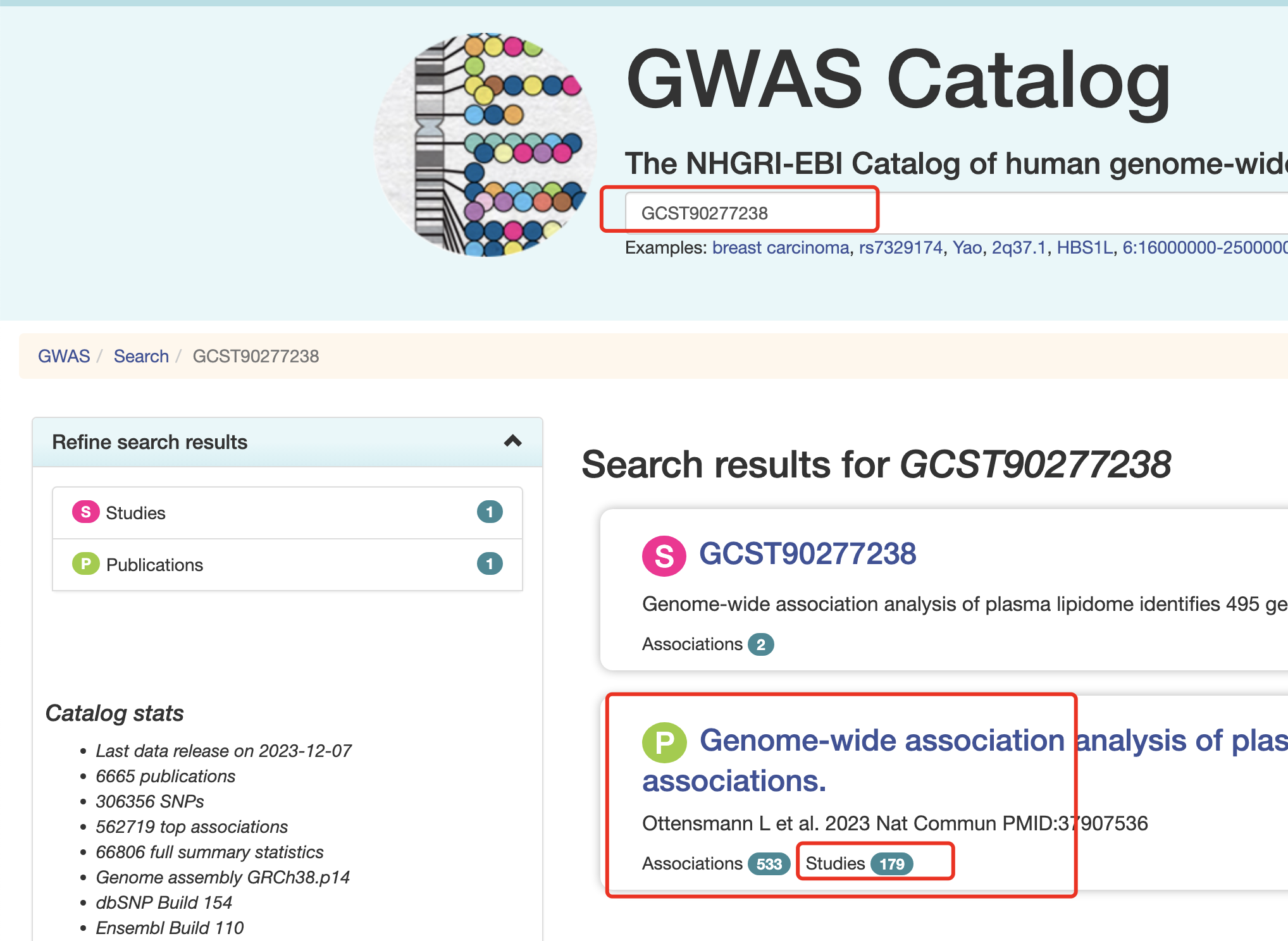
里面就链接到了 :https://www.ebi.ac.uk/gwas/publications/37907536
普通人肯定是觉得这么多链接整理起里很麻烦,如下所示:
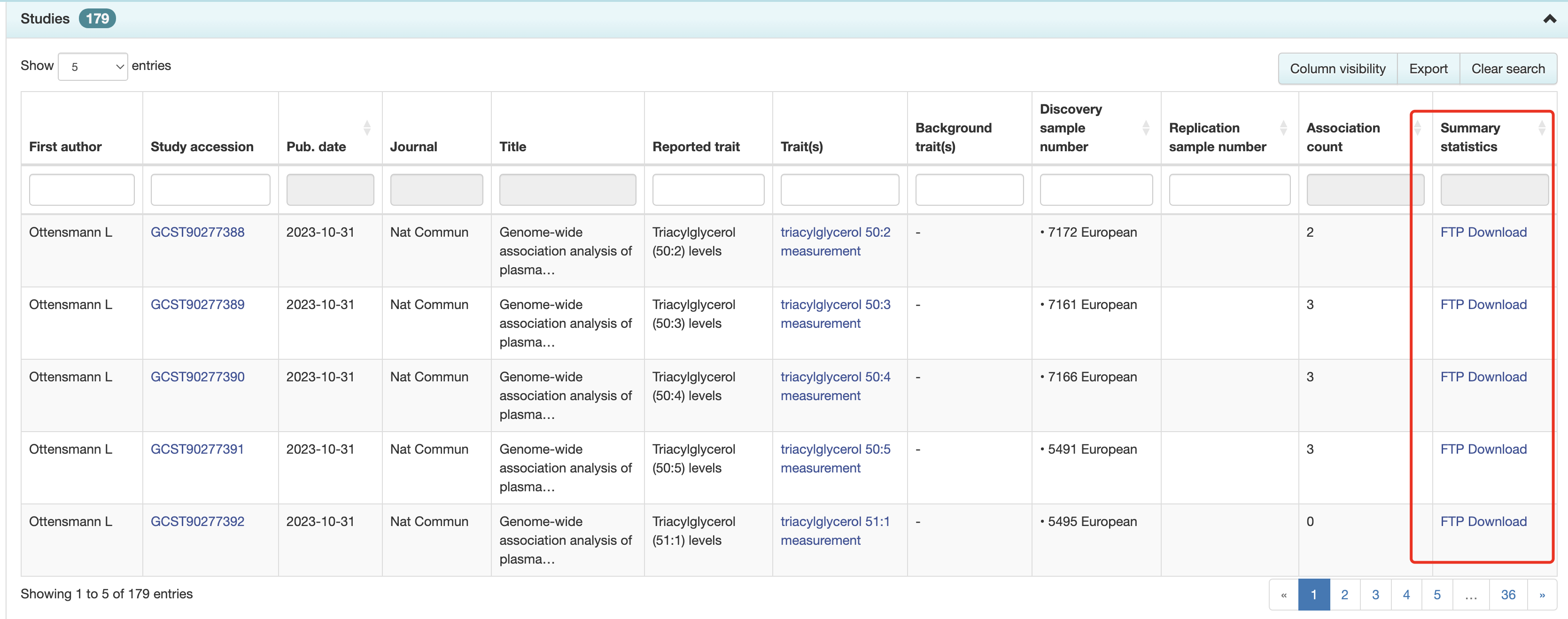
实际上,随意进入其中一个 https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/GCST90277239/
就可以看到里面的规律啦,一般来说大家需要的就是里面的 .tsv.gz 后缀的文件而已:
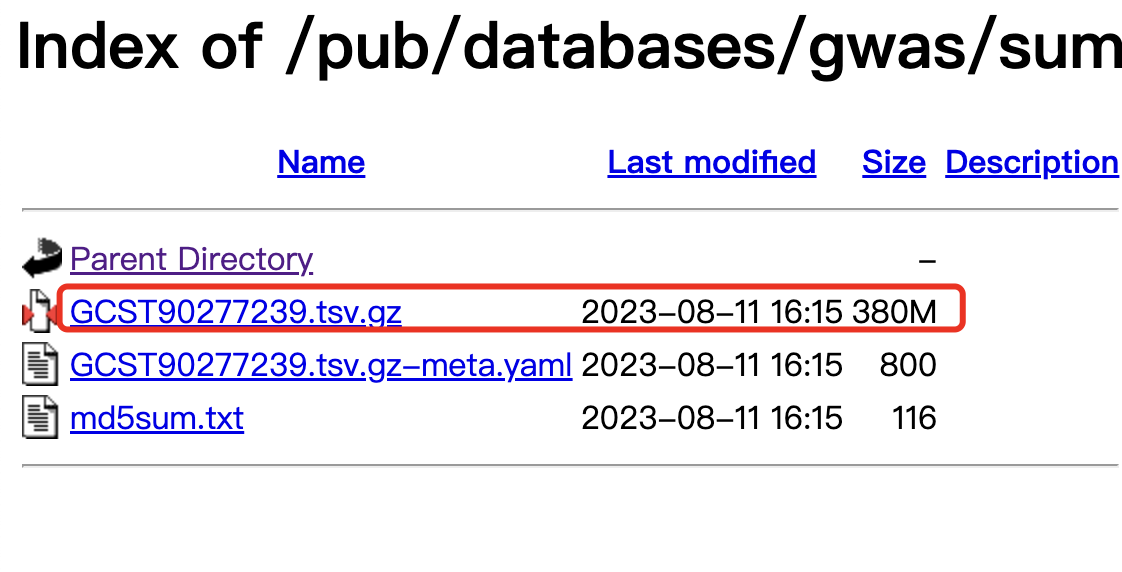 其实很容易整理一下这个链接的规律,
其实很容易整理一下这个链接的规律,
- https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/GCST90277238/GCST90277238.tsv.gz
- https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/GCST90277239/GCST90277239.tsv.gz
从 EBI(European Bioinformatics Institute)的 FTP 服务器上获取 GWAS(Genome-Wide Association Studies)的汇总统计数据文件的链接。根据链接的规律,可以得出一些模式和含义:
- 共同部分:
https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/是所有链接的共同部分,表示这是 GWAS 汇总统计数据的基础路径。
- 项目范围:
GCST90277001-GCST90278000表示这些数据属于 GWAS Catalog(GCST) 中的一个范围,包括从GCST90277001到GCST90278000的多个项目。
- 具体项目目录:
GCST90277238和GCST90277239是具体项目的目录,每个目录对应一个特定的 GWAS 项目。
- 文件名规律:
GCST90277238.tsv.gz和GCST90277239.tsv.gz是文件名,表示这是 GWAS 项目的汇总统计数据文件。.tsv.gz扩展名表明这是一个经过压缩的以制表符分隔的文本文件。
只需要一个简单的循环即可,前面的文章数据在GWAS catalog ,里面的索引号是 GCST90277238-GCST90277416 ,一个简单的 Shell 脚本示例,使用了 wget 命令和一个 for 循环:
#!/bin/bash
# 设置基础路径
base_url="https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/"
# 设置下载目录
download_dir="./" # /path/to/your/download/directory
# 循环下载指定范围的文件
for ((i=90277238; i<=90277416; i++)); do
# 构建完整的下载链接
file_url="${base_url}GCST${i}/GCST${i}.tsv.gz"
# 使用wget下载文件到指定目录
wget -P "$download_dir" "$file_url"
done
请替换 "/path/to/your/download/directory" 为你希望保存文件的目录路径。这个脚本会下载范围内的每个文件,并保存到指定的目录中。确保你的系统上已经安装了 wget 命令。你可以将上述代码保存到一个文件(比如 download_files.sh),然后通过 chmod +x download_files.sh 赋予执行权限,并运行 ./download_files.sh 执行脚本。
也可以很轻松的替换成为其它编程语言,比如在R语言中,你可以使用download.file函数来完成这个任务。以下是一个R脚本的例子:
# 设置基础路径
base_url <- "https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/"
# 设置下载目录
download_dir <- "/path/to/your/download/directory"
# 循环下载指定范围的文件
for (i in 90277238:90277416) {
# 构建完整的下载链接
file_url <- paste0(base_url, "GCST", i, "/GCST", i, ".tsv.gz")
# 使用download.file下载文件到指定目录
download.file(file_url, destfile = file.path(download_dir, paste0("GCST", i, ".tsv.gz")))
}
请替换 "/path/to/your/download/directory" 为你希望保存文件的目录路径。你可以将上述代码保存到一个R脚本文件(比如 download_files.R),然后通过source("download_files.R")执行脚本。
在Python中,你可以使用requests库来下载文件。以下是一个Python脚本的例子:
import os
import requests
# 设置基础路径
base_url = "https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/GCST90277001-GCST90278000/"
# 设置下载目录
download_dir = "/path/to/your/download/directory"
# 循环下载指定范围的文件
for i in range(90277238, 90277417):
# 构建完整的下载链接
file_url = f"{base_url}GCST{i}/GCST{i}.tsv.gz"
# 使用requests下载文件到指定目录
response = requests.get(file_url, stream=True)
file_path = os.path.join(download_dir, f"GCST{i}.tsv.gz")
with open(file_path, 'wb') as file:
for chunk in response.iter_content(chunk_size=8192):
file.write(chunk)
请替换 "/path/to/your/download/directory" 为你希望保存文件的目录路径。你需要确保你的Python环境中已经安装了requests库。你可以将上述代码保存到一个Python脚本文件(比如 download_files.py),然后通过python download_files.py执行脚本。
真的是基础不牢地动山摇啊
再怎么强调生物信息学数据分析学习过程的计算机基础知识的打磨都不为过,我把它粗略的分成基于R语言的统计可视化,以及基于Linux的NGS数据处理:
把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
Linux的6个阶段也跨越过去 ,一般来说,每个阶段都需要至少一天以上的学习:
- 第1阶段:把linux系统玩得跟Windows或者MacOS那样的桌面操作系统一样顺畅,主要目的就是去可视化,熟悉黑白命令行界面,可以仅仅以键盘交互模式完成常规文件夹及文件管理工作。
- 第2阶段:做到文本文件的表格化处理,类似于以键盘交互模式完成Excel表格的排序、计数、筛选、去冗余、查找、切割、替换、合并、补齐,熟练掌握awk、sed、grep这文本处理的三驾马车。
- 第3阶段:元字符,通配符及shell中的各种扩展,从此linux操作不再神秘!
- 第4阶段:高级目录管理:软硬链接,绝对路径和相对路径,环境变量。
- 第5阶段:任务提交及批处理,脚本编写解放你的双手。
- 第6阶段:软件安装及conda管理,让linux系统实用性放飞自我。