前些天有一个人给了我这样的留言,让我我现在很迷茫:
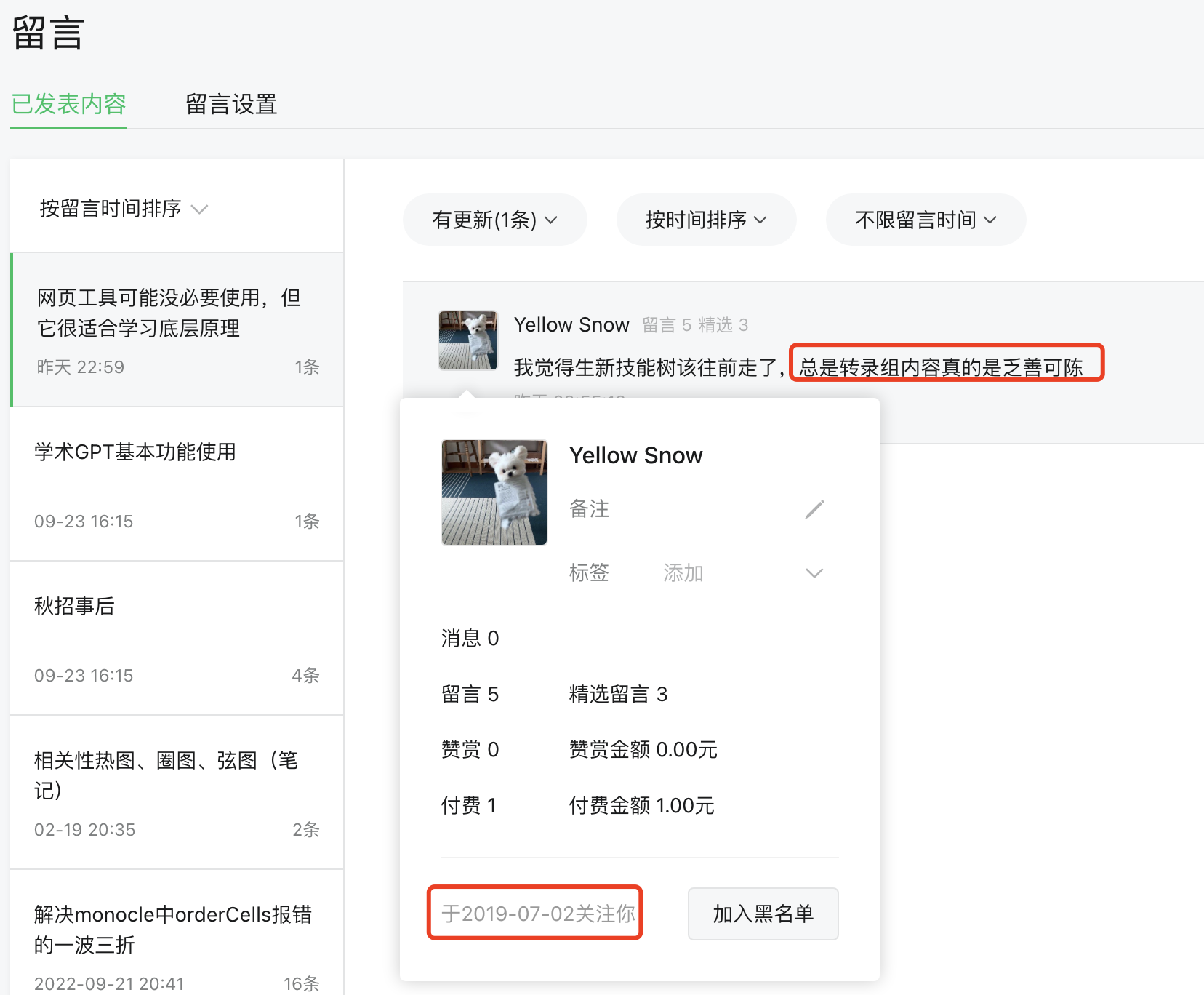
这个人已经是四五年的老粉了,确实,如果他一路看过来,很容易发现我们其实一直是三板斧(编程基础+ngs多组学+单细胞)。很单调,是有点乏善可陈,但是我个人确实是感觉写什么都很难突破过去的自己,这个公众号创作是越来越搞不下去了。。。。
虽然说我们偶尔也有一些其它专题,比如流式细胞仪数据处理:
- cytof数据拆分
- cytof数据处理难点之修改FCS文件
- cytof数据处理难点之合并两个不同panel的数据集
- cytof数据处理难点之细胞数量平衡
- cytof数据处理难点之细胞亚群继续分群
- cytofWorkflow之亚群比例差异分析(六)
- cytofWorkflow之人工注释生物学亚群(五)
- cytofWorkflow之聚类分群(四)
- cytofWorkflow之基本质量控制(三)
- cytofWorkflow之构建SingleCellExperiment对象(二)
- cytofWorkflow之读入FCS文件(一)
以及表观调控领域最经典的甲基化芯片数据处理:
- 甲基化探针相对于基因来说太多了怎么办
- EpiDISH-根据甲基化信号值推断样品的细胞成分
- 850K甲基化芯片的TSS附近信号值曲线绘制
- 学徒作业:给你8个甲基化探针, 你在tcga数据库进行任意探索
- 甲基化信号值矩阵差异分析哪家强
- TCGA数据库的各个癌症甲基化芯片数据重新分析
- 甲基化信号值矩阵差异分析主要图表
- 450K芯片上面的甲基化探针到底需要进行哪些过滤
也有些临床数据挖掘实践,比如泛癌数据分析:
- 指定通路绘制gsea图热图和火山图
- 所有的肿瘤都有恶性增殖的特性吗
- 肿瘤高表达基因一定是有差的预后吗
- 癌基因都是肿瘤的风险因子吗
- 癌基因一定在肿瘤部位高表达吗
- 如果你一定要TCGA数据库的转录组测序的TPM表达量矩阵,不妨自己进行转换啊!
- 泛癌水平的批量生存分析
- 生存分析有必要把连续值依据中位值进行高低分组变成分类变量吗
- estimate或者CIBERSORT结果真的是很好的临床预后指标吗
- 引入肿瘤分子分型能提供临床分期的预后能力吗
也是有一些小众数据挖掘,比如免疫组库:
- 使用IMonitor进行免疫组库分析
- 使用MiXCR进行免疫组库分析
- 使用igblast进行免疫组库分析
- 认识免疫组库测序数据
- 从IMGT数据库下载免疫组库相关fasta序列
- 通过IMGT数据库认知免疫组库
还有肿瘤相关的DNA测序数据分析,比如肿瘤外显子的:
- 使用R包deconstructSigs根据已知的signature进行比例推断
- 使用R包SomaticSignatures进行denovo的signature推断
- 下载TCGA所有癌症的maf文件做signature分析
- TCGA各大癌症的somatic突变全景图鸟瞰(动态交互)
- 根据肿瘤突变信息计算一系列指标
- 你真的理解体细胞突变吗?
- 突变位点生存分析
- 比较不同的肿瘤somatic突变的signature
- 一步一步用Snakemake搭建gatk4生成正常样本的germline突变数据库的流程
- 学徒作业-两个基因突变联合看生存效应
- 多位点取样外显子测序看食管癌的肿瘤内部突变异质性(逆向收费读文献2020-15)
- SNV突变(96种)频谱的制作
但是这样的笔记无论是从质的角度还是量的角度,都没办法撑得起来一个生物信息学领域论坛(生信技能树),仅仅是我个人的学习经历的笔记合辑。
而我一个人的创作就必然会陷入“舒适区”怪圈,即使是强迫自己去不停地学习各种技术做整理,反而是耽误了自己的成长,总不能说一直就学习学习基础吧。这条路我五年前已经是走过一次了:
学徒第1月,基础知识介绍掌握 https://www.mubu.com/doc/38tEycfrQg
学徒第2月,RNA-seq数据分析实战训练 https://www.mubu.com/doc/38y7pmgzLg
学徒第3月,WES数据分析实战训练 https://www.mubu.com/doc/1iDucLlG5g
学徒第4月-01-ChIP-seq数据分析实战训练 https://www.mubu.com/doc/11taEb9ZYg
学徒第4月-02-ATAC-seq数据处理实战 https://www.mubu.com/doc/2DG1mC2kdg
学徒第4月-03-表观调控(RNA-seq和ChIP-seq的联合分析) https://www.mubu.com/doc/3Bd4aieYug
学徒第5月-01-甲基化芯片(450K或者850K)数据处理 https://www.mubu.com/doc/1cwlFgcXMg
学徒第5月-02-LncRNA实战 https://www.mubu.com/doc/ISk-Ev1tg
学徒第5月-03-miRNA-seq实战演练 https://www.mubu.com/doc/7A3T8hpUlLv
现在难道是还得逼着自己再来一次吗?
未来的生物信息学知识点整理的路该如何走?如何才能号召更多人,或者说如何更好的组织有志之士参与协作整理呢?
01-生物信息学绘图大全 https://www.mubu.com/doc/3L0wkgGUVg
02-跟着bioconductor包学生物信息学 https://www.mubu.com/doc/1WKFG-171g
03-生物信息学必知必会 https://www.mubu.com/doc/3i-F76DfYg
04-转录组笔记推文任务列表(半年期) https://www.mubu.com/doc/14uneHKvPg
04-转录组下游分析知识点面面俱到 https://www.mubu.com/doc/3pRgWLU5Lg
算起来,我都做了快十年了,如果说十年间一直在把这三板斧(编程基础+ngs多组学+单细胞)反复咀嚼,确实是说不过去:
01-TCGA数据挖掘文献整理 https://www.mubu.com/doc/36AU00FIxWf
02-GTEx和ENCODE等大型国际NGS计划文献整理 https://www.mubu.com/doc/5D5dC7Fep7f
03-转录组2023马拉松授课大纲 https://www.mubu.com/doc/47d_2fEmcqv
04-数据挖掘本科毕业设计辅导 https://www.mubu.com/doc/1L99bY0csg
05-单细胞初识(生信技能树+单细胞天地) https://www.mubu.com/doc/3347KRIo18v
单细胞数据一站式分析平台的开发及推广 https://www.mubu.com/doc/2RYc4vgN2R_
临床数据挖掘36计 https://www.mubu.com/doc/7vQl-BKxSbv
生物信息学百款软件 https://www.mubu.com/doc/nMIuO1vG5_
亲爱的读者朋友们,大家有什么好的建议吗?