长期以来,我们就分享了:《 细胞亚群细分的时候仍然是要选择harmony等算法去除样品差异 》,也就是说目前的单细胞转录组数据分析环节因为通常都是多个样品正好,所以harmony整合是必不可少的, 尤其是第一层次降维聚类分群和后面的细分亚群。而且通常我们拿到了肿瘤相关的单细胞转录组的表达量矩阵后的第一层次降维聚类分群通常是:
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibro or CD31+,PECAM1,endo)
参考我前面介绍过 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的 fibro 和endo进行细分,并且编造生物学故事的。
但是,其中上皮细胞细分亚群就有待商榷,因为里面有恶性肿瘤上皮细胞以及正常的上皮细胞,所以我们通常是需要把这些上皮细胞harmony整合,然后区分正常细胞和恶性细胞后,这个时候跑的是inferCNV流程,详见:肿瘤单细胞转录组拷贝数分析结果解读和应用,但是里面的纯粹的恶性肿瘤细胞被挑选好后,因为是来源于多个不同的病人, 就不一定要继续harmony整合的,详见:使用harmony等算法去除单细胞样品差异得谨慎 。
其实是取决于的研究目标,如果你想研究的是肿瘤内部异质性(ITH),一般来说是针对每个个体进行NMF等找关键基因列表,然后汇总,所以不需要harmony整合的。但是并不是说研究ITH是所有人的必须,比如2024-CELL-黑色素瘤-免疫治疗-单细胞文章,标题是:《A TCF4-dependent gene regulatory network confers resistance to immunotherapy in melanoma》,就是:We grouped all malignant cells in copy-number variation (CNV) genomic clusters using inferred CNV profiles.
然后针对这些恶性肿瘤细胞看到了每个病人的各自的恶性肿瘤细胞的特异性,使用了harmony整合,如下所示:

反正只需要跑降维聚类分群流程,肯定是有多个亚群,而且每个亚群都有top的基因和通路或者转录因子,就可以进行生物学命名:
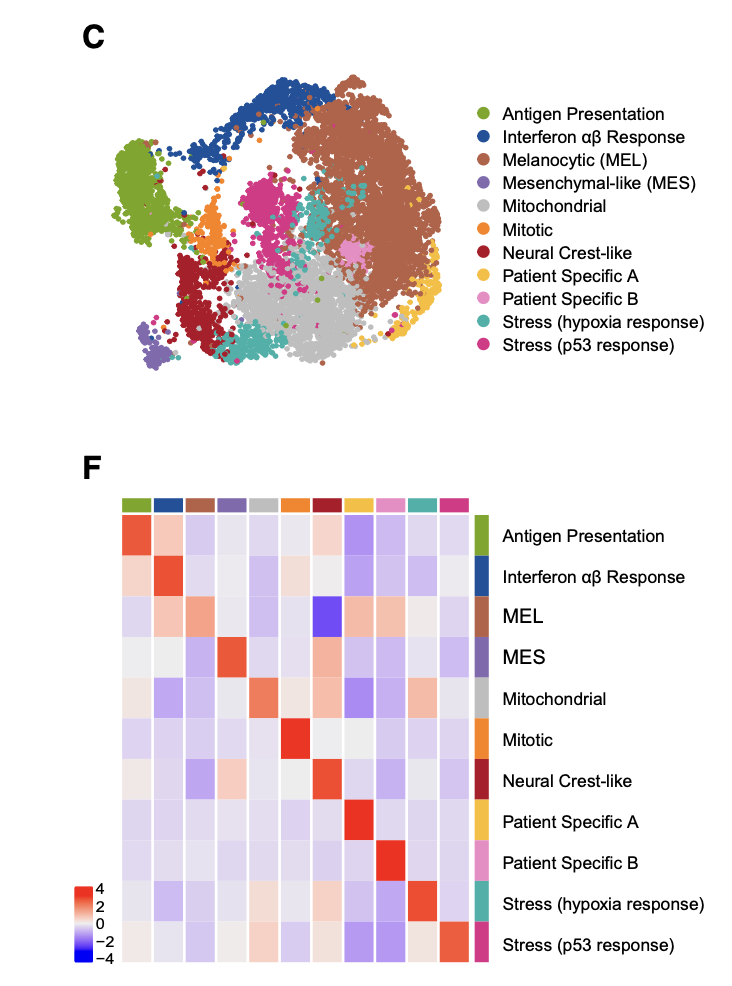
这样的单细胞转录组数据分析的标准降维聚类分群,并且进行生物学注释后的流程可以参考前面的例子:人人都能学会的单细胞聚类分群注释 ,我们演示了第一层次的分群。如果你对单细胞数据分析还没有基础认知,可以看基础10讲: