昨天介绍了:泛癌层面的表观调控单细胞多组学资源,是从11种癌症类型的201名患者中获取了225个样本做了snATAC-seq与snRNA-seq两个单细胞技术,数据量非常可观,对做癌症研究的小伙伴们来说意义重大。
今天再介绍一下 NCI-supported CPTAC 计划的新鲜出炉的泛癌层面的蛋白质组学资源文章;《Pan-cancer proteogenomics characterization of tumor immunity》,如下所示,可以看到是10种不同癌症的1,000多个肿瘤样品哦:

详细的10种癌症,以及样品数量如下所示:
- breast cancer (BC, n = 113),
- clear cell renal cell carcinoma (CCRCC, n = 103),
- colon cancer (CO, n = 96),
- glioblastoma (GBM, n = 99),
- head and neck squamous carcinoma (HNSCC, n = 110),
- lung squamous carcinoma (LSCC, n = 108),
- lung adenocarcinoma (LUAD, n = 110),
- ovarian cancer (OV, n=82),
- pancreatic ductal adenocarcinoma (PDAC, n = 140),
- uterine cancer (UCEC, n=95).
虽然说CPTAC(临床蛋白质组肿瘤分析联盟,Clinical Proteomic Tumor Analysis Consortium)主要是quantita- tive proteomics, and phosphoproteomics.这两个蛋白质组技术,实际上也有whole-genome sequencing (WGS), RNA-seq这两个传统组学技术。
主要是根据表达量矩阵进行免疫相关基因列表对全部的癌症的全部的样品进行打分,推断样品里面的免疫细胞亚群组分的比例情况;
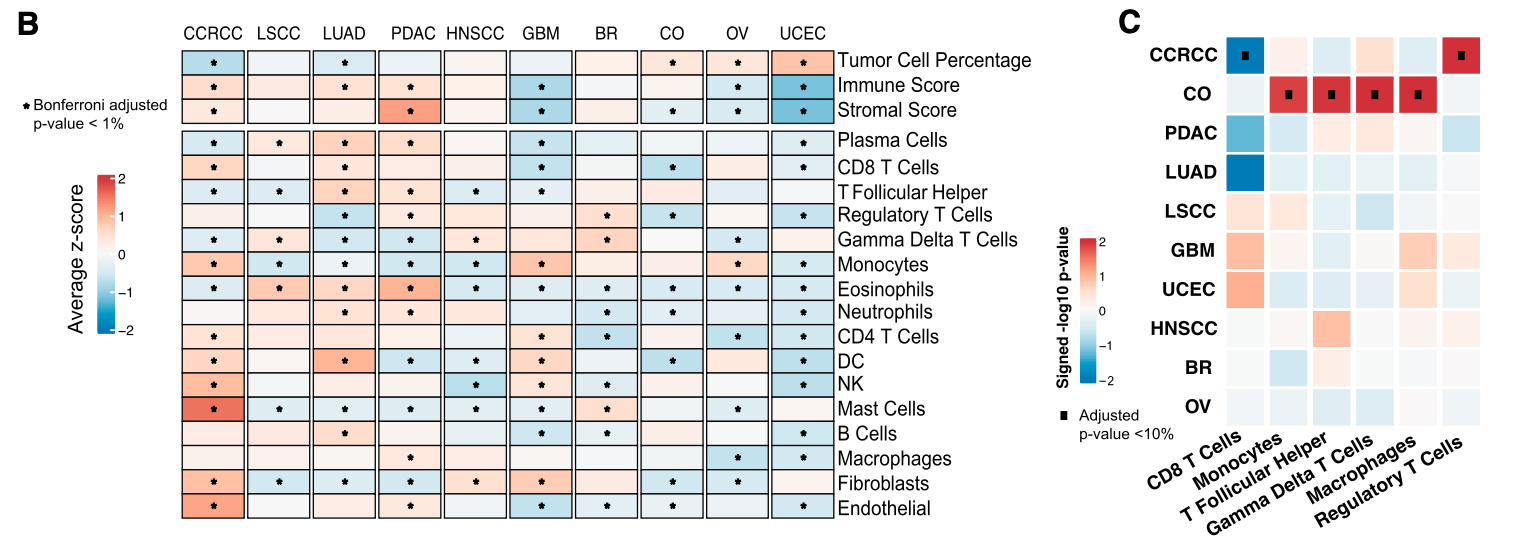
感兴趣的可以自行研读这个蛋白质组学资源文章;《Pan-cancer proteogenomics characterization of tumor immunity》,
大家感兴趣的可能是数据的公开程度,比昨天介绍了:泛癌层面的表观调控单细胞多组学资源的要好一点,起码表达量矩阵这样的我们很容易下载处理的信息是公开的,我也点进去看了看,非常齐全!

另外,让我们生信工程师比较感兴趣的可能是很多数据分析方法,软件工具的使用: