大清早的(2024-02-15 07:20)看到了《北京未来基因诊断高精尖创新中心》公众号宣传了他们单位的最新nature文章,详见:Nature | 白凡课题组与王红阳院士团队联合揭示中国人群肝癌全基因组变异及演化特征。本来呢,这样的资源型文章一般来说我们就看一下即可,因为人类遗传资源保护越来越严格了,基本上不可能看到病人的测序数据的公开,所以很难二次利用!
但是,朋友圈转发这个文章的越来越多,以至于我不得不还是在春节期间打开电脑瞟了一下这篇文献,文章标题短小精悍:《Deep whole-genome analysis of 494 hepatocellular carcinomas》,甚至于在标题里面并不需要突出中国人群这样的 关键词了,文章讲清楚了两件大事:
- Here we initiated the Chinese Liver Cancer Atlas (CLCA) project
- and performed deep whole-genome sequencing (average depth, 120×) of 494 HCC tumours.
但是文章不仅仅是接近500个肝癌患者的全基因组,其实还有部分转录组测序:We also performed RNA sequencing (RNA-seq) analysis of 239 tumours from this cohort,同样的转录组也是很难拿到测序数据,但是作者给出来了 Supplementary Table 2,TPM matrix of RNA-seq cases.
既然说是资源型文章,所以文章的侧重点就是这些ngs组学数据分析啦,让我们肃然起敬的是其数据整理格式,以及文章配套网页工具和全部的代码。
虽然说, 因为人类遗传资源保护越来越严格了,我们无法获取测序数据,但是实际上,绝大部分人要来了这个测序数据也没什么意义,每个病人仅仅是肿瘤样品就200多个G的左右两个测序数据文件,如下所示:
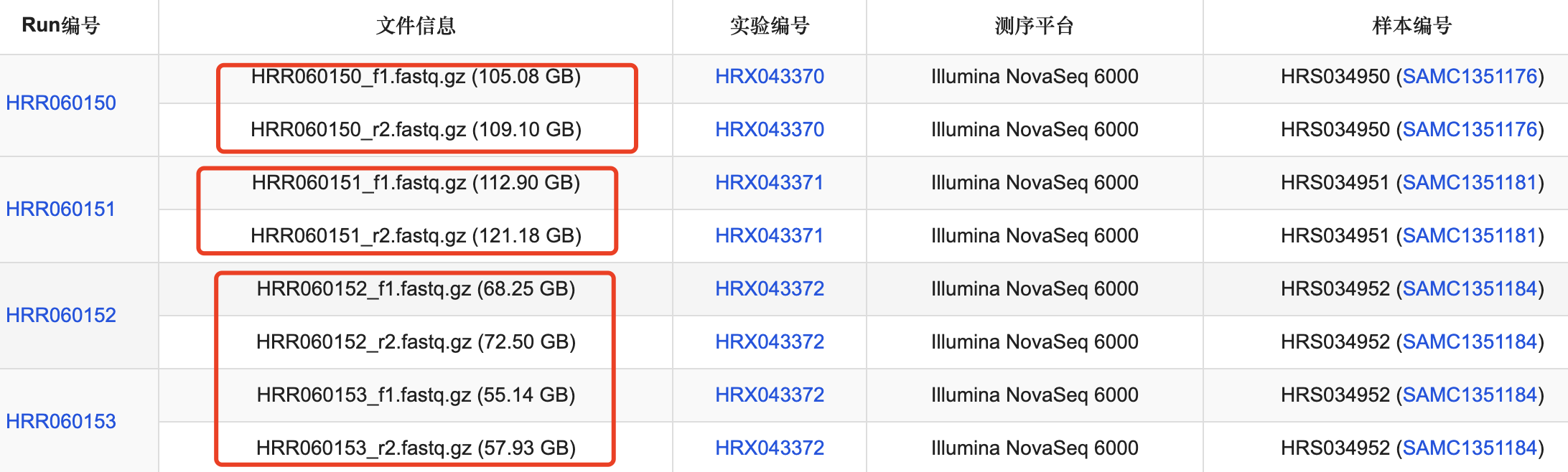
也就是说这500多个病人就是100T的原始测序数据,每个病人还得要配对的正常组织的测序,因为肿瘤样品需要高深度测序,为了节省测序费用,配对的正常组织可以十分之一的测序量即可。总体上来说,110T的人类的WGS的原始测序数据,绝大部分课题组都可以放弃啦!哪怕是我们做肿瘤测序数据分析教学,通常也是《肿瘤外显子》这个技术,在《生信菜鸟团》有一个专栏,该专栏的目录(节选)如下: - (一)读文献并且下载测序数据
- (二)质控与去接头
- (三)比对
- (四)比对结果的质控
- (番外篇)bam文件载入igv可视化
- (五)GATK的最佳实践
- (六)vcf文件的注释及ANNOVAR的使用
- (七)maftools可视化
- (八)不同注释软件的比较(上):安装及使用
- (八)不同注释软件的比较(中):注释后转成maf文件
- (八)不同注释软件的比较(下):可视化比较maf文件
如果大家啊确实是有这个数据量,我相信,绝大部分科研服务公司肯定是会“跪舔”大家的,所以这个时候讨论上游数据分析意义不大了!超齐全的生物信息学配套材料
虽然说,我们绝大部分人都可以直接放弃上面的测序原始数据,毕竟是110T的文件,但是,这个nature文章的优点就是超齐全的生物信息学配套材料,总体上来说是:
- 在GitHub (https://github.com/ChongJenniferZhang/CLCA_WGS). 可以看到
- 在Zenodo (https://doi.org/10.5281/zenodo.7260221). 可以看到全部的 Singularity containers
- 在网页工具 (http://lifeome.net/database/liver) 可以看到
其中github代码大家可以自行下载阅读,里面的代码我看了看,还不错,都是常规的shell脚本,所以很容易学习 :
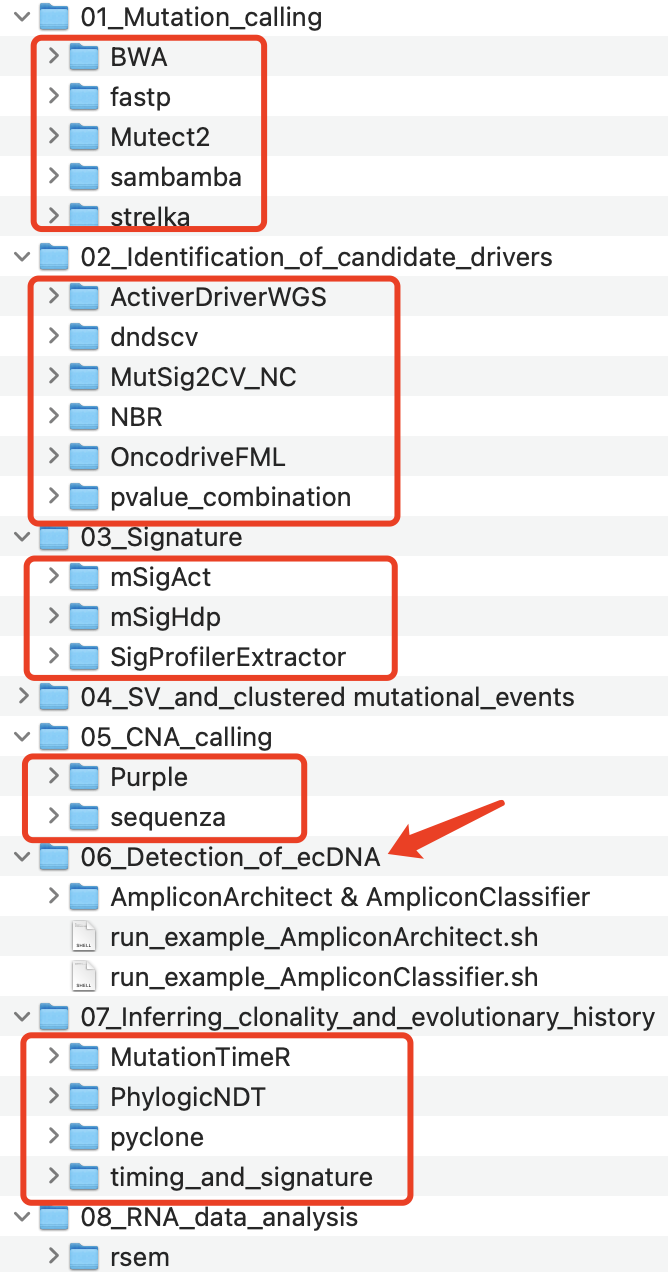
但是Zenodo的Singularity容器技术也超出了普通入门级别生信工程师的能力,先略过。
我们直接看最精彩的网页工具,里面每个条目里面的都是一些成熟的资源:
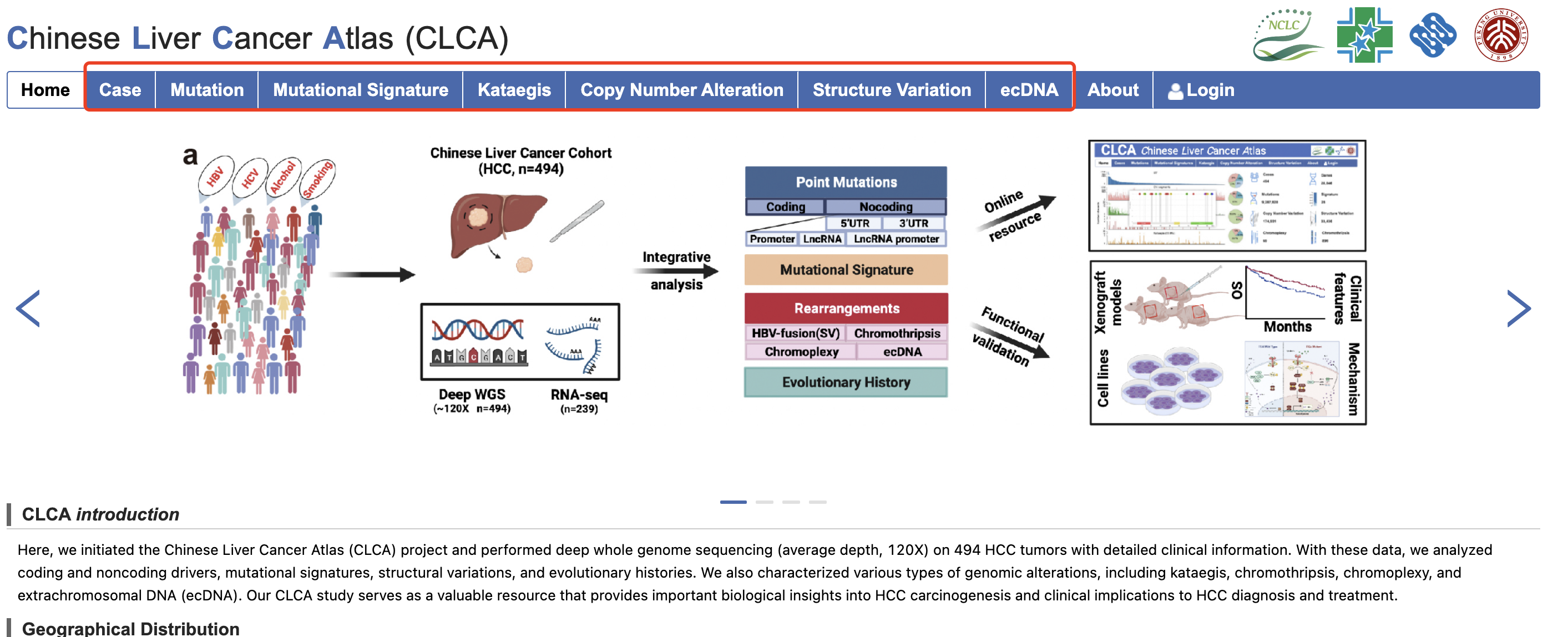
我简单的下载看了看:ls -lh |cut -d" " -f6- 29K 2 15 09:09 Cases_20240215.xlsx 13M 2 15 09:16 Copy_Number_Alteration_20240215.xlsx 36K 2 15 09:17 EcDNA_Detail_20240215.xlsx 27K 2 15 09:14 Kataegis_20240215.xlsx 6.5K 2 15 09:14 Mutational_Signatures_20240215.xlsx 20M 2 15 09:13 Mutations_20240215.xlsx 2.2M 2 15 09:16 Structure_Variation_20240215.xlsx网页里面提供的文件,足够大家做各式各样的下游分析了,基本上就跟 tcga数据挖掘类似的,比如下面的需求:
- 1、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)的预后(生存曲线)
- 2、探索各类肿瘤与对照的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(箱线图)
- 3、探索各类肿瘤与对照的全局(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(差异分析流程)
- 4、探索各类肿瘤中两个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平相关性(散点图)
- 5、探索各类肿瘤中多个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平总结(热图)
- 6、探索各类肿瘤中单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)与所有其它分子相关性并且排序
- 7、探索各类肿瘤中单个基因突变或者单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的预后(生存曲线)
- 8、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)分组后的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)特性的分布
来一个简单的学徒专业吧,读取那个Cases_20240215.xlsx文件,然后看看 Province ,Gender ,Age, Hepatitis Cirrhosis/Fibrosis BCLC ,Edmondson ,Smoking, Alcohol ,Multiple lesions ,这些临床性状是否有统计学显著的生存意义哦,绘制好生存分析曲线哈。目前没有结合单细胞或者gwas
其实通读全文,这个nature文章在肿瘤全基因组数据分析层面已经是非常完善了,在附件都有详细的方法学描述:

比如基因组重排就涉及到: - copy number alterations (CNAs),
- structural variations (SVs),
- HBV integrations,
- ecDNAs,
- three forms of clustered alterations, kataegis, chromothripsis, and chromoplexy
每个分析点都是大量的 图表,而作为一个nature文章也不可能说什么都来一壶,其实可以衍生的分析还有很多很多,比如结合单细胞或者gwas,就有很多结果,留给大家发挥啦!肝癌其它多组学
在咱们中国大陆,其实并不缺肝癌相关的病人和样品,所以早期已经是有了大量的其它组学队列研究,比如2019-CELL-复旦大学-多组学-蛋白质,文章是:《Integrated Proteogenomic Characterization of HBV- Related Hepatocellular Carcinoma》
如果大家有肝癌方面的疾病背景知识,其实可以系统性梳理一下相关的资源,做数据库网页工具合辑,做更高维度的挖掘。起码这个nature文章就很少涉及到他们的肿瘤基因组数据和转录组数据的联合分析。其它肿瘤相关多组学资源(中国人群的)
如果能想清楚上面的nature文章的数据的挖掘思路,其实就可以很容易迁移到其它癌症啦,类似的公开的资源真的是数不胜数。而且很多队列研究是早期的,那个时候人类遗传资源保护并不是很严格,都是有测序原始数据公开的哦:
中国人群的三阴性乳腺癌多组学队列 - Fudan University Shanghai Cancer Center Triple Negative Breast Cancer (FUSCCTNBC)
- https://www.ncbi.nlm.nih.gov/sra/?term=SRP157974
- https://www.ebi.ac.uk/ena/browser/view/PRJNA486023
中国人群的肾癌多组学队列 - https://www.ebi.ac.uk/ena/browser/view/PRJNA596338 转录组
- https://www.ebi.ac.uk/ena/browser/view/PRJNA596359 肿瘤外显子
中国人群的肺癌队列: - 源于文章:《Comprehensive genomic and immunological characterization of Chinese non-small cell lung cancer patients 》
当然了,也不一定要仅限于中国人群啦,也可以看其它国家地区, 比如德国 Prostate Cancer Systems Biology,2020年12月的文章,是:《Convergent network effects along the axis of gene expression during prostate cancer progression》, - https://www.ebi.ac.uk/ena/browser/view/PRJNA579899 转录组
- https://www.ebi.ac.uk/ena/browser/view/PRJNA577801 肿瘤外显子