看到了《单细胞天地》公众号分享了一个中山大学孙逸仙纪念医院的新鲜出炉的单细胞文章:PDGFRα+ITGA11+成纤维细胞通过 ITGA11-SELE 相互作用促进早期癌症的淋巴血管侵袭和淋巴转移,是13个scRNA样本,包括4个癌旁,6个淋巴血管侵犯(LVI)positive,3个LVI negative。对应的数据集是:GSE222315
很容易整理它们后读取,常规的单细胞转录组降维聚类分群代码可以看 :链接: https://pan.baidu.com/s/1bIBG9RciAzDhkTKKA7hEfQ?pwd=y4eh ,基本上大家只需要读入表达量矩阵文件到r里面就可以使用Seurat包做全部的流程。我们进行如下所示的第一层次降维聚类分群,可以看到每个单细胞亚群其实都还算是泾渭分明的:
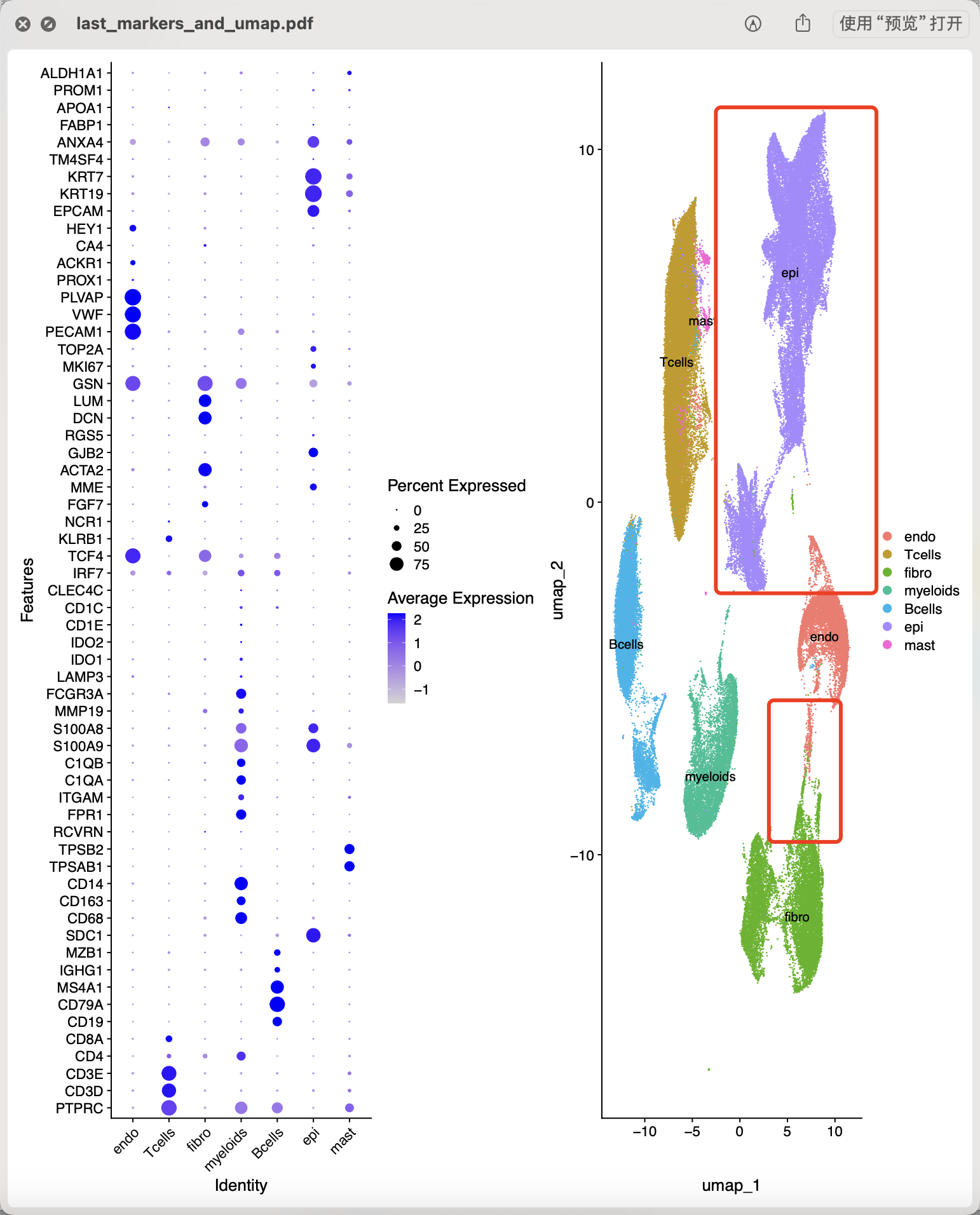
但是,如果我们提取里面的成纤维细胞亚群后,重新进行降维聚类分群,可以看到里面仍然是可以区分出来 t细胞,髓系免疫细胞,内皮细胞,以及上皮细胞 :
这个成纤维亚群里面可以细分出来内皮细胞是可以理解的,因为的文章的第一层次降维聚类分群就可以看到其实是成纤维和内皮细胞是有混杂的。但是为什么有 t细胞和髓系免疫细胞呢,以及上皮细胞。而且这些混杂的细胞亚群并没有具体的样品来源偏好性。从质量控制图也看不出来特殊之处。
上面的图如果看标记基因,可以得到4是t细胞,5是髓系,7是内皮细胞,8是上皮细胞。而且它们都略微有有一点点成纤维细胞亚群特异性基因的高表达,这就是为什么它们会在第一层次降维聚类分群的时候被错误的分配到成纤维亚群里面吧。
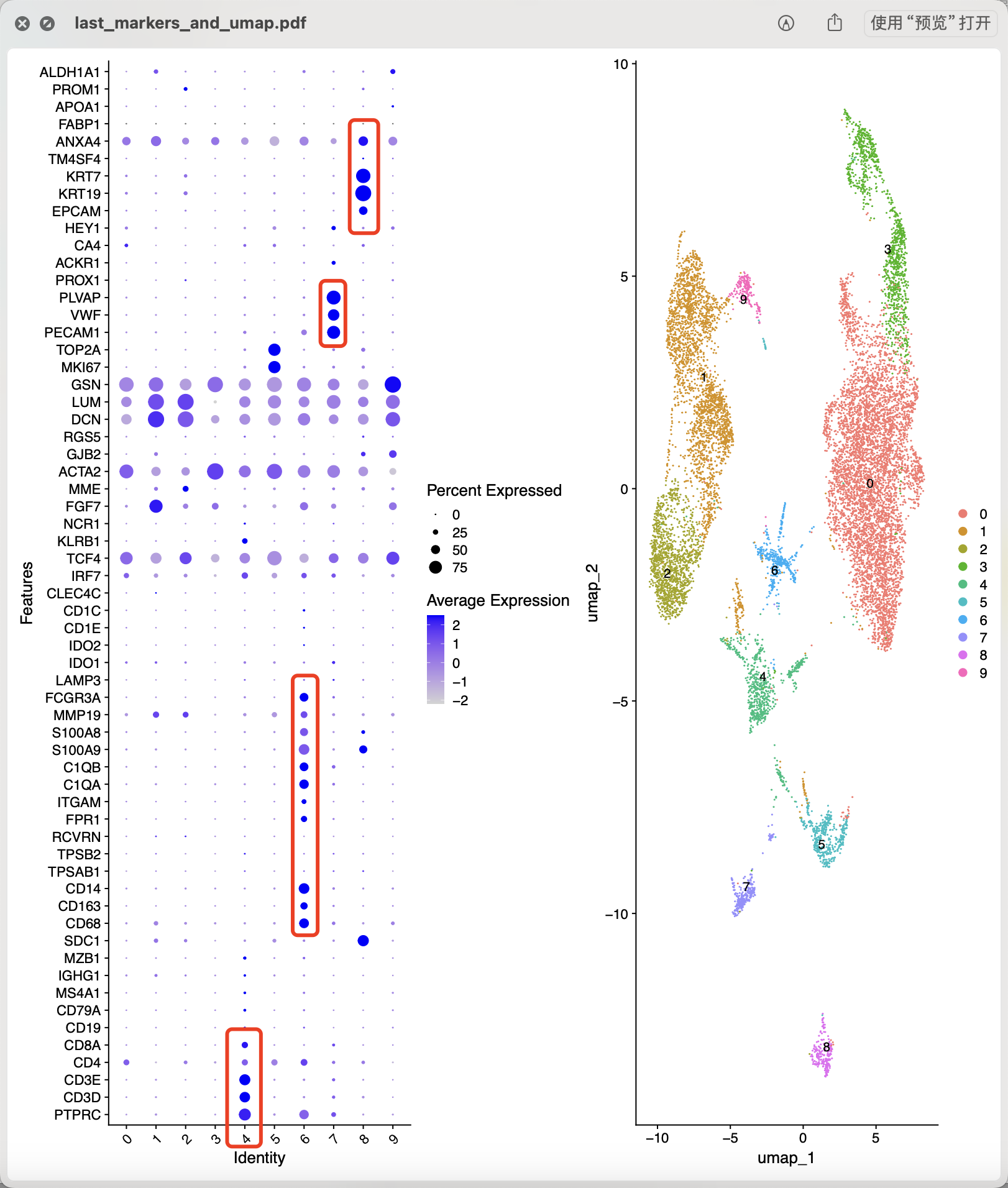
同理,我们针对第一层次降维聚类分群里面的上皮细胞进行细分的时候,也是可以看到里面有t和b这样的淋巴系免疫细胞 :

这个时候可以看到2是t细胞而6是b细胞亚群,它们并没有高表达上皮细胞的特异性基因,但是它们之所以在第一层次降维聚类分群的时候被错误的分配到上皮细胞亚群,其实是因为它们处于增殖这个状态而已,而我们在第一层次降维聚类分群的时候把编号为6的增殖亚群全部分配给了上皮细胞,所以细分上皮细胞的时候就能看到增殖亚群里面的细微的淋巴系免疫细胞啦。

而且可以看到上面的2和8是两种截然不同的b细胞哦,我们在第一层次降维聚类分群的时候其实把它们统一成为了b细胞哈!因为是纯手工的人为的主观的定义亚群:
#定义细胞亚群
celltype[celltype$ClusterID %in% c( 2,8),2]='Bcells'
celltype[celltype$ClusterID %in% c( 1 ),2]='Tcells'
celltype[celltype$ClusterID %in% c( 4 ),2]='myeloids'
celltype[celltype$ClusterID %in% c(5),2]='endo'
celltype[celltype$ClusterID %in% c(9),2]='mast'
celltype[celltype$ClusterID %in% c(3,7),2]='fibro'
celltype[celltype$ClusterID %in% c(0,6),2]='epi'
而且每个亚群的细分,我们都有标准代码啦,如下所示 :
rm(list=ls())
options(stringsAsFactors = F)
source('../../scRNA_scripts/lib.R')
###### step1:导入数据 ######
sce.all.int = readRDS('../../2-harmony/sce.all_int.rds')
colnames(sce.all.int@meta.data)
load('../../phe.Rdata')
table(phe$celltype)
sce.all=sce.all.int[,phe$celltype %in% c('epi' ) ]
sce.all=CreateSeuratObject(
counts = sce.all@assays$RNA$counts,
meta.data = sce.all@meta.data
)
同理,我们对其它的每个单细胞亚群(髓系免疫细胞,内皮细胞,b细胞,t细胞等等)都可以继续细分,但是只要是细分就会发现里面仍然是会混入其它亚群。而且大家混入的原因都不一样,有因为处于同样的生物学过程而混入,比如增殖,也有是因为确实表达了多种细胞亚群特异性基因而被混入,可能是双细胞,也有可能是全新的发现。