看到了一个2022的单细胞转录组文章:《Decoding the multicellular ecosystem of vena caval tumor thrombus in clear cell renal cell carcinoma by single-cell RNA sequencing》,纳入了 19 surgical tissue specimens (discovery cohort) from treatment-naïve ccRCC patients (ccRCC, n = 8) ,分别是;
- Tumor thrombus (n = 8),
- paired primary tumors (n = 8)
- adjacent renal tissues (n = 3)
总共是14万多细胞,分别在 62,035 cells from TTs, 58,695 cells from PTs, and 20,075 cells from ARTs ,第一层次降维聚类分群如下所示:
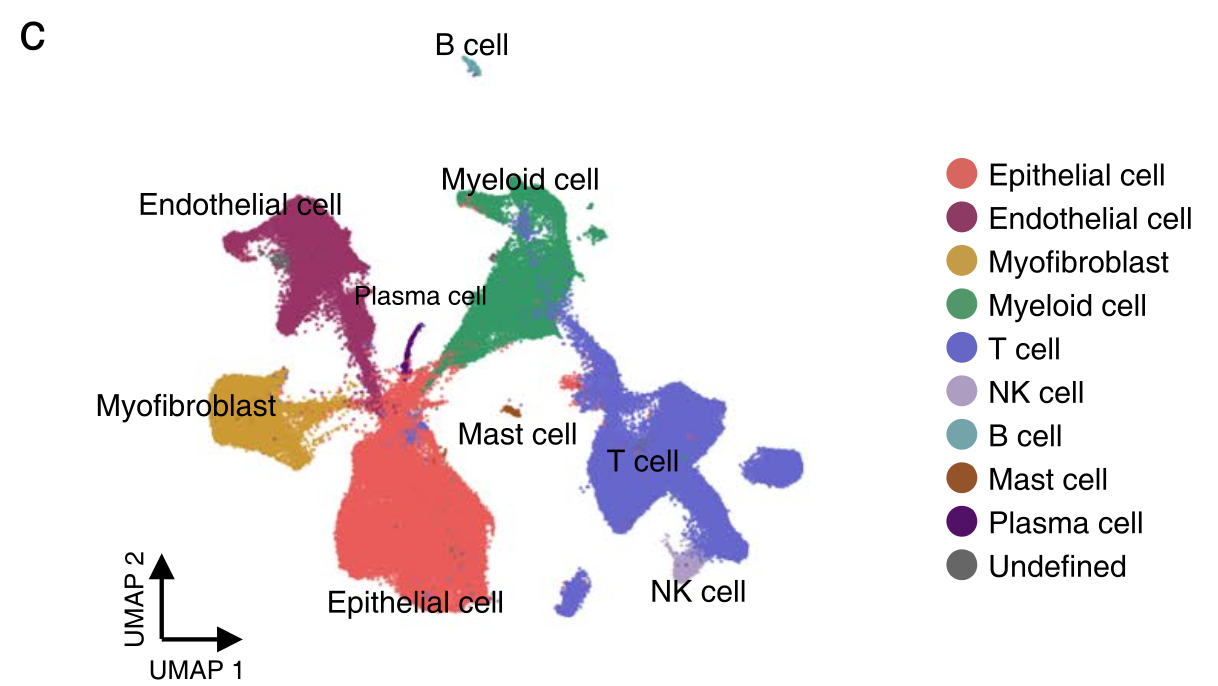
如果是肿瘤领域的我们通常是进行如下所示的分类: - immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibro or CD31+,PECAM1,endo)
参考我五年前介绍过的 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的 fibro 和endo进行细分,并且编造生物学故事的。而且我们已经积累了心肝脾肺肾等多个器官的上皮细胞的细分亚群, 以及免疫细胞里面的髓系和B细胞细分亚群: - B细胞细分亚群
- 髓系免疫细胞细分亚群
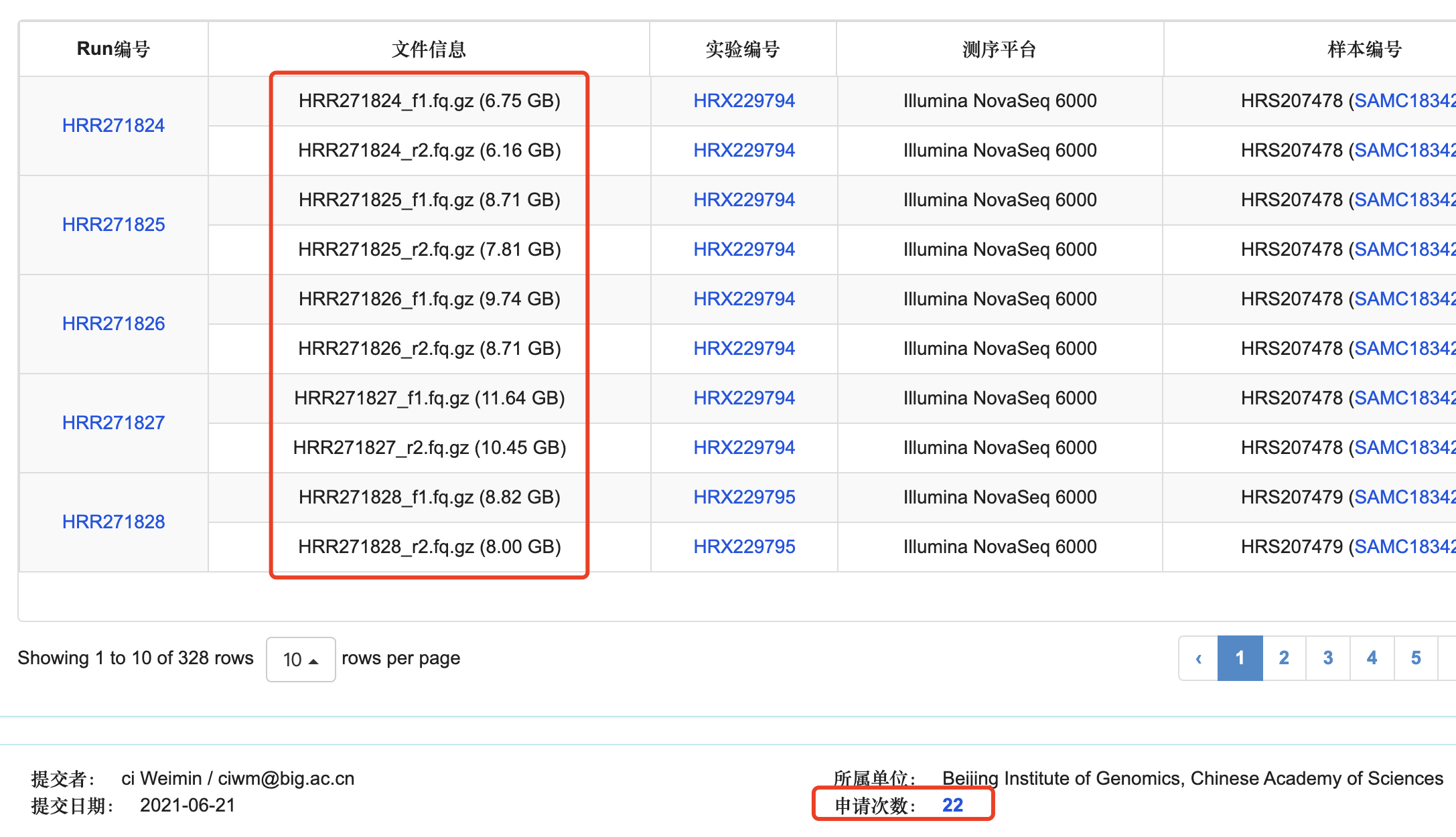
可以看到文献里面的数据在,链接是:https://ngdc.cncb.ac.cn/gsa-human/browse/HRA000963
上面标记的很清楚是 Controlled access , 而且可以看到页面记录了有22次申请,如下所示:
我就搜索了这个id在谷歌学术,确实是发现了有对应的数据挖掘文章:《Histone demethylase KDM4B contributes to advanced clear cell renal carcinoma and association with copy number variations and cell cycle progression》是2023年3月发表的,单位是上海的交通大学,跟前面的数据产出单位不一样。
说明了咱国产生信数据库肯定是可以申请成功的,只需要按照网页介绍的流程走下去即可。
就是好奇怪的这个数据挖掘文章提到了他们使用的是 single- cell transcriptome sequencing data of 29,831 ccRCC cells from eight patients were enrolled in our analysis. 而原文明明说的8个样品起码有六万多个单细胞啊,凭空就少了一半,有点尴尬。而且这个数据挖掘文章的降维聚类分群有有点尴尬,给一个第一层次的生物学命名有那么困难吗?文章里面就出现了一些单细胞亚群编号,是不是有点弱啊!
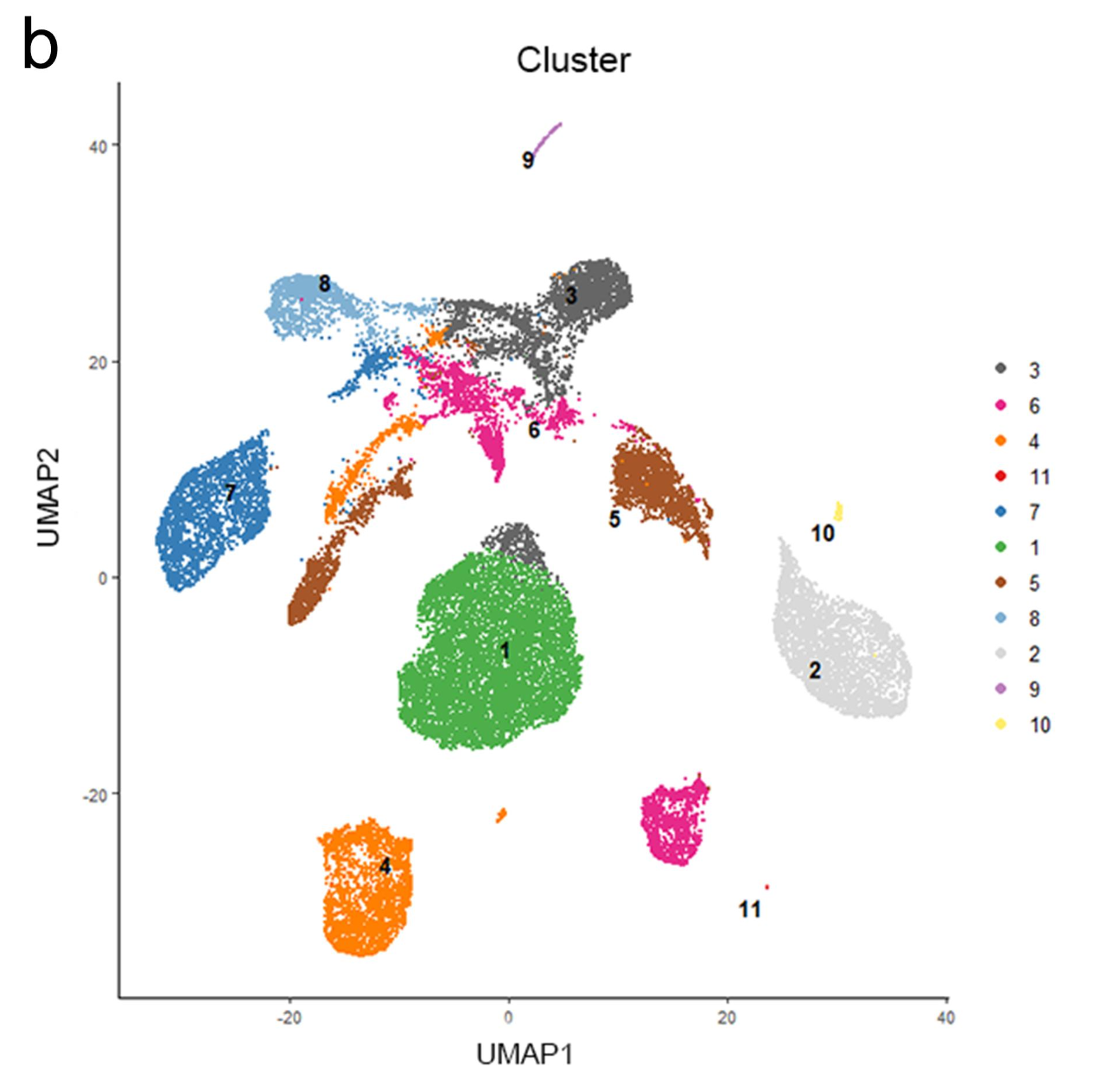
当然了,如果文献对应的数据集本身是公开的就更好了,就无需走这个申请审核的流程,应该是会更加促进科研交流,起码可以提高文献的引用情况哈, 比如下面的这些数据集的全流程处理: