有小伙伴看了我们在单细胞天地公众号推荐的文献:多组学来量化原位和侵袭的肢端黑色素瘤差异后,发现了里面的免疫细胞的髓系免疫细胞里面的巨噬细胞的一个图没有意思,求组想知道如何分析拿到数据并且可视化。
如下所示的巨噬细胞极化的打分结果散点图:
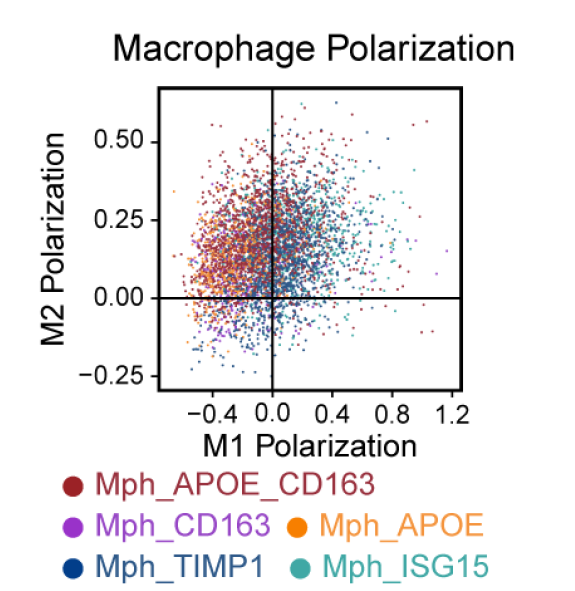
简单的读一下原文就可以看到:
- Myeloid cells were extracted and re-clustered to define subgroups in detail. Subclusters were identified at the resolution 0.9, including macrophages (CD68, CD163), 等髓系免疫细胞
- Macrophages were further divided into five subsets, including Mph_APOE, Mph_CD163, Mph_APOE_CD163, Mph_ISG15, and Mph_TIMP1. 这个时候巨噬细胞细分就没有明确的生物学名字,都是靠高表达量基因来区分它们。
- M1 and M2 polarization scores of macrophages were calculated by the ‘AddModuleScore’ function
巨噬细胞极化现象在单细胞水平其实很难区分
很早之前我们就总结过虽然虽然M1和M2的分类深入人心,但是在单细胞水平里面正确的做法可能是放弃M1和M2,详见:M1和M2的巨噬细胞差异就在CD86和CD163吗,很多单细胞文章都表明了巨噬细胞的M1和M2极化相关基因在单细胞水平是正相关,比如:
- https://www.sciencedirect.com/science/article/pii/S0092867418307232
- https://www.biorxiv.org/content/10.1101/566968v1.full
详见:【巨噬细胞新分类体系(放弃传统M1和M2)】,一切都是数据结果合理的挑选和解释而已。大家在自己的单细转录组数据降维聚类分群的时候也可以看到CXCL9 and SPP1的排他性,跟前面提到的TREM2联合SPP1去和FOLR2基因的排他性类似的。另外,我们通常是并不会选择提高分辨率这个手段来获取精细的单细胞亚群,而是取巨噬细胞子集后,继续进行降维聚类分群后再命名的策略。参考:取单细胞亚群子集细分的时候一定会出现干扰亚群(所以不要惊慌),,这样的话你就可以看到细胞亚群里面的混杂因素,而且可以手动删除到干扰因素。
之前我在生信技能树视频号直播一个文章的单细胞转录组数据(GSE208706)处理,文章是:《Amphiregulin from regulatory T cells promotes liver fibrosis and insulin resistance in non-alcoholic steatohepatitis》,就是基本的降维聚类分群而已,几分钟就搞定了,里面也有巨噬细胞的细分情况。
首先呢,需要有巨噬细胞极化的基因列表,比如我曾经看到过的文献里面就给出来了如下所示的:

代码演示这个巨噬细胞极化的打分结果散点图
首先需要一个Seurat对象,并且是已经是降维聚类分群成功了的哦,如下所示:
library(SeuratData) #加载seurat数据集
getOption('timeout')
options(timeout=10000)
# InstallData("pbmc3k")
data("pbmc3k")
sce <- UpdateSeuratObject(pbmc3k)
table(sce$seurat_annotations)
library(tidyverse)
sce <- sce %>% NormalizeData %>% FindVariableFeatures %>% ScaleData
然后就打分,然后散点图:
gl = list(
Tcells = c( 'CD3D', 'CD3E', 'CD4','CD8A'),
Bcells = c( 'CD19', 'CD79A', 'MS4A1')
)
# AddModuleScore 得到的是每个细胞中算出来的我们感兴趣的基因的表达均值
sce = AddModuleScore(object = sce,features = gl )
colnames(sce@meta.data)
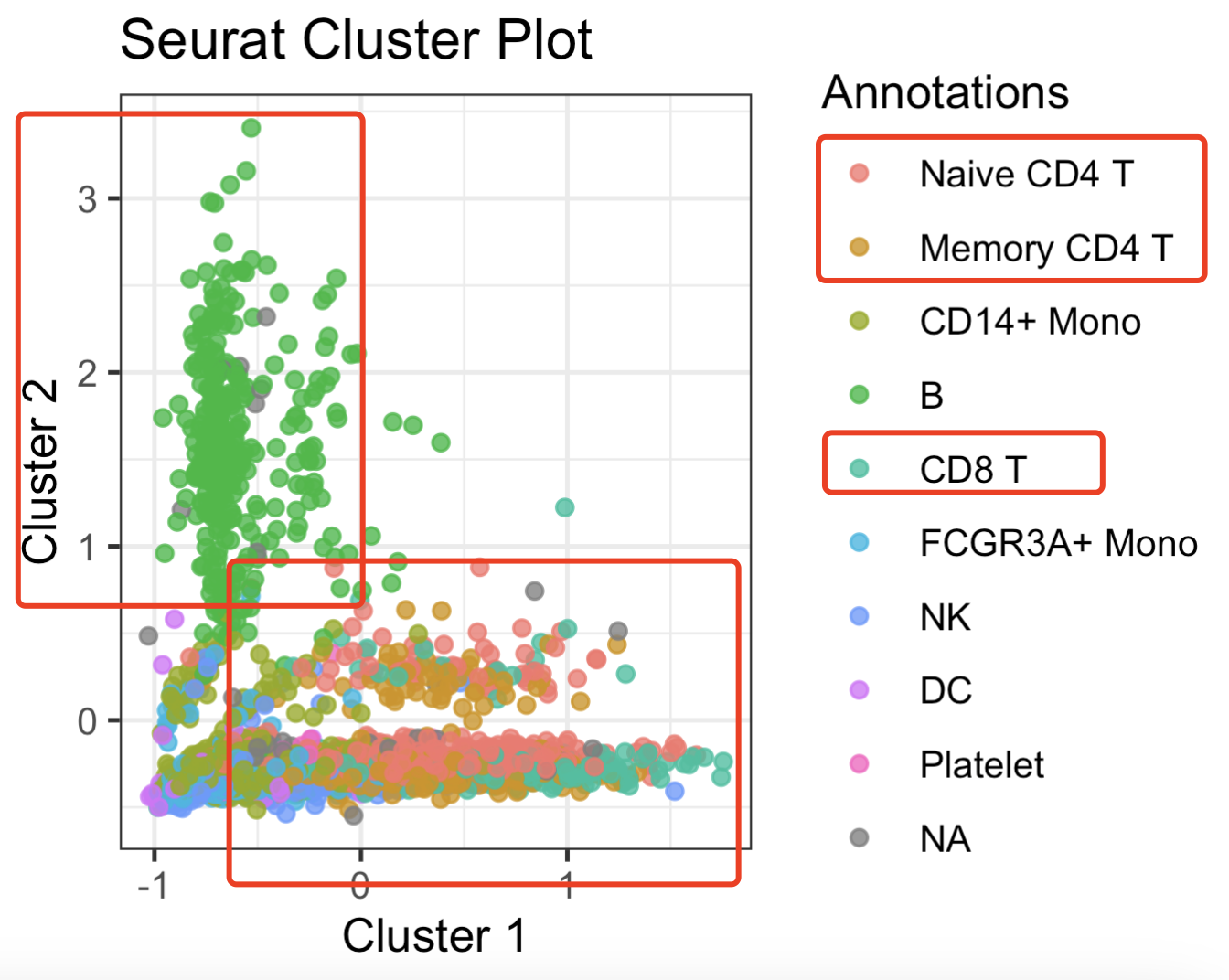
plot(sce$Cluster1,sce$Cluster2,col=sce$seurat_annotations)
当然了,图可以美化,就看大家自己的能力了,很明显的是t细胞和b细胞打分当然是倾向于在t细胞和b细胞里面显著,如下所示:
这个时候我们还可以借助于人工智能大模型了解一下巨噬细胞极化是巨噬细胞在不同微环境中对特定信号的响应而表现出不同表型和功能状态的过程。巨噬细胞是免疫系统的关键组成部分,具有多种功能,包括吞噬病原体、组织修复、免疫调节等。巨噬细胞极化通常分为两大类:M1型(经典激活型)和M2型(替代激活型),但实际的极化状态可能更为复杂和多样。
M1型巨噬细胞(经典激活型)
- 由促炎细胞因子如干扰素-γ (IFN-γ) 和脂多糖 (LPS) 激活。
- 表现出促炎和抗肿瘤的特性。
- 产生高浓度的活性氧 (ROS) 和一氧化氮 (NO)。
- 参与病原体清除和启动免疫应答。
M2型巨噬细胞(替代激活型)
- 由抗炎细胞因子如白介素-4 (IL-4)、白介素-13 (IL-13) 和免疫球蛋白G (IgG) 激活。
- 表现出抗炎、促进组织修复和免疫抑制的特性。
- 参与组织愈合、抑制炎症和调节免疫反应。
巨噬细胞极化的基因列表
巨噬细胞极化涉及多种基因的表达变化,以下是一些代表性的基因:
M1型巨噬细胞相关基因:
- TNF-α (Tumor Necrosis Factor-alpha):促炎细胞因子。
- IL-1β (Interleukin-1 beta):促炎细胞因子。
- IL-6 (Interleukin-6):调节免疫反应。
- IL-12:促进Th1型免疫应答。
- iNOS (Inducible Nitric Oxide Synthase):产生一氧化氮。
- COX-2 (Cyclooxygenase-2):参与炎症反应。
M2型巨噬细胞相关基因:
- IL-10 (Interleukin-10):抗炎细胞因子。
- CD206 (Cluster of Differentiation 206):参与抗原呈递。
- CD163:清除循环中的血红蛋白。
- Arg1 (Arginase-1):参与L-精氨酸代谢。
- Fizz1 (Fibrosis-associated protein 1):与组织修复相关。
- MRC1 (Macrophage mannose receptor 1):参与病原体识别。
巨噬细胞极化的意义
巨噬细胞极化对于理解炎症、肿瘤、自身免疫疾病等多种病理过程至关重要。通过调节巨噬细胞的极化状态,可能有助于开发新的治疗策略。
注意事项
- 巨噬细胞极化是一个动态过程,受多种因素影响。
- 极化状态可能在不同疾病和生理条件下有所不同。
- 研究巨噬细胞极化通常需要综合考虑基因表达、蛋白质水平和细胞功能。
了解巨噬细胞极化的机制和相关基因列表有助于深入研究巨噬细胞在健康和疾病中的作用。
学徒作业
处理之前我们在生信技能树的视频号直播一个文章的单细胞转录组数据(GSE208706)处理,文章是:《Amphiregulin from regulatory T cells promotes liver fibrosis and insulin resistance in non-alcoholic steatohepatitis》,第一层次降维聚类分群后提前里面的髓系免疫细胞然后细分亚群,拿到里面的巨噬细胞后继续细分亚群然后进行如上所示的巨噬细胞极化的打分结果散点图。