前面我们分享了流式细胞这个技术在单细胞转录组课题的应用,详见:流式细胞筛选能保证多大程度的细胞亚群纯度呢,也就是说其实它并不能保证我们百分百获取的都是目标单细胞亚群从而对它进行细致的 探索。这一点也是大家总是在微信交流群提问,我才特意整理的:
那么流式细胞都删不掉的亚群有什么特殊之处吗?
比如 2022的文章:《IL-4-Induced Quiescence of Resting Naive B Cells Is Disrupted in Systemic Lupus Erythematosus》,虽然说是很清楚做了b细胞的流式细胞筛选哦,数据集是 GSE136035 ,但是里面仍然是有t细胞和髓系的混杂。甚至这个降维聚类分群结果看起来跟血液的pbmc区别也不是很大了,详见:流式细胞筛选能保证多大程度的细胞亚群纯度呢 里面的最后一个案例图:
GSM4039805 SLE#1_single_cell_RNAseq
GSM4039807 SLE#2_single_cell_RNAseq
GSM4039809 SLE#3_single_cell_RNAseq
GSM4039813 HC#1_single_cell_RNAseq
GSM4039815 SLE#2_T1_single_cell_RNAseq
GSM4039819 HC#1_T1_single_cell_RNAseq
接下来,让我们看看另外一个数据集:2019-GSE139360-3个SLE的患者pbmc单细胞 ,它是没有经过流式细胞处理的,完全的pbmc样品,如下所示:
GSM4217718 scRNA_SLE1_PBMC
GSM4217719 scRNA_SLE2_PBMC
GSM4217720 scRNA_SLE3_PBMC
也是同样的降维聚类分群,第一层次如下所示:
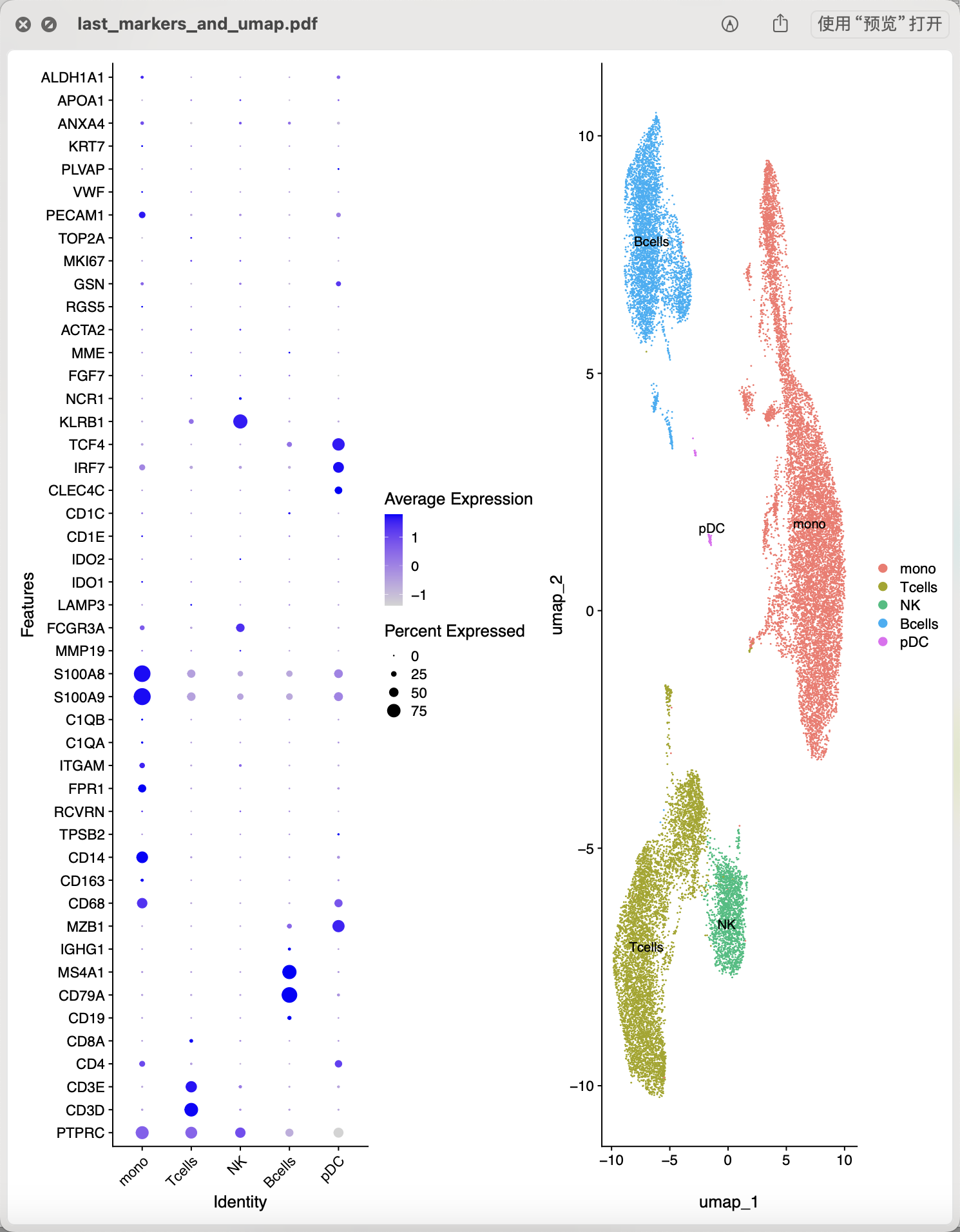
这两个数据集都是有SLE病人的单细胞转录组,而且都有巨噬细胞和单核细胞这样的髓系免疫细胞,以及t细胞和nk细胞。是不是可以看看它们的差异情况?
学徒作业
针对有流式细胞筛选的数据集 GSE136035 ,以及完全的pbmc数据集GSE139360,各自的降维聚类分群,然后看看它们的单核细胞是否有很大的区别。
另外在 公共数据库验证出来了就是对的吗 可以看到作者定义的 SSR4+ cluster 就很尴尬了其实是流式细胞技术的缺陷让b细胞里面混入了pDC这个单细胞亚群,参考:流式细胞筛选能保证多大程度的细胞亚群纯度呢。而上面的数据集:2019-GSE139360-3个SLE的患者pbmc单细胞 ,它是没有经过流式细胞处理的,完全的pbmc样品,也是有pDC的,也需要大家比较一下这两个pDC的区别哈!