最近听到了太多的声音,说不需要让临床医师做科研,耽误了他们的治病救人的本职工作不说,还破坏了纯洁的学术氛围。对于这样的说法我是嗤之以鼻的,因为没有临床实践提出来科学问题医学科研基本上都是空中楼阁啊!
哪怕是一个生命科学领域的博士生,没有上过临床,对科研的想象也是有限度的,比如大家都知道可以做癌症研究,普通人能想到的实验设计是可以探索癌症的生物学特性、机制、治疗响应以及疾病进展。以下是一些常见的实验设计类型,包括但不限于:
- 癌症与正常组织的差异分析:
- 比较癌症组织和正常组织的基因表达、蛋白质表达、代谢物水平等,以识别癌症特异性的生物标志物。
- 癌症转移与否的差异分析:
- 对原发性肿瘤和转移性肿瘤样本进行比较,以揭示与转移相关的分子机制和潜在靶点。
- 耐药与否的差异分析:
- 研究对治疗药物敏感的癌症细胞与耐药癌症细胞之间的差异,以了解耐药性的分子基础。
- 癌症发展阶段的差异分析:
- 比较癌症早期和晚期样本,以识别癌症进展过程中的分子变化。
- 癌症亚型之间的差异分析:
- 根据不同的癌症亚型(如乳腺癌的Luminal、Basal等)进行比较,以了解它们之间的生物学差异。
- 治疗响应与非响应的差异分析:
- 对接受相同治疗的患者样本进行比较,以区分治疗响应者和非响应者的特征。
- 细胞周期和凋亡分析:
- 研究癌症细胞在不同生物学过程中的变化,如细胞周期调控和程序性细胞死亡。
- 肿瘤微环境分析:
- 研究肿瘤细胞与其周围基质细胞、免疫细胞和细胞外基质的相互作用。
- 癌症干细胞研究:
- 探索癌症干细胞的特性,以及它们在癌症发生、发展和治疗中的作用。
- 基因编辑和功能验证:
- 使用CRISPR-Cas9等基因编辑技术敲除或敲入特定基因,以研究其功能和在癌症中的作用。
- 药物筛选和组合治疗:
- 筛选潜在的抗癌药物,并研究不同药物组合的协同效应。
- 患者来源的异种移植(PDX)模型:
- 使用患者来源的肿瘤细胞在免疫缺陷小鼠上建立模型,以研究肿瘤生物学和评估治疗方法。
- 生物信息学和系统生物学分析:
- 利用计算方法分析大规模的生物数据,构建癌症的系统生物学模型。
- 临床样本的纵向研究:
- 收集患者在治疗前后的样本,以研究疾病动态变化和治疗的影响。
- 跨疾病比较研究:
- 比较不同类型癌症之间的分子特征,以发现共同的生物学机制。
这些实验设计有助于全面理解癌症的复杂性,并为开发新的诊断工具、治疗策略和预防措施提供科学依据。
但是如果没有上过临床,仅仅是能根据文献看到了癌症的发生发展情况以及名字而已:
一个人怎么可能凭空想象到肝细胞癌(HCC)中的微血管侵犯(MVI)是指肿瘤细胞侵入肿瘤周围小血管(毛细血管、小静脉或血窦)内腔的现象。MVI是HCC患者肿瘤复发和预后不良的重要风险因素。理解和识别MVI对制定治疗决策和预测结果至关重要,比如2023年6月份的单细胞文献:《Single-cell dissection of the multicellular ecosystem and molecular features underlying microvascular invasion in HCC》,做了单细胞转录组数据(46,789 individual cells from 10 samples of MVI+ (MVI present) and MVI- (MVI absent) patients), 对应的数据集是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE242889
GSM7774394_1NT_P21.tar.gz 83.3 Mb
GSM7774395_2NT_P24.tar.gz 102.8 Mb
GSM7774396_3NT_P25.tar.gz 34.4 Mb
GSM7774397_4NT_P29.tar.gz 173.6 Mb
GSM7774398_5NT_P36.tar.gz 76.3 Mb
GSM7774399_1T_C21.tar.gz 122.1 Mb
GSM7774400_2T_C24.tar.gz 208.8 Mb
GSM7774401_3T_C25.tar.gz 161.8 Mb
GSM7774402_4T_C29.tar.gz 203.0 Mb
GSM7774403_5T_C36.tar.gz 146.4 Mb
大家可以读取上面的表达量矩阵文件,只需要下载上面的每个样品的压缩包文件解压即可:
tree -h
[ 416] .
├── [ 160] 1NT_P21
│ ├── [145K] barcodes.tsv
│ ├── [639K] genes.tsv
│ └── [ 83M] matrix.mtx
├── [ 160] 1T_C21
│ ├── [146K] barcodes.tsv
│ ├── [682K] genes.tsv
│ └── [121M] matrix.mtx
├── [ 160] 2NT_P24
│ ├── [143K] barcodes.tsv
│ ├── [662K] genes.tsv
│ └── [102M] matrix.mtx
├── [ 160] 2T_C24
│ ├── [156K] barcodes.tsv
│ ├── [683K] genes.tsv
│ └── [208M] matrix.mtx
├── [ 160] 3NT_P25
│ ├── [ 68K] barcodes.tsv
│ ├── [1.4M] genes.tsv
│ └── [ 33M] matrix.mtx
├── [ 160] 3T_C25
│ ├── [177K] barcodes.tsv
│ ├── [1.4M] genes.tsv
│ └── [160M] matrix.mtx
├── [ 160] 4NT_P29
│ ├── [203K] barcodes.tsv
│ ├── [713K] genes.tsv
│ └── [173M] matrix.mtx
├── [ 160] 4T_C29
│ ├── [205K] barcodes.tsv
│ ├── [704K] genes.tsv
│ └── [202M] matrix.mtx
├── [ 160] 5NT_P36
│ ├── [131K] barcodes.tsv
│ ├── [1.4M] genes.tsv
│ └── [ 75M] matrix.mtx
└── [ 160] 5T_C36
├── [156K] barcodes.tsv
├── [1.4M] genes.tsv
└── [145M] matrix.mtx
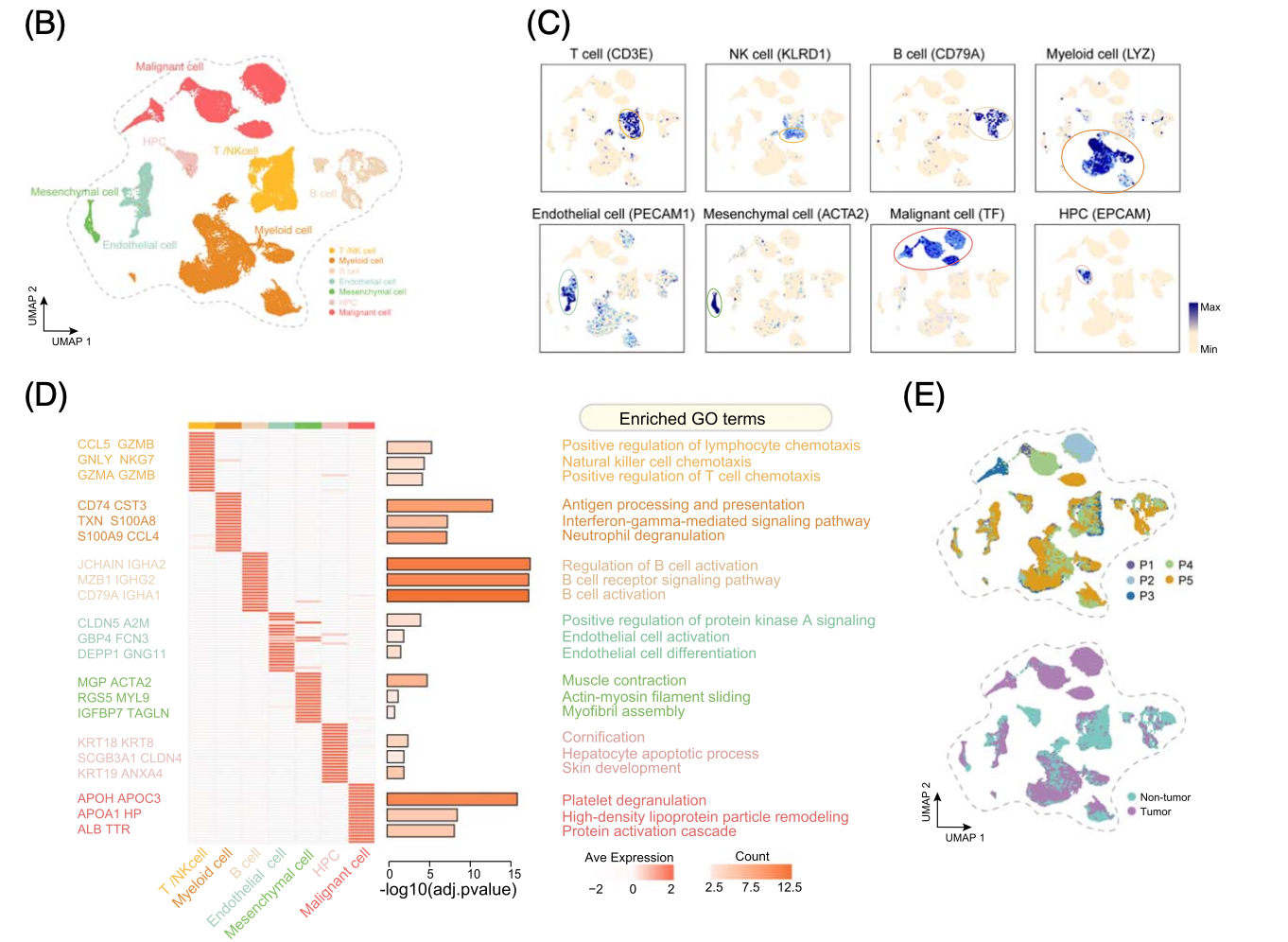
然后一个简简单单的循环读取即可走Seurat的降维聚类分群啦,比如文章里面的第一层次降维聚类分群:
- 7,419 T/NK T cell and NK cell cells (CD3E and killer cell lectin like receptor D1)
- 3,982 B cells (CD79A)
- 18,164 myeloid cells (lysozyme)
- 3,258 endothelial cells (plate- let and endothelial cell adhesion molecule 1)
- 1,008 mesenchymal cells (Actin Alpha 2)
- 1,506 hepatic progenitor cells [hepatic progenitor cells (HPCs)
- epithelial cell adhesion molecule] and 11,452 malignant cells (Transferrin)
如下所示展示单细胞亚群分类和标记基因的结果:
然后就是每个单细胞亚群细致的探索罢了,做文章类似的生物信息学分析:
- preprocessing and quality control of scRNA- seq data
- cell type determination and marker gene signature identification
- doublets removal
- collection and calculation of functional gene module signatures and scores
- pathway analysis
- Single-Cell rEgulatory Network Inference and Clustering analysis
- cell developmental trajectory
- cell-cell communication analysis
- copy number variation analysis from scRNA-seq
- infiltration estimation of cell subsets and infiltration estimation of the CLAMT