生物信息学是一个交叉学科,结合了生物学、计算机科学和信息技术,用于处理和分析生物数据,特别是大规模的组学数据。
这里给大家推荐一下一本关于生物信息学(Bioinformatics)的专著,专注于组学(Omics)技术及其数据分析,标题也是朴实无华哦:《Bioinformatics for Omics Data》,另外就是非常值得强调的是书籍居然是2011年的!

以下是对书籍内容的整理和介绍:
第一部分:组学生物信息学基础
- 组学技术、数据和生物信息学原理 - 介绍组学技术的基础知识和生物信息学的核心原理。
- 组学数据的数据标准 - 讨论数据标准在数据共享和重用中的重要性。
- 组学数据管理和注释 - 描述如何管理和注释组学数据。
- 跨组学研究项目中的数据和知识管理 - 探讨跨学科研究项目中数据管理的策略。
- 组学数据的统计分析原理 - 介绍适用于组学数据的统计分析基础。
- 连接组学数据级别的统计方法和模型 - 讨论如何通过统计方法整合不同级别的组学数据。
- 时间序列组学数据集的分析 - 分析时间序列数据集以揭示生物过程。
- -Omes的使用和滥用 - 讨论“-Omes”术语的使用,如基因组学、蛋白质组学等。
第二部分:组学数据和分析轨迹
- 高通量测序数据的计算分析 - 介绍高通量测序技术及其数据分析方法。
- 病例对照研究中单核苷酸多态性的分析 - 研究遗传变异与疾病之间的关系。
- 拷贝数变异数据的生物信息学 - 探讨拷贝数变异的检测和分析。
- 从扫描仪到浏览器的ChIP-Chip数据处理 - 描述ChIP-Chip实验的数据流程。
- 基因表达分析揭示全局机制和疾病 - 利用基因表达数据来理解生物学机制和疾病。
- RNomics的生物信息学 - 研究RNA层面的生物信息学,包括非编码RNA。
- 定性和定量蛋白质组学的生物信息学 - 探讨蛋白质组学数据分析。
- 基于质谱的代谢组学的生物信息学 - 描述代谢物的鉴定和定量。
第三部分:应用组学生物信息学
- 组学数据解释的计算分析工作流程 - 介绍组学数据分析的工作流程。
- 组学数据的整合、仓储和分析策略 - 讨论如何整合和分析大规模组学数据。
- 信号通路、互作组重建和功能分析的组学数据整合 - 整合不同组学数据以研究信号通路和功能。
- 来自时间依赖组学数据的网络推断 - 从时间序列数据推断生物网络。
- 组学与文献挖掘 - 结合组学数据和文献信息来提取生物学知识。
- 临床数据背景下的组学-生物信息学 - 讨论组学数据在临床医学中的应用。
- 基于组学的病理生理过程识别 - 利用组学数据来识别疾病相关的生物学过程。
- 基于组学的生物标记物发现的数据挖掘方法 - 应用数据挖掘技术来发现生物标记物。
- 癌症靶标识别的集成生物信息学分析 - 集成分析用于癌症治疗靶标的发现。
- 基于组学的分子靶标和生物标记物识别 - 识别新的药物靶标和生物标记物。
这本书籍为读者提供了从基础理论到高级应用的全面介绍,涵盖了组学数据分析的多个方面,适合生物信息学、系统生物学和相关领域的研究人员和学生阅读。
组学技术其实已经更新换代
上面的书籍其实是2011的了,那个时候我都还没有开始学习生物信息学。等我掌握了这些ngs技术的时候,我在生信技能树自媒体矩阵整理和分享了自己擅长的几乎全部的ngs组学数据处理,有文字版内容,以及视频在b站。
因为个人时间精力问题,我自己的b站课程仅仅是ngs多组学以及单细胞技术的教程。而且很多都是五六年前的了,比如下面这些:
- 免费视频课程《RNA-seq数据分析》
- 免费视频课程《WES数据分析》
- 免费视频课程《ChIP-seq数据分析》
- 免费视频课程《ATAC-seq数据分析》
- 免费视频课程《TCGA数据库分析实战》
- 免费视频课程《甲基化芯片数据分析》
- 免费视频课程《影像组学教学》
- 免费视频课程《LncRNA-seq数据》
- 免费视频课程《GEO数据挖掘》
现如今到2024,组学又更进一步了,比如:《Multi-scale signaling and tumor evolution in high- grade gliomas》,如下所示的 (B) Features quantified on 14 data platforms (excluding single-nuclei sequencing and multiplex imaging).
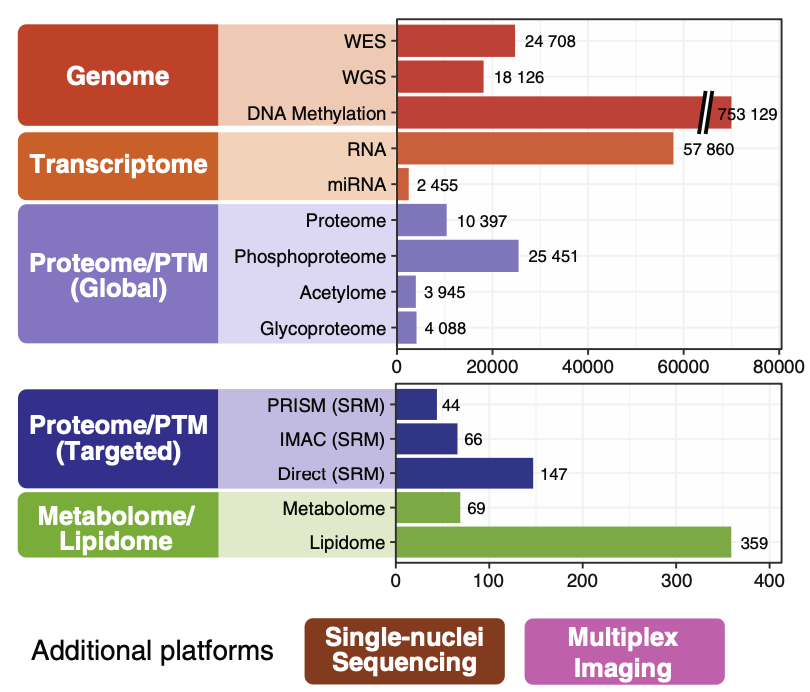
而且文章里面的数据也是公开可以获取的,分门别类整理好了 :
-
Genomic Data Commons (GDC): CPTAC WES, WGS, DNA-methylation
- 内容: 该链接指向国家癌症研究所(NCI)的基因组数据共享(GDC)项目,特别是针对临床蛋白质组学技术评估(CPTAC)计划的数据。这里包含了全外显子测序(WES)、全基因组测序(WGS)和DNA甲基化数据。
- 目的: 这些数据用于研究癌症基因组的变异、基因表达调控以及疾病相关基因的甲基化状态。
-
Proteomic Data Commons (PDC): CPTAC proteome, phosphoproteome, acetylome, glycoproteome, metabolome, and lipidome data
- 内容: 蛋白质组数据共享(PDC)项目包含了CPTAC计划中的蛋白质组、磷酸化蛋白质组、乙酰化蛋白质组、糖蛋白质组、代谢组和脂质组数据。
- 目的: 这些数据有助于理解蛋白质表达、翻译后修饰、代谢途径以及细胞信号传导在癌症中的作用。
-
Cancer Data Service (CDS): CPTAC multiome snATAC seq data
- 内容: 癌症数据服务(CDS)提供了CPTAC计划中的多组学,以及单细胞水平的ATAC测序数据。
- 目的: 这些数据用于研究染色质可及性、转录因子结合位点以及基因调控网络。
-
The Cancer Imaging Archive (TCIA): CODEX and histopathology images
- 内容: 癌症影像档案库(TCIA)包含了癌症相关的医学影像数据,如CODEX(癌症数字切片档案库)和组织病理学图像。
- 目的: 这些图像数据用于癌症的影像学研究、计算机辅助诊断和病理学分析。
这些数据链接提供了丰富的生物医学信息资源,对癌症生物学、精准医疗和转化研究具有重要价值。研究人员可以利用这些数据进行多维度的分析,以发现新的生物标记物、理解疾病机制和开发新的治疗方法。需要注意的是,访问和使用这些数据可能需要遵循特定的数据使用协议和隐私保护规定。
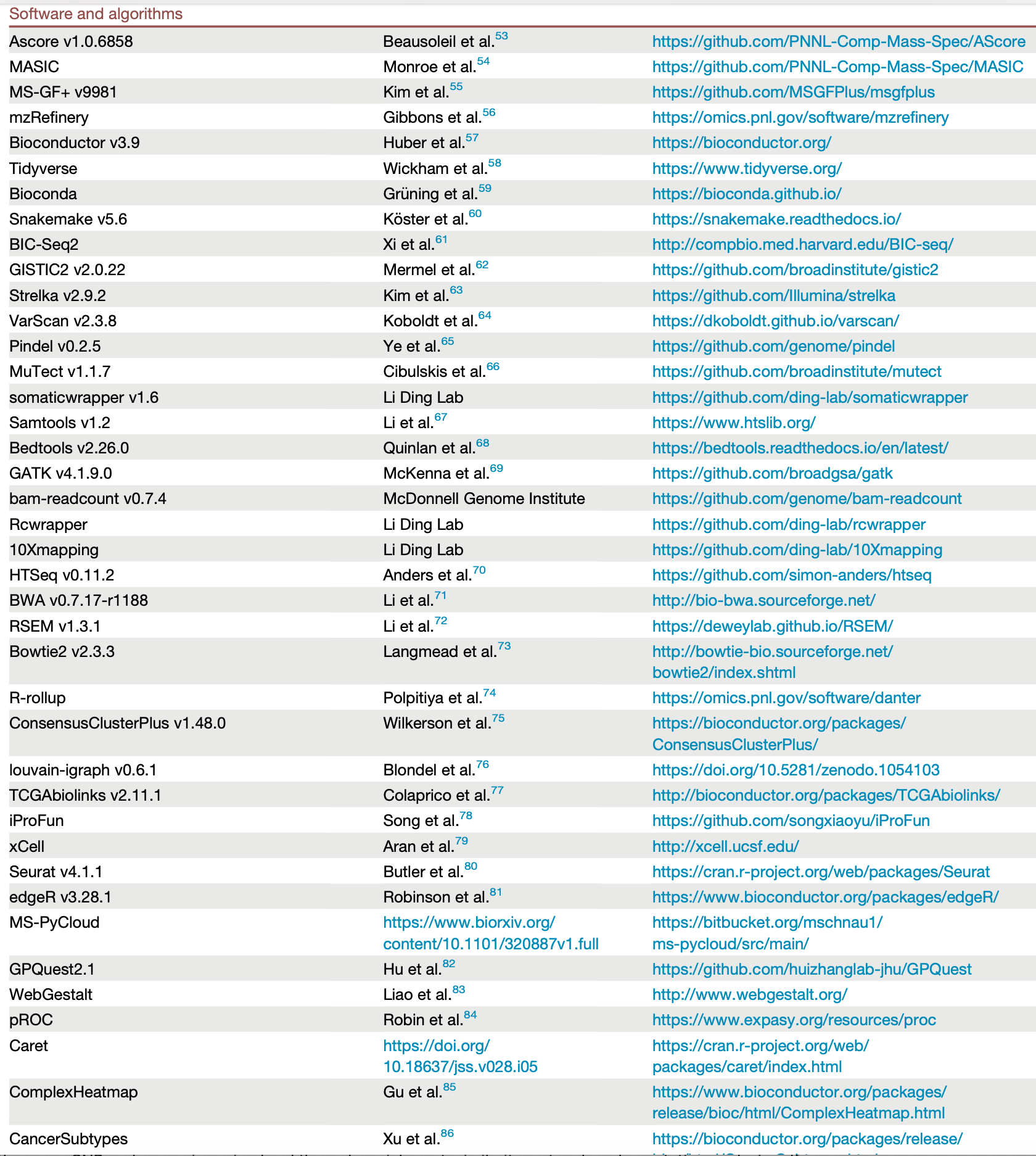
为了处理这些数据,涉及到的软件工具已经是高达几十个,一般来说只有比较大的课题组才能有足够数量的生信工程师能hold住这样的规模的复杂的数据: