如果只是画网络图,那么只需要把所有的点,按照算好的坐标画出来,然后把所有的连线也画出即可!
如果只是画网络图,那么只需要把所有的点,按照算好的坐标画出来,然后把所有的连线也画出即可!
接下来, 我们直接看看R里面是如何画网络图的,我们首推一个包:networkD3/
http://www.zhihu.com/question/19605599
> my_fun(10) [1] -2 2 2 0 [1] 2 3 1 1 [1] 0 4 2 1 [1] -4 5 3 1 [1] 4 6 1 2 [1] 6 7 1 3 [1] 8 8 1 4 [1] 6 9 2 4 [1] 2 10 3 4 [1] 10 11 1 5 [1] 8 12 2 5 [1] 8 如果我们赌11次,可以看到,我最后会剩余8块钱,每次输赢的情况都反应在里面了,可以自己模拟多次看看! 因为我只赌了11次,所以很快,如果我赌1000次,而且还想检验一下10000次模拟结果,就会比较慢了! 我首先使用进度条模拟一下结果,代码如下:还是比较慢的##Time difference of 1.861802 mins 我用了apply,好像时间是节省了一些,不过聊胜于无!
> library(pbapply) > start=Sys.time() > results=pbsapply(1:10000,function(i){my_fun(1000)}) |++++++++++++++++++++++++++++++++++++++++++++++++++| 100% > Sys.time()-start ##9.909524 secs Time difference of 1.301539 mins 那接下来我们分析一下模拟的结果吧
> summary(results) Min. 1st Qu. Median Mean 3rd Qu. Max. -64580 974 998 985 1020 1110 可以看到,我们平均下来是会赢钱的,而且赢面很大,赢的次数非常多!!! 但是,我们看下面的图就知道,一个很诡异的结果! 而且,我这里用的模拟胜负,并不是很好
这其实还只是小批量模拟,如果要模拟百亿次,首先我的笔记本肯定不行,cpu太破了,如果用服务器,就需要用并行计算啦! ##下面是用多核并行计算的代码,大家有兴趣可以自己去玩一下 library(parallel) cl.cores <- detectCores() cl <- makeCluster(cl.cores) clusterExport(cl, "judge") start=Sys.time() results=parSapply( cl=cl, 1:10000, my_fun(1000) ) Sys.time()-start ##4.260994 secs stopCluster(cl)
我们可以直接使用R的bioconductor里面的一个包,GOstats里面的函数来做超几何分布检验,看看每条pathway是否会富集
我们直接读取用limma包做好的差异分析结果
setwd("D:\\my_tutorial\\补\\用limma包对芯片数据做差异分析")
DEG=read.table("GSE63067.diffexp.NASH-normal.txt",stringsAsFactors = F)
View(DEG)
我们挑选logFC的绝对值大于0.5,并且P-value小雨0.05的基因作为差异基因,并且转换成entrezID
probeset=rownames(DEG[abs(DEG[,1])>0.5 & DEG[,4]<0.05,])
library(hgu133plus2.db)
library(annotate)
platformDB="hgu133plus2.db";
EGID <- as.numeric(lookUp(probeset, platformDB, "ENTREZID"))
length(unique(EGID))
#[1] 775
diff_gene_list <- unique(EGID)
这样我们的到来775个差异基因的一个list
首先我们直接使用R的bioconductor里面的一个包,GOstats里面的函数来做超几何分布检验,看看每条pathway是否会富集
library(GOstats)
library(org.Hs.eg.db)
#then do kegg pathway enrichment !
hyperG.params = new("KEGGHyperGParams", geneIds=diff_gene_list, universeGeneIds=NULL, annotation="org.Hs.eg.db",
categoryName="KEGG", pvalueCutoff=1, testDirection = "over")
KEGG.hyperG.results = hyperGTest(hyperG.params);
htmlReport(KEGG.hyperG.results, file="kegg.enrichment.html", summary.args=list("htmlLinks"=TRUE))
结果如下:
但是这样我们就忽略了其中的原理,我们不知道这些数据是如何算出来的,只是由别人写好的包得到了结果罢了。
事实上,这个包的这个hyperGTest函数无法就是包装了一个超几何分布检验而已。
如果我们了解了其中的统计学原理,我们完全可以写成一个自建的函数来实现同样的功能。
本来想着是如何习惯R里面的list对象,来实现hash的功能,无意中发现了居然还有这个包
https://cran.r-project.org/web/packages/hash/hash.pdf
我简单看了看文档,的确跟perl里面的hash功能类似,非常好用。
创建一个hash很简单
h <- hash( keys=letters, values=1:26 )
h <- hash( letters, 1:26 )
类似于下面的列表
L=setNames(as.list(LETTERS),1:26)
如果是列表要提取keys需要用names函数,如果需要提取values,需要用unlist函数,而用了hash之后,这两个函数就可以独自运行啦
还有很多其它函数,其实在list中也可以实现,主要是看你对哪种语法更熟悉,感觉自己现在编程能力差不多算是小有所成了,看什么都一样了,要是以前学R的时候看到了这个hash包,我肯定会很兴奋的!
clear signature(x = "hash"): Remove all key-value pairs from hash
del signature(x = "ANY", hash = "hash"): Remove specified key-value pairs from hash
has.key signature(key = "ANY", hash = "hash"): Test for existence of key
is.empty signature(x = "hash"): Test if no key-values are assigned
length signature(x = "hash"): Return number of key-value pairs from the hash
keys signature(hash = "hash"): Retrieve keys from hash
values signature(x = "hash"): Retrieve values from hash
copy signature(x = "hash"): Make a copy of a hash using a new environment.
format signature(x = "hash"): Internal function for displaying hash
AnnDbBimap对象是R里面的bioconductor系列包的基础对象,在探针数据里面会包装成ProbeAnnDbMap,跟go通路相关又是GOTermsAnnDbBimap对象。
但是它都是AnnDbBimap对象衍生过来的
主要存在于芯片系列的包和org系列的包,其实AnnDbBimap对象就是list对象的包装,比如下面这些例子:
ls("package:hgu133plus2.db")
| [1] "hgu133plus2" "hgu133plus2.db" "hgu133plus2_dbconn" |
| [4] "hgu133plus2_dbfile" "hgu133plus2_dbInfo" "hgu133plus2_dbschema" |
| [7] "hgu133plus2ACCNUM" "hgu133plus2ALIAS2PROBE" "hgu133plus2CHR" |
| [10] "hgu133plus2CHRLENGTHS" "hgu133plus2CHRLOC" "hgu133plus2CHRLOCEND" |
| [13] "hgu133plus2ENSEMBL" "hgu133plus2ENSEMBL2PROBE" "hgu133plus2ENTREZID" |
| [16] "hgu133plus2ENZYME" "hgu133plus2ENZYME2PROBE" "hgu133plus2GENENAME" |
| [19] "hgu133plus2GO" "hgu133plus2GO2ALLPROBES" "hgu133plus2GO2PROBE" |
| [22] "hgu133plus2MAP" "hgu133plus2MAPCOUNTS" "hgu133plus2OMIM" |
| [25] "hgu133plus2ORGANISM" "hgu133plus2ORGPKG" "hgu133plus2PATH" |
| [28] "hgu133plus2PATH2PROBE" "hgu133plus2PFAM" "hgu133plus2PMID" |
| [31] "hgu133plus2PMID2PROBE" "hgu133plus2PROSITE" "hgu133plus2REFSEQ" |
| [34] "hgu133plus2SYMBOL" "hgu133plus2UNIGENE" "hgu133plus2UNIPROT" |
那么我们可以随便挑选包中的一个数据分析一下
x <- hgu133plus2SYMBOL
xlist=as.list(x)
我们查看X对象,可知,它是object of class "ProbeAnnDbMap",而这个对象,就是常见的list对象,被包装了一下, 只有我们明白了它和list对象到底有什么区别,才算是真正搞懂了这一系列包
但是这个ProbeAnnDbMap对象,在GO等包里面会被包装成更复杂的对象-GOTermsAnnDbBimap,但是对他们的理解都大同小异。
我们先回顾一下在R语言里面的list的基础知识:
R 的 列表(list)是一个以对象的有序集合构成的对象。 列表中包含的对象又称为它的分量(components)。
分量可以是不同的模式或者类型, 如一个列表可以包括数值向量,逻辑向量,矩阵,复向量, 字符数组,函数等等
如果你会perl的话,可以把它理解成hash。
分量常常会被编号的(numbered),并且可以利用它来 访问分量
列表的分量可以被命名,这种情况下 可以通过名字访问。
特别要注意一下 Lst[[1]] 和 Lst[1] 的差别。 [[...]] 是用来选择单个 元素的操作符,而 [...] 是一个更为一般 的下标操作符。
因此前者得到的是列表 Lst 中的第一个对象, 并且含有分量名字的命名列表(named list)中分量的名字会被排除在外的。
后者得到的则是列表 Lst 中仅仅由第一个元素 构成的子列表。如果是命名列表, 分量名字会传给子列表的。
那么接下来我们就看看x和xlist的区别。
它们里面的数据都是affymetrix公司出品的人类的hgu133plus2芯片的探针ID与基因symbol的对应关系
如果我想拿到所有的探针ID,那么对于AnnDbBimap对象,需要用mappedkeys(x),对于普通的list对象,需要用names(xlist).
对于普通的list对象,如果我想看前几个元素,直接head就可以了,但是对于AnnDbBimap对象,数据被封装好了,需要先as.list,然后才能head
mapped_probes <- mappedkeys(x)
PID2=head(mapped_probes)
那么,如果我们想根据以下探针ID来查看它们在这些数据里面被对应着哪些基因symbol 呢?
> PID2 #这是一串探针ID,后面的操作都需要用的
[1] "1053_at" "117_at" "121_at" "1255_g_at" "1316_at" "1320_at"
如果是对于AnnDbBimap对象,我们可以用mget函数来操作,取多个探针的对应基因symbol
accnum <- mget(PID2, env=hgu133plus2ACCNUM);
gname <- mget(PID2, env=hgu133plus2GENENAME)
gsymbol <- mget(PID2, env=hgu133plus2SYMBOL)
mget函数返回的就是普通的list函数了,可以直接查看了。
如果是对于普通的list对象,我们想取多个探针的对应基因symbol也是非常简单的,xlist[PID2]即可。
那么我们不禁有问了,既然它们两个功能完全一样,何苦把它包装成一个对象了,我直接操作list对象不就好了,学那么多规矩干嘛?
所以,重点就来了
> length(mapped_probes)
[1] 42125
> length(names(xlist))
[1] 54675
看懂了吗?
但是,事实上用处也不大,你觉得下面这两个有区别吗?
SYMBOL <- AnnotationDbi::as.list(hgu133plus2SYMBOL)
SYMBOL <- SYMBOL[!is.na(SYMBOL)];
x <- hgu133plus2SYMBOL
mapped_probes <- mappedkeys(x)
SYMBOL <- AnnotationDbi::as.list(x[mapped_probes])
PS,在R里面创建一个list对象是非常简单的
The setNames() built-in function makes it easy to create a hash from given key and value lists.(Thanks to Nick K for the better suggestion.)
Usage: hh <- setNames(as.list(values), keys)
Example:
players <- c("bob", "tom", "tim", "tony", "tiny", "hubert", "herbert")
rankings <- c(0.2027, 0.2187, 0.0378, 0.3334, 0.0161, 0.0555, 0.1357)
league <- setNames(as.list(rankings), players)
经常会有人问这样的问题I have list of 10,000 Entrez IDs and i want to convert the multiple Entrez IDs into the respective gene names. Could someone suggest me the way to do this?等等类似的基因转换,能做的基因转换的方法非常多,以前不懂编程的时候,都是用各种网站,而最常用的就是ensembl的biomart了,它支持的ID非常多,高达几百种,好多ID我到现在都不知道是什么意思。
现在学会编程了,我比较喜欢的是R的一些包,是bioconductor系列,一般来说,其中有biomart,org.Hs.eg.db,annotate,等等。关于biomart我就不再讲了,我前面的博客至少有七八篇都提到了它。本次我们讲讲简单的, 我就以把gene entrez ID转换为gene symbol 为例子把。
当然,首先要安装这些包,并且加载。
if("org.Hs.eg.db" %in% rownames(installed.packages()) == FALSE) {source("http://bioconductor.org/biocLite.R");biocLite("org.Hs.eg.db")}
suppressMessages(library(org.Hs.eg.db)) 我比较喜欢这样加载包
library(annotate) #一般都是这样加载包
如果是用org.Hs.eg.db包,首先你只需要读取你的待转换ID文件,构造成一个向量,tmp,然后只需要symbols <- org.Hs.egSYMBOL[as.character(tmp)]就可以得到结果了,返回的symbols是一个对象,需要用toTable这个函数变成数据框。但是这样转换容易出一些问题,比如如果你的输入数据tmp,里面含有一些无法转换的gene entrez ID,就会报错。
而且它支持的ID转换很有限,具体看看它的说明书即可:https://www.bioconductor.org/packages/release/data/annotation/manuals/org.Hs.eg.db/man/org.Hs.eg.db.pdf
org.Hs.eg.db
org.Hs.eg_dbconn
org.Hs.egACCNUM
org.Hs.egALIAS2EG
org.Hs.egCHR
org.Hs.egCHRLENGTHS
org.Hs.egCHRLOC
org.Hs.egENSEMBL
org.Hs.egENSEMBLPROT
org.Hs.egENSEMBLTRANS
org.Hs.egENZYME
org.Hs.egGENENAME
org.Hs.egGO
org.Hs.egMAP
org.Hs.egMAPCOUNTS
org.Hs.egOMIM
org.Hs.egORGANISM
org.Hs.egPATH
org.Hs.egPMID
org.Hs.egREFSEQ
org.Hs.egSYMBOL
org.Hs.egUCSCKG
org.Hs.egUNIGENE
org.Hs.egUNIPROT
如果是用annotate包,首先你还是需要读取你的待转换ID文件,构造成一个向量,tmp,然后用getSYMBOL(as.character(tmp), data='org.Hs.eg')这样直接就返回的还是以向量,只是在原来向量的基础上面加上了names属性。说明书:http://www.bioconductor.org/packages/3.3/bioc/manuals/annotate/man/annotate.pdf
然后你可以把转换好的向量写出去,如下:
1 A1BG
2 A2M
3 A2MP1
9 NAT1
10 NAT2
12 SERPINA3
13 AADAC
14 AAMP
15 AANAT
16 AARS
PS:如果是芯片数据,需要把探针的ID转换成gene,那么一般还需要加载特定芯片的数据包才行:
platformDB <- paste(eset.mas5@annotation, ".db", sep="") #这里需要确定你用的是什么芯片
cat("the annotation is ",platformDB,"\n")
if(platformDB %in% rownames(installed.packages()) == FALSE) {source("http://bioconductor.org/biocLite.R");tmp=try(biocLite(platformDB))}
library(platformDB, character.only=TRUE)
probeset <- featureNames(eset.mas5)
rowMeans <- rowMeans(exprSet)
library(annotate) # lookUp函数是属于annotate这个包的
EGID <- as.numeric(lookUp(probeset, platformDB, "ENTREZID"))
理论上我前面提到的GEOquery包就可以根据一个GSE索引号来获取NCBI提供的所有关于这个GSE索引号的数据了,包括metadata,表达矩阵,soft文件,还有raw data
但是很多时候,那个metadata并不是很整齐,而且一个个下载太麻烦了,所以就需要用R的bioconductor的另一个神奇的包了GEOmetadb
它的示例:http://bioconductor.org/
library(GEOmetadb)
if(!file.exists('GEOmetadb.sqlite')) getSQLiteFile()
file.info('GEOmetadb.sqlite')
con <- dbConnect(SQLite(),'GEOmetadb.sqlite')
dbDisconnect(con)
但是一般不会成功,因为这个包把它的GEOmetadb.sqlite文件放在了国外网盘共享,在国内很难访问,推荐大家想办法下载到本地
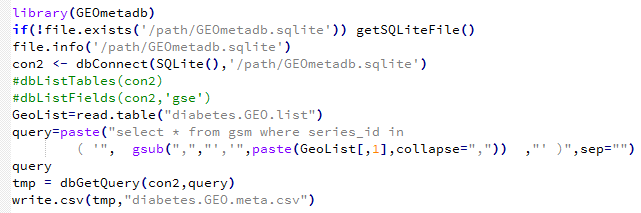 用这个代码就会成功了,需要自己下载GEOmetadb.sqlite文件然后放在指定目录:/path/GEOmetadb.sqlite 需要自己修改
我们的diabetes.GEO.list文件内容如下:
GSE1009
GSE10785
GSE1133
GSE11975
GSE121
GSE12409
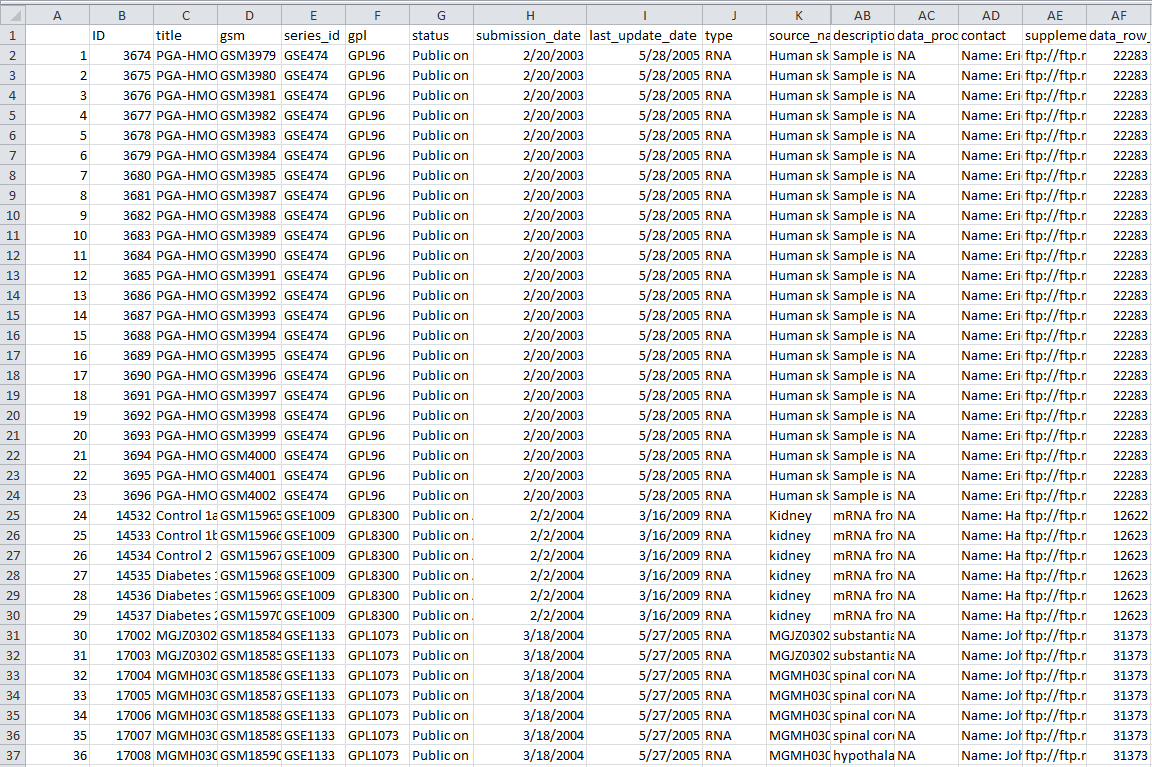
那么会产生的表格文件如下:共有32列数据信息,算是蛮全面的了
用这个代码就会成功了,需要自己下载GEOmetadb.sqlite文件然后放在指定目录:/path/GEOmetadb.sqlite 需要自己修改
我们的diabetes.GEO.list文件内容如下:
GSE1009
GSE10785
GSE1133
GSE11975
GSE121
GSE12409
那么会产生的表格文件如下:共有32列数据信息,算是蛮全面的了
一直以来我都是随便看了点R的编程教程,因为我学了一点点C,所以还算有基础,现在基本上简单看看教程就能懂一门语言了,区别只是熟练度而已。R用得比较多,所以还算擅长,但是很多快捷应用的地方,我总是寄希望于到时候再查资料,所以没能用心的记住,这次花了点时间好好整理了一下R里面关于数据操作的重点,我想,以后再碰到类似的数据处理要求,应该很快能解决了把。
在R中,和排序相关的函数主要有三个:sort(),rank(),order()。
sort(x)是对向量x进行排序,返回值排序后的数值向量。
rank()是求秩的函数,它的返回值是这个向量中对应元素的“排名”。
order()的返回值是对应“排名”的元素所在向量中的位置。
其中sort(x)等同于x[order(x)]
下面以一小段R代码来举例说明:
> x<-c(97,93,85,74,32,100,99,67)
> sort(x)
[1] 32 67 74 85 93 97 99 100
> order(x)
[1] 5 8 4 3 2 1 7 6
> rank(x)
[1] 6 5 4 3 1 8 7 2
> x[order(x)]
[1] 32 67 74 85 93 97 99 100
其中比较有用的order,它可以用来给数据框进行排序
dat[order(dat[,1]),] 以该数据框的第一列进行排序
dat[order(dat[,1],dat[,2]),] 以该数据框的第一列为主要次序,第二列为次要序列进行排序
在R里面除了简单的对两个向量求交集并集补集之外,比较重要的就是match和 %in% 了,需要重点讲讲。
#首先对集合A,B,C赋值
> A<-1:10
> B<-seq(5,15,2)
> C<-1:5
> #求A和B的并集
> union(A,B)
[1] 1 2 3 4 5 6 7 8 9 10 11 13 15
> #求A和B的交集
> intersect(A,B)
[1] 5 7 9
> #求A-B
> setdiff(A,B)
[1] 1 2 3 4 6 8 10
> #求B-A
> setdiff(B,A)
[1] 11 13 15
> #检验集合A,B是否相同
> setequal(A,B)
[1] FALSE
> #检验元素12是否属于集合C
> is.element(12,C)
[1] FALSE
> #检验集合A是否包含C
> all(C%in%A)
[1] TRUE
> all(C%in%B)
从上面可以看到%in%这个操作符只返回逻辑向量TRUE 或者FALSE,而且返回值应该与%in%这个操作符前面的向量程度相等。也就是说它相当于遍历了C里面的一个个元素,判断它们是否在B中出现过,然后返回是或者否即可。
而match(C,B)的结果就很不一样了,它的返回结果同样与前面的向量等长,但是它并非返回逻辑向量,而是遍历了C里面的一个个元素,判断它们是否在B中出现过,如果出现就返回在B中的索引号,如果没有出现,就返回NA。
>B
[1] 5 7 9 11 13 15
>C
[1] 1 2 3 4 5
>match(C,B)
[1] NA NA NA NA 1
>C%in%B
[1] FALSE FALSE FALSE FALSE TRUE
这是一个需要安装的包,起得就是R里面最经典的把长型数据变宽,和把宽数据拉长的作用。
其中melt函数是把很宽的数据拉长,它就是需要指定几列数据是保证不被融合的, 其余每一列数据都必须被融合到一列了,融合后的这一列数据每个元素旁边就用列名来标记该数据来自于哪一列。
# example of melt function
library(reshape)
mdata <- melt(mydata, id=c("id","time"))
融合后的数据如下:
| id | time | variable | value |
| 1 | 1 | x1 | 5 |
| 1 | 2 | x1 | 3 |
| 2 | 1 | x1 | 6 |
| 2 | 2 | x1 | 2 |
| 1 | 1 | x2 | 6 |
| 1 | 2 | x2 | 5 |
| 2 | 1 | x2 | 1 |
| 2 | 2 | x2 | 4 |
可以看到variable列里面有两个不同的元素,说明是把旧数据中的两列给融合了,融合后的一个很长的列就是value
而cast函数的功能就是把刚才融合好的数据给还原。
# cast the melted data
# cast(data, formula, function)
subjmeans <- cast(mdata, id~variable, mean)
timemeans <- cast(mdata, time~variable, mean)
可以看到它们返回的结果是:
subjmeans
| id | x1 | x2 |
| 1 | 4 | 5.5 |
| 2 | 4 | 2.5 |
timemeans
| time | x1 | x2 |
| 1 | 5.5 | 3.5 |
| 2 | 2.5 | 4.5 |
可以看到cast函数比较复杂一点,formula公式右边的变量是需要拆开的variable,这一列变量有多少不重复元素,就新增多少列,左边的变量是行标记了,其余所有数据都会被计算一下再放在合适的位置。
在reshape2这个包里面还进化出来dcast和acast函数,功能大同小异。
这个函数的功能非常强大,类似于SQL语句里面的join系列函数
测试数据如下,它们这两个表的连接是作者名
如果要实现类似sql里面的inner join 功能,则用代码
m1 <- merge(authors, books, by.x = "surname", by.y = "name")
如果要实现left join功能则用代码
m1 <- merge(authors, books, by.x = "surname", by.y = "name",all.x=TRUE)
right join功能代码
m1 <- merge(authors, books, by.x = "surname", by.y = "name",all.y=TRUE)
all join功能代码
m1 <- merge(authors, books, by.x = "surname", by.y = "name",all=TRUE)
关于单变量匹配的总结就是这些,但对于多变量匹配呢,例如下面两个表,需要对k1,k2两个变量都相等的情况下匹配
x <- data.frame(k1 = c(NA,NA,3,4,5), k2 = c(1,NA,NA,4,5), data = 1:5)
y <- data.frame(k1 = c(NA,2,NA,4,5), k2 = c(NA,NA,3,4,5), data = 1:5)
匹配代码如下merge(x, y, by = c("k1","k2")) #inner join
另外一个多行匹配的例子如下:
我们的测试数据如上,这两个表的连接在于作者名。下面这两个语句等价
merge(authors, books, by=1:2)
merge(authors, books, by.x=c("FirstName", "LastName"),
by.y=c("AuthorFirstName", "AuthorLastName"),
all.x=TRUE)
都可以把两张表关联起来。
当然,在我搜索资料的时候,发现了另外一个解决问题的方法:
A[with(A, paste(C1, C2, sep = "\r")) %in% with(B, paste(C1, C2, sep="\r")), ]
参考:http://blog.sina.com.cn/s/blog_6caea8bf0100spe9.html
http://blog.sina.com.cn/s/blog_6caea8bf010159dt.html
http://blog.csdn.net/u014801157/article/details/24372441
http://www.statmethods.net/management/reshape.html
http://r.789695.n4.nabble.com/Matching-multiple-columns-in-a-data-frame-td890832.html
http://bbs.pinggu.org/thread-3234639-1-1.html
http://www.360doc.com/content/14/0821/13/18951041_403561544.shtml
懂点芯片数据分析的都应该知道,芯片设计的时候,对一个基因设计了多个探针,这样设计是为了更好的捕获某些难以发现的基因,或者重点研究某些基因。
但是对我们的差异分析不方便,所以我们只分析哪些有对应了entrez ID的探针,而且对每个entrez ID,我们只需要挑选它表达量最高的那个探针。
所以就演化为一个编程问题:分组求最大值,多公共列合并!
如果是在R语言里面,那么首先这个table的表示形式如下
> esetDataTable[1:10,c(7,8)]
EGID rowMeans
1000_at 5595 1840.04259751826
1001_at 7075 799.075414422572
1002_f_at 1557 50.4884096416177
1003_s_at 643 142.372008051308
1004_at 643 211.65300963049
1005_at 1843 4281.29318032004
1006_at 4319 38.5784289213085
1007_s_at NA 1489.98158531843
1008_f_at 5610 4013.576753977
1009_at 3094 3070.50648167305
我们首先看看一个R语言函数处理方式吧,这个是比较容易想到的算法,但是用到了循环,非常的不经济,计算量很大。
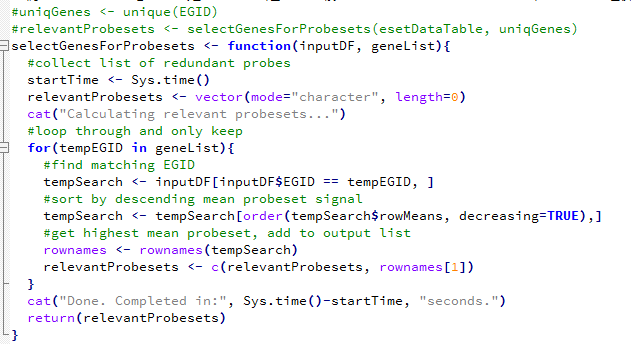
因为里面涉及到了双重循环。我进行了人工计时,这个程序耗费了一分钟十二秒,程序里面的计时器有点问题。
然后我再讲一个精简版的算法
dat=esetDataTable[,c(7,8)]
dat=as.data.frame(apply(dat,2,as.numeric))
dat$prob=rownames(esetDataTable)
首先可以得到需要提取的数据所在行的两个值
match_row=aggregate(dat,by=list(dat[,1]),max)
类似于这句SQL语句:SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID
现在要根据match_row表去原表esetDataTable里面提取我们的探针ID数据。

这样就完美解决了问题,我们的合并后的数据的prob列,就是前面那个函数计算了一分多钟的返回的非冗余探针ID向量,relevantProbesets,但是这次只花了不到一秒钟。
tmp_prob=merge(dat,match_row,by=c("EGID","rowMeans"))
setdiff(as.character(relevantProbesets),as.character(tmp_prob$prob))
length(union(as.character(relevantProbesets),as.character(tmp_prob$prob)))
setdiff(as.character(tmp_prob$prob),as.character(relevantProbesets))
我顺便检查了一下上面那个复杂的R函数跟我这次精简版的结果,发现这次才是对的,上面那个错了。
你们能发现上面那个为什么错了吗?
如果是在mysql里面它的表现形式如下;
mysql> select row_names,EGID,rowMeans from esetDataTable order by EGID limit 10;
+------------+-------+------------------+
| row_names | EGID | rowMeans |
+------------+-------+------------------+
| 38912_at | 10 | 85.8820246154773 |
| 41654_at | 100 | 301.720067595558 |
| 907_at | 100 | 273.100008206028 |
| 2053_at | 1000 | 354.207060199715 |
| 2054_g_at | 1000 | 33.8472900312781 |
| 40781_at | 10000 | 425.007640082848 |
| 35430_at | 10001 | 152.885791914329 |
| 35431_g_at | 10001 | 181.915087187117 |
| 35432_at | 10001 | 184.869017764782 |
| 31366_at | 10002 | 44.9716205901791 |
+------------+-------+------------------+
10 rows in set (0.05 sec)
如果要用SQL来做同样的事情需要下面这个语句,这个就非常简单啦!
select b.*
from test.esetDataTable b
inner join (
SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID
) as c on b.EGID=c.EGID and b.rowMeans=c.val
结果是:8640 rows in set (4.56 sec) 看起来mysql还没有R语言快速,但是这个mysql语句很明显也不是很高效,但是我的mysql水平有限。
稍微解释一下这个mysql语句,其中SELECT max(a.rowMeans) as val, a.EGID FROM test.esetDataTable a group by a.EGID类似于R里面的match_row=aggregate(dat,by=list(dat[,1]),max),就是把每个entrez ID对应着的最大值提取出来成一个新的表
而inner join on b.EGID=c.EGID and b.rowMeans=c.val 就是我们的tmp_prob=merge(dat,match_row,by=c("EGID","rowMeans")) 根据两列来合并两个数据框
毕竟不是计算机科班出身,很多计算机概念我其实并不清楚,很多时候对一个任务的解决只是凭着自己的悟性来模仿接触到的做法,大多数情况下并无可厚非,能完成任务即可,但是碰到多人协作的环境,往往就出了问题。
而condor其实很少见的。
condor_q 可以用来查看任务提交情况
任务提交模式还是挺有用的
[[ ]] double brackets
(())Double parentheses
{{}}double curly brackets
我们必须要记住的是下面
[] 相当于test,作逻辑判断
$( ) 与` ` (反引号) 都是用来做命令替换用
${ } 吧... 它其实就是用来作变量替换用的啦
(())就是用来计算的,相当于expr函数。
http://tldp.org/LDP/abs/html/index.html
我们首先看看一对的括号
首先[]是用来逻辑判断的,必须有空格
if [ -f binom.py ]
then
echo 'binom.py exists'
fi
或者
nub=$((i%4))
#echo $nub
if [ $nub == 0 ];then
echo "we need to sleep 4 hours"
sleep 14000
fi
这个[]操作符等价于test函数
if test $1 -gt 0
then
echo "$1 number is positive"
fi
但是都必须有空格!!!
参考:http://www.freeos.com/guides/lsst/ch03sec02.html
关于shell的test操作符还有很多http://tldp.org/LDP/abs/html/fto.html
( ) 将command group 置于 sub-shell 去执行,也称 nested sub-shell。
{ } 则是在同一个 shell 内完成,也称为non-named command group。
补充一个: {} 还可以做变量扩展 {5..9} 或者 {abcd}e, 自己运行一下就知道效果啦
这两个差异很小,而且一般用不着,就不讲了。
那么这一对的括号加上了$符号后又变成了上面鬼东西呢?
当然,只有:$( ) 与${ }才是合法的。
在 bash shell 中,$( ) 与` ` (反引号) 都是用来做命令替换用(command substitution)的。
在操作上,用$( ) 或` ` 都无所谓,用$( )的优点是:
1, ` ` 很容易与' ' ( 单引号)搞混乱,尤其对初学者来说
2, 在多层次的复合替换中,` ` 须要额外的跳脱( \` )处理,而$( ) 则比较直观
再让我们看${ } 吧... 它其实就是用来作变量替换用的啦。
一般情况下,$var 与${var} 并没有啥不一样。
但是用${ } 会比较精确的界定变量名称的范围,比方说:
[code][/code]
$ A=B
$ echo $AB
还可以用来截取变量,这个就很多花样啦
# 是去掉左边(在鉴盘上# 在$ 之左边)
% 是去掉右边(在鉴盘上% 在$ 之右边)
单一符号是最小匹配﹔两个符号是最大匹配
然后我们看看两对的括号:
nub=$((i%4)) 等价于$nub=`expr $i % 1` ;
((i++)) 等价于$i=`expr $i + 1` ;
所以(())就是用来计算的,而且里面的变量不需要$来标记啦
(在 $(( )) 中的变量名称,可于其前面加$ 符号来替换,也可以不用)
在(())前面加上$只是为了把计算结果给保存而已。
而两个中括号和两个大括号都是不合法的!
如何使用自己写的私人模块
/www/modules/MyMods/Foo.pm
/www/modules/MyMods/Bar.pmAnd then where I use them:
use lib qw(/www/modules);useMyMods::Foo; useMyMods::Bar;
As reported by "perldoc -f use":
It is exactly equivalent to
BEGIN { require Module; import Module LIST; }
except that Module must be a bareword.
Putting that another way, "use" is equivalent to:
require-ing that file name, andimport-ing that package.So, instead of calling use, you can call require and import inside a BEGIN block:
BEGIN{require'../EPMS.pm';
EPMS->import();}And of course, if your module don't actually do any symbol exporting or other initialization when you call import, you can leave that line out:
BEGIN{require'../EPMS.pm';}基因的chr,start,end都是已知的
学术一点讲述这个问题:已知CNV数据在染色体上的position如chr1:2075000-2930999,怎样批量获取其对应的Gene Symbol呢(批量)
数据如下:
head gene_position.hg19 //共21629行
1 chr19 58858171 58874214 A1BG ENSG00000121410
2 chr12 9220303 9268558 A2M ENSG00000175899
3 chr12 9381128 9386803 A2MP1 ENSG00000256069
9 chr8 18027970 18081198 NAT1 ENSG00000171428
10 chr8 18248754 18258723 NAT2 ENSG00000156006
12 chr14 95058394 95090390 ENSG00000273259
13 chr3 151531860 151546276 AADAC ENSG00000114771
14 chr2 219128851 219134893 AAMP ENSG00000127837
15 chr17 74449432 74466199 AANAT ENSG00000129673
16 chr16 70286296 70323412 AARS ENSG00000090861
head pfam.df.hg19.bed //共340960行
chr1 12190 12689 Helicase_C_2 0 + 12190 12689 255,255,0
chr1 69157 69220 7tm_4 0 + 69157 69220 255,255,0
chr1 69184 69817 7TM_GPCR_Srsx 0 + 69184 69817 255,255,0
chr1 69190 69931 7tm_1 0 + 69190 69931 255,255,0
chr1 69490 69910 7tm_4 0 + 69490 69910 255,255,0
现在需要对我们的pfam数据进行注释,根据每一行的chr和pos来看看是属于哪一个基因
总共会有338879 条pfam记录可以注释上基因。
注释之后应该是 head pfam.gene.df.hg19 这个样子
CDK11B chr1 1571423 1573930 Pkinase 0 - 1571423 1573930 255,255,0
CDK11B chr1 1572048 1573921 Pkinase_Tyr 0 - 1572048 1573921 255,255,0
CDK11B chr1 1572120 1572823 Kinase-like 0 - 1572120 1572823 255,255,0
CDK11B chr1 1572120 1572820 Kinase-like 0 - 1572120 1572820 255,255,0
CDK11B chr1 1572120 1572817 Kinase-like 0 - 1572120 1572817 255,255,0
CDK11B chr1 1573173 1573918 Kinase-like 0 - 1573173 1573918 255,255,0
CDK11B chr1 1575747 1577317 Daxx 0 - 1575747 1577317 255,255,0
CDK11B chr1 1576417 1577347 Nop14 0 - 1576417 1577347 255,255,0
CDK11B chr1 1576423 1577332 Mitofilin 0 - 1576423 1577332 255,255,0
CDK11B chr1 1576432 1577317 SAPS 0 - 1576432 1577317 255,255,0
我的第一个程序用的是全基因位点扫描到hash的方法。这样需要扫描13,1390,4974个位点,多于三分之一的基因组,这样是非常浪费内存的,尤其是keys需要多个字节。
我用了256G的服务器都没有运行完。
后来我取巧了把我的gene_position.hg19文件用split命令分成了25个,然后循环25次对pfam.df.hg19.bed 文件进行注释。
这样的确可以解决问了,而且只需要32G的内存的服务器即可,时间也很快,就十多分钟吧。
但这只是取巧的方法,应该要从算法上面优化,首先我仅仅做一个改动,就是不再扫描全基因的位点,对每个基因,我以1K的窗口来取位点进行扫描。这样我判断pfam的坐标时候,也以1K为最小单位进行判断。
这样只需要不到30s就可以出结果,总共注释了303474条pfam记录,还不是最终的338879,因为我这次只注释了基因的1000整数倍基因区间,这样如果pfam记录落在一个基因的起始终止点不到1K位置时就不会被注释。这时候需要对代码进行继续优化。
脚步懒得上传了,在我的有道云笔记里面。
http://note.youdao.com/share/?id=58e66d138e9434284ffa61c53b65abdc&type=note
前面我提到有一个大的运算任务需要很久才完成,所以用到了进度条来监控过程,但并不是改善了计算速度,所以需要用到并行计算,我又在网上找了找。
同样也是一个包,跟matlab的实现过程很像
library(parallel)
cl.cores <- detectCores() #检查当前电脑可用核数。
cl <- makeCluster(cl.cores) #使用刚才检测的核并行运算
#这里用clusterEvalQ或者par开头的apply函数族就可以进行并行计算啦
stopCluster(cl)
R-Doc里这样描述makeCluster函数:Creates a set of copies of R running in parallel and communicating over sockets. 即同时创建数个R进行并行运算。在该函数执行后就已经开始并行运算了,电脑可能会变卡一点。尤其在执行par开头的函数时。
在并行运算环境下,常用的一些计算方法如下:
1、clusterEvalQ(cl,expr)函数利用创建的cl执行expr。这里利用刚才创建的cl核并行运算expr。expr是执行命令的语句,不过如果命令太长的话,一般写到文件里比较好。比如把想执行的命令放在Rcode.r里:clusterEvalQ(cl,source(file="Rcode.r"))
2、par开头的apply函数族。这族函数和apply的用法基本一样,不过要多加一个参数cl。一般如果cl创建如上面cl <- makeCluster(cl.cores)的话,这个参数可以直接用作parApply(cl=cl,…)。当然Apply也可以是Sapply,Lapply等等。注意par后面的第一个字母是要大写的,而一般的apply函数族第一个字母不大写。
另外要注意,即使构建了并行运算的核,不使用parApply()函数,而使用apply()函数的话,则仍然没有实现并行运算。换句话说,makeCluster只是创建了待用的核,而不是并行运算的环境。
参考:http://www.r-bloggers.com/lang/chinese/1131
然后我模仿着用并行计算实现自己的需求
#it did work very fast
library(parallel)
cl.cores <- detectCores()
cl <- makeCluster(cl.cores)
clusterExport(cl, "all_dat_t") #这里是重点,因为并行计算里面用到了自定义函数
clusterExport(cl, "all_prob_id") #但是这个函数需要用到这两个数据,所以需要把这两个数据加载到并行计算环境里面
prob_202723_s_at=parSapply( #我这里用的parSapply来实现并行计算
cl=cl, #其中cl是我前面探测到的core数量,
deviation_prob, #deviation_prob是我待并行处理的向量
test_pro #这里其实应该是一个自定义函数,我这里就不写出来了,对上面的deviation_prob向量的每个探针都进行判断
)
我也是临时在网上搜索到的教程,然后简单看了一下就实现了,其实就是就用到了一个名称为tcltk的包,直接查看函数tkProgressBar就可以知道怎么用啦!
下面是网上的一个小的示例代码(么有实际意义,仅为举例而已):
library(tcltk2)
u <- 1:2000
plot.new()
pb <- tkProgressBar("进度","已完成 %", 0, 100)
for(i in u) {
x<-rnorm(u)
points(x,x^2,col=i)
info <- sprintf("已完成 %d%%", round(i*100/length(u)))
setTkProgressBar(pb, i*100/length(u), sprintf("进度 (%s)", info), info)
}
close(pb)#关闭进度条
但是下面的代码是我模仿上面这个教程自己实现的。
[R]
# 以下是实现进度条
library(tcltk2)
plot.new()
pb <- tkProgressBar("进度","已完成 %", 0, 100)
prob_202723_s_at_value=rep(0,length(deviation_prob))
start_time=Sys.time() #这里可以计时,因为要实现进度条的一般都是需要很长运算时间
for (i in 1:length(deviation_prob)) {
tmp=test_pro(deviation_prob[i]) #test_pro是我自定义的一个函数,判断该探针是否符合要求。
if (length(tmp)!=0){prob_202723_s_at_value[i]=tmp}
info <- sprintf("已完成 %d%%", round(i*100/length(deviation_prob))) #进度条就是根据循环里面的i来看看循环到哪一步了
setTkProgressBar(pb, i*100/length(deviation_prob), sprintf("进度 (%s)", info), info)
}
close(pb)#关闭进度条
end_time=Sys.time()
cat(end_time-start_time)
[/R]
结论:从数据框里面取某列数据,三种方法的时间消耗区别很大,直接用索引值,是最快的,而用$符号其次,用列名最慢。
我在R里面建立了一个表达量矩阵,列名是一个个样品,行是一个个探针,矩阵值是该探针在该样品测定的表达量。
那么,如果我要看看名为"202723_s_at"的探针的表达向量与其它所有探针的表达向量的相关系数,我可以用以下三种方法:
> system.time(apply(all_dat_t,2,function(x) cor(all_dat_t$"202723_s_at",x)))
user system elapsed
22.93 0.03 23.03
> system.time(apply(all_dat_t,2,function(x) cor(all_dat_t[,"202723_s_at"],x)))
Timing stopped at: 92.02 5.32 97.66
太耗时间了,省去
> system.time(apply(all_dat_t,2,function(x) cor(all_dat_t[,grep(prob,names(all_dat_t))],x)))
Timing stopped at: 13.55 0.04 13.66
> prob_num=grep(prob,names(all_dat_t))
> system.time(apply(all_dat_t,2,function(x) cor(all_dat_t[,prob_num],x)))
user system elapsed
8.14 0.01 8.17
可以看出,如果我首先根据探针名,grep出它在该表达量矩阵的列数,然后用列数来提取它的表达量是最快的,而且时间改善非常明显!
我们再探究一下cor函数的效率问题
探究的矩阵有54675个变量,每个变量均有189个观测值,如果取这个大矩阵的部分变量来求相关系数,结果如下!
> system.time(cor(all_dat_t[,1:10]))
user system elapsed
0.001 0.000 0.001
> system.time(cor(all_dat_t[,1:100]))
user system elapsed
0.003 0.000 0.003
> system.time(cor(all_dat_t[,1:1000]))
user system elapsed
0.107 0.002 0.108
> system.time(cor(all_dat_t[,1:10000]))
user system elapsed
11.102 0.849 11.983
> system.time(cor(all_dat_t)) 约六分钟也是可以搞定的
但是如果cor(all_dat_t),六分钟后得到的相关系数矩阵约32G,非常恐怖!
但是它很明显没有把这个32G相关系数矩阵存储到内存,因为我的机器本来就16G内存。我至今不能明白R具体实现机理
bioconductor系列的包都是一样的安装方式:
source("http://bioconductor.org/biocLite.R")
biocLite("ConsensusClusterPlus")
这个包是我见过最简单的包, 加载只有做好输入数据,只需要一句话即可运行,然后默认输出所有结果
> d[1:5,1:5]
d = sweep(d,1, apply(d,1,median,na.rm=T))
bioconductor系列的包都是一样的安装方式:
source("http://bioconductor.org/biocLite.R")
biocLite("GEOquery")
以前GEO数据库主要是microarray的芯片数据,后来有了RNA-seq,如果同时做多个样品的RNA-seq,表达量矩阵后来也可以上传到GEO数据库里面,只有看到文献里面有提到GEO数据库,都可以通过这个R包俩进行批量下载,其实就是网页版的一个API调用而已:
这个函数有很多参数,除非你需要下载的文件,那么就设置destdir到你喜欢的目录,如果只需要表达量数据就不用了。
#Download GDS file, put it in the current directory, and load it:
gds858 <- getGEO('GDS858', destdir=".")
如果使用了GSEMatrix=TRUE这个参数,那么除了下载soft文件,还有表达量矩阵文件,可以直接用read.table读取那个文件。
#Or, open an existing GDS file (even if its compressed):
gds858 <- getGEO(filename='GDS858.soft.gz')
下面这个下载的是GSE对象,GDS对象还有大一点